正在加载图片...
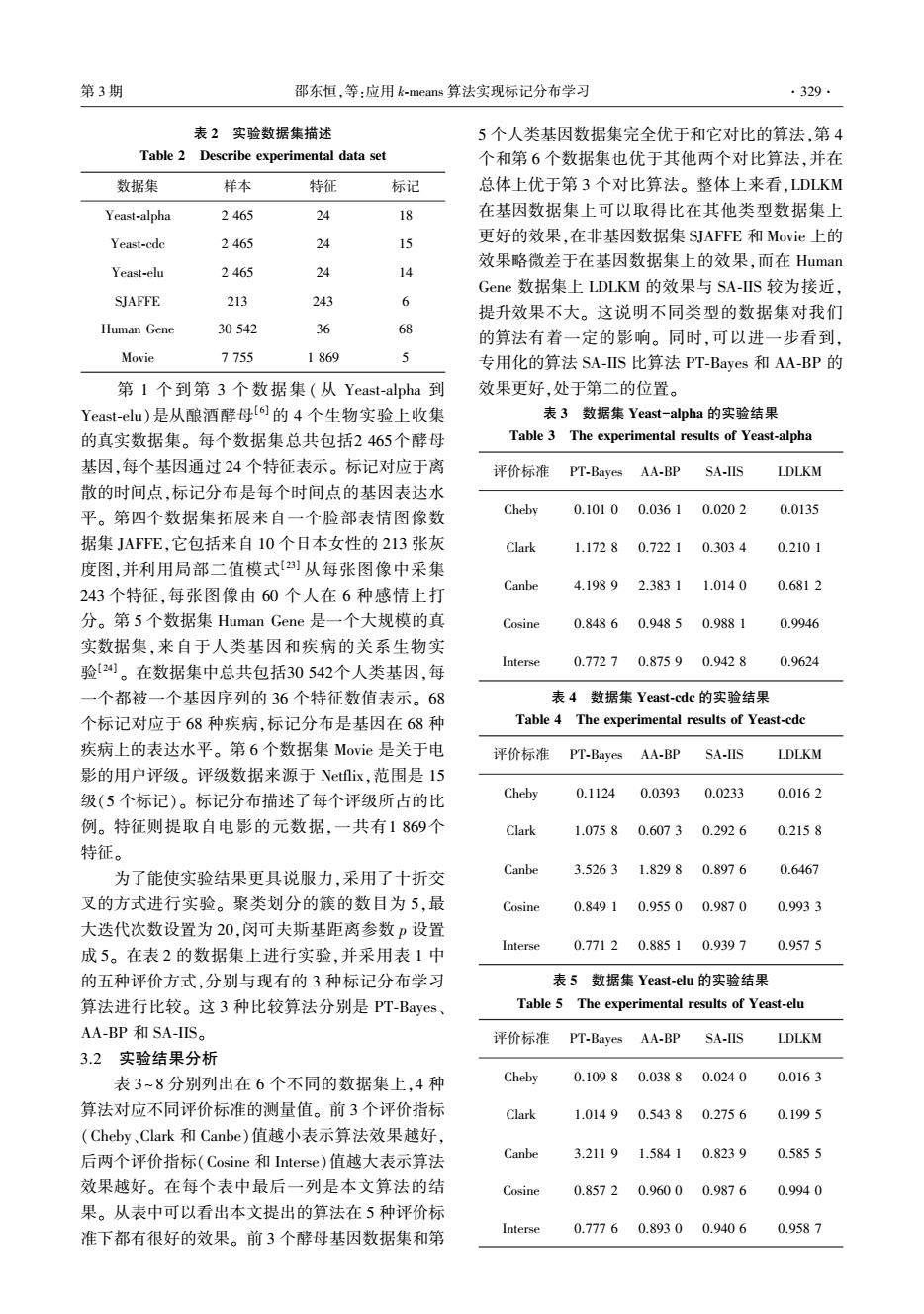
第3期 邵东恒,等:应用k-means算法实现标记分布学习 ·329· 表2实验数据集描述 5个人类基因数据集完全优于和它对比的算法,第4 Table 2 Describe experimental data set 个和第6个数据集也优于其他两个对比算法,并在 数据集 样本 特征 标记 总体上优于第3个对比算法。整体上来看,LDLKM Yeast-alpha 2465 24 18 在基因数据集上可以取得比在其他类型数据集上 Yeast-cde 2465 24 15 更好的效果,在非基因数据集SJAFFE和Movie上的 效果略微差于在基因数据集上的效果,而在Human Yeast-elu 2465 24 14 Gene数据集上LDLKM的效果与SA-IIS较为接近, SJAFFE 213 243 6 提升效果不大。这说明不同类型的数据集对我们 Human Gene 30542 36 68 的算法有着一定的影响。同时,可以进一步看到, Movie 7755 1869 J 专用化的算法SA-IIS比算法PT-Bayes和AA-BP的 第1个到第3个数据集(从Yeast-alpha到 效果更好,处于第二的位置。 Yeast-elu)是从酿酒酵母6的4个生物实验上收集 表3数据集Yeast--alpha的实验结果 的真实数据集。每个数据集总共包括2465个酵母 Table 3 The experimental results of Yeast-alpha 基因,每个基因通过24个特征表示。标记对应于离 评价标准PT-Bayes AA-BP SA-IIS LDLKM 散的时间点,标记分布是每个时间点的基因表达水 平。第四个数据集拓展来自一个脸部表情图像数 Cheby 0.1010 0.0361 0.0202 0.0135 据集JAF℉E,它包括来自10个日本女性的213张灰 Clark 1.17280.7221 0.3034 0.2101 度图,并利用局部二值模式]从每张图像中采集 Canbe 4.1989 2.3831 1.0140 243个特征,每张图像由60个人在6种感情上打 0.6812 分。第5个数据集Human Gene是一个大规模的真 Cosine 0.8486 0.9485 0.9881 0.9946 实数据集,来自于人类基因和疾病的关系生物实 验[24】。在数据集中总共包括30542个人类基因,每 Interse 0.7727 0.8759 0.9428 0.9624 一个都被一个基因序列的36个特征数值表示。68 表4数据集Yeast-.cdc的实验结果 个标记对应于68种疾病,标记分布是基因在68种 Table 4 The experimental results of Yeast-cdc 疾病上的表达水平。第6个数据集Movie是关于电 评价标准 PT-Bayes AA-BP SA-IIS LDLKM 影的用户评级。评级数据来源于Netflix,范围是15 级(5个标记)。标记分布描述了每个评级所占的比 Cheby 0.1124 0.0393 0.0233 0.0162 例。特征则提取自电影的元数据,一共有1869个 Clark 1.0758 0.6073 0.2926 0.2158 特征。 为了能使实验结果更具说服力,采用了十折交 Canbe 3.5263 1.8298 0.8976 0.6467 叉的方式进行实验。聚类划分的簇的数目为5,最 Cosine 0.8491 0.95500.9870 0.9933 大迭代次数设置为20,闵可夫斯基距离参数p设置 成5。在表2的数据集上进行实验,并采用表1中 Interse 0.77120.8851 0.9397 0.9575 的五种评价方式,分别与现有的3种标记分布学习 表5数据集Yeast-.elu的实验结果 算法进行比较。这3种比较算法分别是PT-Bayes、 Table 5 The experimental results of Yeast-elu AA-BP和SA-IIS。 评价标准 PT-Bayes AA-BP SA-IIS LDLKM 3.2实验结果分析 表3~8分别列出在6个不同的数据集上,4种 Cheby 0.1098 0.0388 0.0240 0.0163 算法对应不同评价标准的测量值。前3个评价指标 Clark 1.01490.5438 0.2756 0.1995 (Cheby、Clark和Canbe)值越小表示算法效果越好, 后两个评价指标(Cosine和Interse)值越大表示算法 Canbe 3.2119 1.5841 0.8239 0.5855 效果越好。在每个表中最后一列是本文算法的结 Cosine 0.85720.9600 0.9876 0.9940 果。从表中可以看出本文提出的算法在5种评价标 准下都有很好的效果。前3个酵母基因数据集和第 Interse 0.77760.89300.9406 0.9587表 2 实验数据集描述 Table 2 Describe experimental data set 数据集 样本 特征 标记 Yeast⁃alpha 2 465 24 18 Yeast⁃cdc 2 465 24 15 Yeast⁃elu 2 465 24 14 SJAFFE 213 243 6 Human Gene 30 542 36 68 Movie 7 755 1 869 5 第 1 个到第 3 个数据集 ( 从 Yeast⁃alpha 到 Yeast⁃elu)是从酿酒酵母[6] 的 4 个生物实验上收集 的真实数据集。 每个数据集总共包括2 465个酵母 基因,每个基因通过 24 个特征表示。 标记对应于离 散的时间点,标记分布是每个时间点的基因表达水 平。 第四个数据集拓展来自一个脸部表情图像数 据集 JAFFE,它包括来自 10 个日本女性的 213 张灰 度图,并利用局部二值模式[23] 从每张图像中采集 243 个特征,每张图像由 60 个人在 6 种感情上打 分。 第 5 个数据集 Human Gene 是一个大规模的真 实数据集,来自于人类基因和疾病的关系生物实 验[24] 。 在数据集中总共包括30 542个人类基因,每 一个都被一个基因序列的 36 个特征数值表示。 68 个标记对应于 68 种疾病,标记分布是基因在 68 种 疾病上的表达水平。 第 6 个数据集 Movie 是关于电 影的用户评级。 评级数据来源于 Netflix,范围是 15 级(5 个标记)。 标记分布描述了每个评级所占的比 例。 特征则提取自电影的元数据,一共有1 869个 特征。 为了能使实验结果更具说服力,采用了十折交 叉的方式进行实验。 聚类划分的簇的数目为 5,最 大迭代次数设置为 20,闵可夫斯基距离参数 p 设置 成 5。 在表 2 的数据集上进行实验,并采用表 1 中 的五种评价方式,分别与现有的 3 种标记分布学习 算法进行比较。 这 3 种比较算法分别是 PT⁃Bayes、 AA⁃BP 和 SA⁃IIS。 3.2 实验结果分析 表 3~8 分别列出在 6 个不同的数据集上,4 种 算法对应不同评价标准的测量值。 前 3 个评价指标 (Cheby、Clark 和 Canbe)值越小表示算法效果越好, 后两个评价指标(Cosine 和 Interse)值越大表示算法 效果越好。 在每个表中最后一列是本文算法的结 果。 从表中可以看出本文提出的算法在 5 种评价标 准下都有很好的效果。 前 3 个酵母基因数据集和第 5 个人类基因数据集完全优于和它对比的算法,第 4 个和第 6 个数据集也优于其他两个对比算法,并在 总体上优于第 3 个对比算法。 整体上来看,LDLKM 在基因数据集上可以取得比在其他类型数据集上 更好的效果,在非基因数据集 SJAFFE 和 Movie 上的 效果略微差于在基因数据集上的效果,而在 Human Gene 数据集上 LDLKM 的效果与 SA⁃IIS 较为接近, 提升效果不大。 这说明不同类型的数据集对我们 的算法有着一定的影响。 同时,可以进一步看到, 专用化的算法 SA⁃IIS 比算法 PT⁃Bayes 和 AA⁃BP 的 效果更好,处于第二的位置。 表 3 数据集 Yeast-alpha 的实验结果 Table 3 The experimental results of Yeast⁃alpha 评价标准 PT⁃Bayes AA⁃BP SA⁃IIS LDLKM Cheby 0.101 0 0.036 1 0.020 2 0.0135 Clark 1.172 8 0.722 1 0.303 4 0.210 1 Canbe 4.198 9 2.383 1 1.014 0 0.681 2 Cosine 0.848 6 0.948 5 0.988 1 0.9946 Interse 0.772 7 0.875 9 0.942 8 0.9624 表 4 数据集 Yeast⁃cdc 的实验结果 Table 4 The experimental results of Yeast⁃cdc 评价标准 PT⁃Bayes AA⁃BP SA⁃IIS LDLKM Cheby 0.1124 0.0393 0.0233 0.016 2 Clark 1.075 8 0.607 3 0.292 6 0.215 8 Canbe 3.526 3 1.829 8 0.897 6 0.6467 Cosine 0.849 1 0.955 0 0.987 0 0.993 3 Interse 0.771 2 0.885 1 0.939 7 0.957 5 表 5 数据集 Yeast⁃elu 的实验结果 Table 5 The experimental results of Yeast⁃elu 评价标准 PT⁃Bayes AA⁃BP SA⁃IIS LDLKM Cheby 0.109 8 0.038 8 0.024 0 0.016 3 Clark 1.014 9 0.543 8 0.275 6 0.199 5 Canbe 3.211 9 1.584 1 0.823 9 0.585 5 Cosine 0.857 2 0.960 0 0.987 6 0.994 0 Interse 0.777 6 0.893 0 0.940 6 0.958 7 第 3 期 邵东恒,等:应用 k⁃means 算法实现标记分布学习 ·329·