正在加载图片...
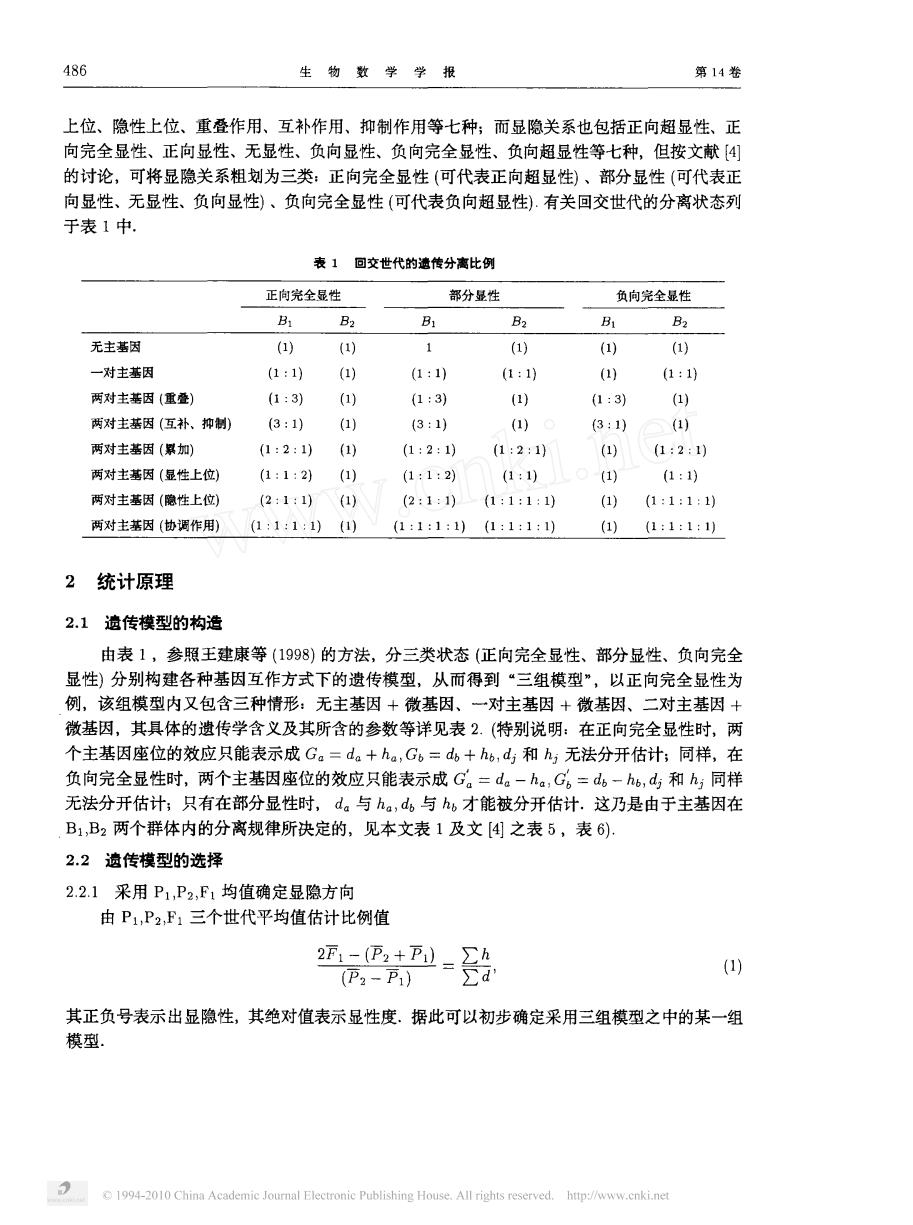
486 生物数学学报 第14卷 上位、隐性上位、重叠作用、互补作用、抑制作用等七种;而显隐关系也包括正向超显性、正 向完全显性、正向显性、无显性、负向显性、负向完全显性、负向超显性等七种,但按文献【4 的讨论,可将显隐关系粗划为三类:正向完全显性(可代表正向超显性)、部分显性(可代表正 向显性、无显性、负向显性)、负向完全显性(可代表负向超显性).有关回交世代的分离状态列 于表1中. 表1回交世代的传分离比例 正向完全显性 部分显性 负向完全显性 B1 B2 B1 B2 B B2 无主因 1 一对主基因 (1:1} (1) 1:10 (1:1) (1) 1:1) 两对主因(重叠 (1:3) 1:3) ) 1:3到 两对主基因(互补、抑制) 3:1) 1) (3:1) (1) 3:1)(1) 两对主基因(累加) (1:2:1) 1:2:1 1:2:1) 两对主基因(显性上位) (1:1:2)(1) (1:1:2 (1:1) (1) (1:1) 两对主基因(隐性上位) (2:1:1)() (2:1:1)(1:1:1:1) 0)(1:1:1:) 两对主基烟(协调作用)(1:1:1:)()(:1:1:)1:1:1:少 (1)(1:1:1:1) 2统计原理 2.1遗传模型的构造 由表1,参照王建康等(1998)的方法,分三类状态(正向完全显性、部分显性、负向完全 显性)分别构建各种基因互作方式下的遗传模型,从而得到“三组模型”,以正向完全显性为 例,该组模型内又包含三种情形:无主基因+微基因、一对主基因+微基因、二对主基因十 微基因,其具体的遗传学含义及其所含的参数等详见表2.(特别说明:在正向完全显性时,两 个主基因座位的效应只能表示成G。=d。+ha,G。=d山+n,d和,无法分开估计,同样,在 负向完全显性时,两个主基因座位的效应只能表示成G。=da-ha,G。=d山-hb,d和h,同样 无法分开估计,只有在部分显性时,d。与h,d。与才能被分开估计.这乃是由于主基因在 B1,B2两个群体内的分离规律所决定的,见本文表1及文[④之表5,表6). 22遗传模型的选择 2.21采用P1,P2,F1均值确定显隐方向 由P1,P2,F三个世代平均值估计比例值 - () P2-P) 其正负号表示出显隐性,其绝对值表示显性度.据此可以初步确定采用三组模型之中的某一组 模型. 1994-010 China Academic Joumal Electronie Publishing House All rights seved htp://www.nkinet生 物 数 学 学 报 第 卷 上位 、 隐性上位 、 重叠作用 、 互补作用 、 抑制作用等七种 而显隐关系也包括正 向超显性 、 正 向完全显性 、 正 向显性 、 无显性 、 负向显性 、 负向完全显性 、 负向超显性等七种 , 但按文献 的讨论 , 可将显隐关系粗划为三类 正 向完全显性 可代表正 向超显性 、 部分显性 可代表正 向显性 、 无显性 、 负向显性 、 负向完全显性 可代表负向超显性 有关 回交世代的分离状态列 于表 中 表 回 交世代的遗传分离比例 正 向完全显性 部分显性 负向完全显性 无主基因 一对主基因 两对主基因 重叠 两对主基因 互补 、 抑制 两对主基因 累加 两对主基因 显性上位 两对主基因 隐性上位 两对主基因 协调作用 统计原理 遗传模型的构造 由表 , 参照王建康等 的方法 , 分三类状态 正 向完全显性 、 部分显性 、 负向完全 显性 分别构建各种基因互作方式下的遗传模型 , 从而得到 “ 三组模型 ” , 以正 向完全显性为 例 , 该组模型 内又包含三种情形 无主基因 微基因 、 一对主基因 微基因 、 二对主基因 微基因 , 其具体的遗传学含义及其所含的参数等详见表 特别说明 在正 向完全显性时 , 两 个主基因座位的效应只能表示成 。 。 。 , 。 。 。, 内 和 无法分开估计 同样 , 在 负向完全显性时 , 两个主基因座位的效应只能表示成 几 。 一 。 , 试 。 一 。, 呜 和 , 同样 无法分开估计 只有在部分显性时 , 。 与 。 , 。 与 。 才能被分开估计 这乃是 由于主基因在 , 两个群体 内的分离规律所决定的 , 见本文表 及文 之表 , 表 遗传模型的选择 采用 , , 均值确定显 隐方 向 由 , , , 三个世代平均值估计 比例值 一 尸。 十 尸 犷 尸 一 尸 艺 ’ 其正负号表示 出显隐性 , 其绝对值表示 显性度 据此可以初步确定采用三组模型之 中的某一组 模型