
主要基因检测方法原理及优缺点 (-PCR PCR(聚合酶链式反应)是一种用于放大扩增特定DNA片段的分子生物学技术,可以看作 是生物体外的特殊DNA复制,其最大特点是能将微量的DNA大幅增加。在存在DNA模 板、引物、dNTPs、.适当缓冲液(Mg2+)的反应混合物中,在热稳定DNA聚合酶的催化下, 对一对寡核苷酸引物所界定的核酸片段进行扩增,这种扩增是以模板DNA与引物之间的变 性、退火、延伸三步反应为一个周期,循环进行,使目标DNA片段得以扩增。 普通PCR原理图 口0Ls物UU可 HHPHBHH五 果2%,GC 模板DNA dTTP 加热经90-95℃ 加热经90-95℃ UU口0U0可(D#)加A经90-95C 0monnouon 1加热经55-60℃ H出UUUg 重复 D0ooonnHHH 加热至70-75℃ H出UU元 3 y HHHH 子代DNA HHH出五之会世参 最初的普通PC技术只能通过对靶基因进行扩增,然后采用琼脂糖凝胶电泳对产物进行分 析,实现简单的定性分析,这是第一代PCR。鉴于(1)第一代PCR采用的核酸燃料对实 验人员及环境造成较大伤害,(2)PC完成之后开盖检测易污染造成假阳性结果,(3)检测 耗时长且操作麻烦易出错及(4)只能做定性检测,二代PC得以发展并成为目前国内PCR 技术主流。 第二代PCR就是荧光定量PCR技术(Real-Time PCR),也叫做qPCR,是指通过应用荧 光染料或荧光标记的特异性探针如Tagman Probe),在PCR反应体系中加入荧光基团对 聚合酶链反应产物进行标记跟踪,实时监控反应过程,结合软件对荧光信号进行分析,实 现基因检测的定性和定量分析。qPC常用于传染病病原体检测,疾病耐药基因研究,药物 疗效考核,遗传疾病诊断。随着反应循环次数的增加,被扩增目的基因片段呈指数增长,通 过实时检测对应的随扩增而变化荧光信号强度,求得C值,利用已知模板浓度的标准品作 对照,即可得出待测标本目的基因数。由于PCR定量过程通过Ct值间接反映,因此称为 第二代PCR。 实时荧光定量PCR原理图
主要基因检测方法原理及优缺点 (一)PCR PCR(聚合酶链式反应)是一种用于放大扩增特定 DNA 片段的分子生物学技术,可以看作 是生物体外的特殊 DNA 复制,其最大特点是能将微量的 DNA 大幅增加。在存在 DNA 模 板、引物、dNTPs、适当缓冲液 (Mg2+) 的反应混合物中,在热稳定 DNA 聚合酶的催化下, 对一对寡核苷酸引物所界定的核酸片段进行扩增,这种扩增是以模板 DNA 与引物之间的变 性、退火、延伸三步反应为一个周期,循环进行,使目标 DNA 片段得以扩增。 普通 PCR 原理图 最初的普通 PCR 技术只能通过对靶基因进行扩增,然后采用琼脂糖凝胶电泳对产物进行分 析,实现简单的定性分析,这是第一代 PCR。鉴于(1)第一代 PCR 采用的核酸燃料对实 验人员及环境造成较大伤害,(2)PCR 完成之后开盖检测易污染造成假阳性结果,(3)检测 耗时长且操作麻烦易出错及(4)只能做定性检测,二代 PCR 得以发展 并成为目前国内 PCR 技术主流。 第二代 PCR 就是荧光定量 PCR 技术(Real-Time PCR),也叫做 qPCR,是指通过应用荧 光染料或荧光标记的特异性探针(如 Taqman Probe),在 PCR 反应体系中加入荧光基团对 聚合酶链反应产物进行标记跟踪,实时监控反应过程,结合软件对荧光信号 进行分析,实 现基因检测的定性和定量分析。qPCR 常用于传染病病原体检测,疾病耐药基因研究,药物 疗效考核,遗传疾病诊断。随着反应循环次数的增加,被扩增目的基因片段呈指数增长,通 过实时检测对应的随扩增而变化荧光信号强度,求得 Ct 值,利用已知模板浓度的标准品作 对照,即可得出待测标本目的基因数。由于 rPCR 定量过程通过 Ct 值间接反映,因此称为 第二代 PCR。 实时荧光定量 PCR 原理图

A)SYBR Green I assay B)TaqMan assay 00 Denaturation 00 Annealing @◆ rter signa Extension SYBR en I dye Tag Tag DNA polymerase ●TaqMan probe 应用荧光染料:SYBR green:在PCR反应体系中,加入过量SYBR荧光染料,SYBR荧光 染料特异性地掺入DNA双链后 发射荧光信号 而不掺入链中的SYBR染料分子不会发 射任何荧光信号,从而保证荧光信号的增加与PC产物的增加完全同步。 应用特异标记探针:Tagman Probe:PCR扩增时在加入一对引物的同时加入一个特异性的 荧光探针,该探针为一寡核苷酸,两端分别标记一个报告荧光基团和一个淬灭荧光基团。 探针完整时,报告基团发射的荧光信号被淬灭基团吸收:PCR扩增时,Tg酶的5-3 外切酶活性将探针酶切降解 ,使报告荧光基团和淬灭荧光基团分离,从而荧光监测系统可接 收到荧光信号,即每扩增一条DNA链,就有一个荧光分子形成,实现了荧光信号的累积与 PCR产物形成完全同步。 荧光染料VS荧光採针 方法 SYBR green Taqman Probe 特点 灵敏度高:使用SYBR可使荧光 具有高适应性和可靠性 实验结米稳定重复性好 放果增强到1000倍以上 特异性更高 适用于扩增序列专一的体系检测、 不雪要设计探针 通用性 方法简便,省时,价格低 靶基因会量过低的定量PCR检测、存在 与靶基因同源序列,易出现非特异性打 在国内外科研中普通使用 增,对特异性要求较高的定量检测 广泛用于传染病珍断和病原定量 含适用子高通量大规模或专一 动物病原体基因的检测 适用场 要求不高的定量PCR检测 生物制品的要定 随着PCR系统的进一步发展已经对检测,一种具备较高灵敏度和特异性的数字PCR技术
应用荧光染料:SYBR green:在 PCR 反应体系中,加入过量 SYBR 荧光染料, SYBR 荧光 染料特异性地掺入 DNA 双链后,发射荧光信号,而不掺入链中的 SYBR 染料分子不会发 射任何荧光信号,从而保证荧光信号的增加与 PCR 产物的增加完全同步。 应用特异标记探针:Taqman Probe:PCR 扩增时在加入一对引物的同时加入一个特异性的 荧光探针,该探针为一寡核苷酸,两端分别标记一个报告荧光基团和一个淬灭 荧光基团。 探针完整时,报告基团发射的荧光信号被淬灭基团吸收;PCR 扩增时,Taq 酶的 5’-3’ 外切酶活性将探针酶切降解,使报告荧光基团和淬灭荧光基团分离,从而荧光监测系统可接 收到荧光信号,即每扩增一条 DNA 链,就有一个荧光分子形成,实现了荧光信号的累积与 PCR 产物形成完全同步。 荧光染料 VS 荧光探针 随着 PCR 系统的进一步发展已经对检测,一种具备较高灵敏度和特异性的数字 PCR 技术

开始被广泛应用于肿瘤临床检测和科学研究中。 数字PCR(Digitai PCR,dPCR)是一种新兴的核酸检测技术.通过将每个核酸分子分配到 避免选择性扩增对 扩增结果的干扰, 皮独之的是、pA甲基化因功电手字P阳R真 可实现核酸模板绝对定量 稀有突 量分析,被称为第三代PCR。 数字PCR原理图 Ao医,世象 通过稀释样品DNA到对应的检测孔中,经过PCR扩增之后,向每个孔里加入特异性荧光 探针与产物杂交,然后直接计数样本中突变型和野生型等位子的数量。dPCR常用于大量正 常细胞群中检测少数含突变的细胞,主要应用于突变分析、等位基因缺失、混杂DNA的 症检测等 三代PCR的技术优劣 方 技术优点 技术缺点 ①最初的PCR技术所采用的核酸染料 会对实验人员和环境造成伤害 -种用于放大护增特定的DNA 第一代 ②PR完成之后需要开盖检测 PCR 片段的分子生物学技术,可看价 容易发生污染造成假阳性结米 是生物体外的特珠DNA复制 ③检测耗时长,操作麻烦,客易出错 ④只能做定性检测 ①不同厂家的试剂和设备所产生的Cg值 ①污染风险低 有一定差异,不具可比性 第二代 ②操作流程更简单、经济高效 ②存在背景值影响,结果易产生偏差 q-PCR ③样品加样量少 ③低拷贝DNA往往难以检测 ④当反应体系中有PCR抑制物存在时 常规的PCR体系经常会受到影响 ①灵敏度更高:d-PCR将传筑 ①模板虞量要求较高:dPCR的模板量超过 PCR反应分割成了数万个体系 强体系量会导致无法定量,过少会导致信号 这些体系独立扩增与检测 过低,降低定量准确度 第三代 ②定量更准确 ②非特异性产物的判断:无论PCR产物是 d-PCR ③抗干扰能力更强:dPCR检测 是特异性产物,只要荧光信号足够强,也 的是修点荧光,即使微体系销假 影响,也不影响最终结果的判读 是会刺黄为阳生结菜,司先是的别的赞 异性要求极高
开始被广泛应用于肿瘤临床检测和科学研究中。 数字 PCR(Digitai PCR, dPCR)是一种新兴的核酸检测技术,通过将每个核酸分子分配到 一个独立的空间内,避免选择性扩增对扩增结果的干扰,可实现核酸模板绝对定量、稀有突 变检测、拷贝数变异、DNA 甲基化、基因重排等检测功能。由于数字 PCR 可以实现直接定 量分析,被称为第三代 PCR。 数字 PCR 原理图 通过稀释样品 DNA 到对应的检测孔中,经过 PCR 扩增之后,向每个孔里加入特异性荧光 探针与产物杂交,然后直接计数样本中突变型和野生型等位子的数量。dPCR 常用于大量正 常细胞群中检测少数含突变的细胞,主要应用于突变分析、等位基因缺失、 混杂 DNA 的 癌症检测等等。 三代 PCR 的技术优劣

(=NGS NGS(高酒量测序技术)是指酒过模板DNA分子的化学修饰。将其锚定在纳米孔或微载体 芯片 利用碱基互补配对原理,在DNA聚合酶链反应或DNA连接酶反应过程中,通过 采集荧光标记信号或化学反应信号,实现碱基序列的解读, -次性可完成几十万至上百万 序列的测定。 NGS是基于PCR和基因芯片发展而来的DNA测序技术。一代测序为合成终止测序,而 湿华测艇性的认7可道终止末球一从而安现边合皮边电 二代测序在DNA复制过 所添加的碱基所携带的特殊标记(一般为荧光分子标记)来确定DNA的序 列。 目前高通量测序的主要平合代表有罗氏公司(Rohe)的454测序仪(Roch GS FLX sequencer)),lumina公司的Solexa基因组分析仪(llumina Genome Analyzer)和ABl的 SOLiD测序仪(ABI SOLiD er). 主流NGS测序平台技术原理、特点及适用范围 公司 测序原理 然骑酸测序 可逆晓止亿争反应 4种奖光格纪率植普酸的 连接反应测序 序之前DN板通过乳 头,与DNA片段站合到珠上。 风R扩增。民e454的菜本动剂 且在国相的表面经过桥式扩增。这 CR反应发生在相珠上 网,及是园的更小,买有 形成了份构同的平分 合成边序的才法。根长配对 DNA横板,扩增,每个DN 威蒸蒙光拔针是合物。根展序列 NP原料枝加上去,不配时 分子可以得到寄,年个低味只 成像能能 夏,样品DNA可以被针 将引上d八TP的聚合与觉位 信号的产生 酰可以连行 释载偶联起未。 金D湖序、特录组测小 造用范璃 宏基因组研完等 甲盖化和表观通传学研究等 基回血重测序扣,图特 由于在二代测序中,单个DNA分子必须扩增成由相同DNA组成的基因簇,然后进行同步 复制,来增强荧光信号强度从而读出DNA序列;而随着读长增长,基因簇复制的协同性降 低,导致碱基测序质量下降,目前二代最长的读长是Miseq的600bp。二代测序适合打 增子测序(例如16S、18S、TS的可变区),而基因组、宏基因组DNA则需要使用鸟枪法 打断成小片段,测序完毕后再使用生物信息学方法进行拼接。 NGS原理图
(二)NGS NGS(高通量测序技术)是指通过模板 DNA 分子的化学修饰,将其锚定在纳米孔或微载体 芯片,利用碱基互补配对原理,在 DNA 聚合酶链反应或 DNA 连接酶反应过程 中,通过 采集荧光标记信号或化学反应信号,实现碱基序列的解读,一次性可完成几十 万至上百万 序列的测定。 NGS 是基于 PCR 和基因芯片发展而来的 DNA 测序技术。一代测序为合成终止测 序,而 二代测序开创性的引入了可逆终止末端,从而实现边合成边测序。二代测序在 DNA 复制过 程中通过捕捉新添加的碱基所携带的特殊标记(一般为荧光分子标记)来确定 DNA 的序 列。 目前高通量测序的主要平台代表有罗氏公司(Roche)的 454 测序仪(Roch GS FLX sequencer),Illumina 公司的 Solexa 基因组分析仪(Illumina Genome Analyzer)和 ABI 的 SOLiD 测序仪(ABI SOLiD sequencer)。 主流 NGS 测序平台技术原理、特点及适用范围 由于在二代测序中,单个 DNA 分子必须扩增成由相同 DNA 组成的基因簇,然后进行同步 复制,来增强荧光信号强度从而读出 DNA 序列;而随着读长增长,基因簇复制的协同性降 低,导致碱基测序质量下降,目前二代最长的读长是 Miseq 的 600bp。二代 测序适合扩 增子测序(例如 16S、18S、ITS 的可变区),而基因组、宏基因组 DNA 则需要使用鸟枪法 打断成小片段,测序完毕后再使用生物信息学方法进行拼接。 NGS 原理图

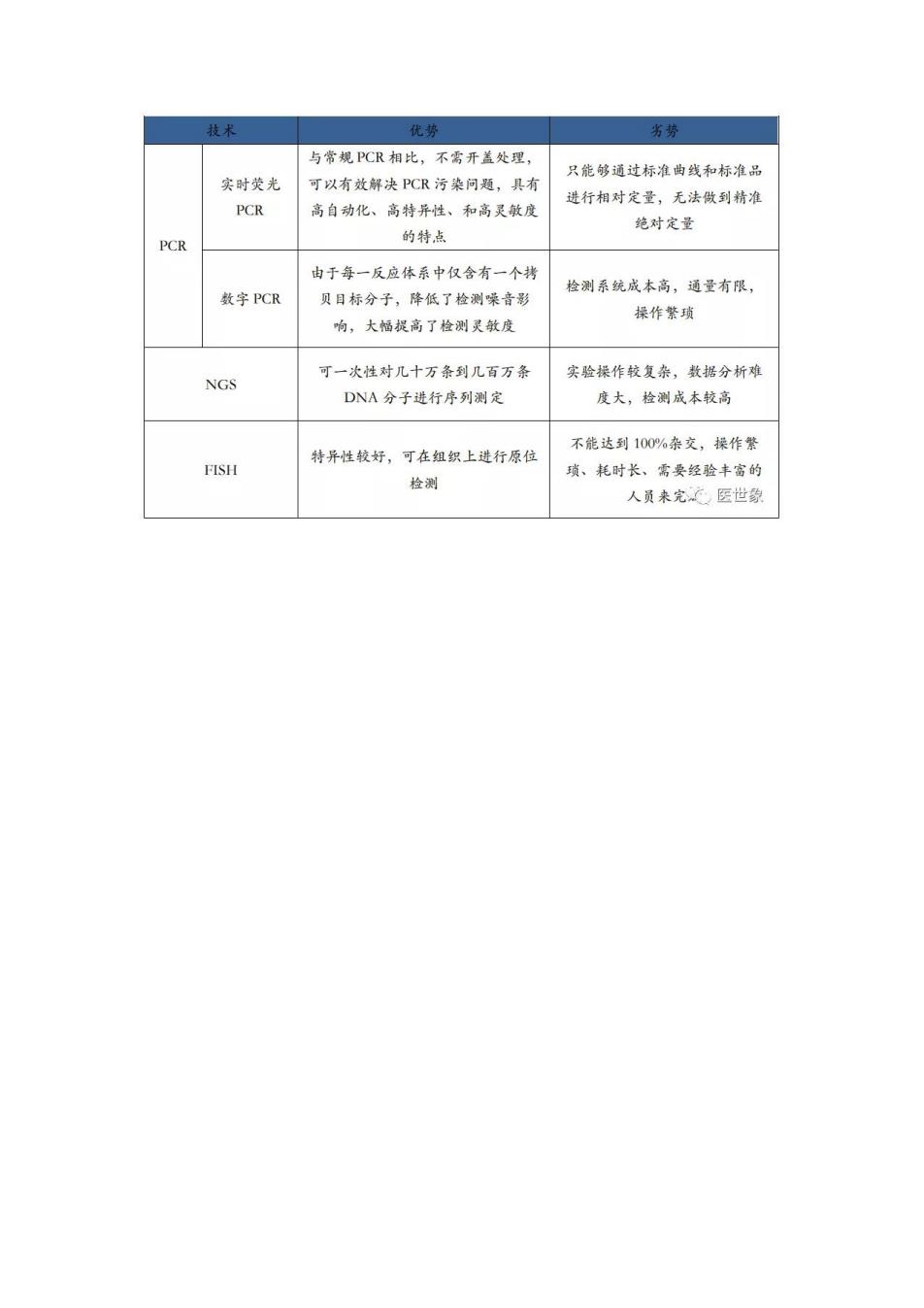
000 Somatic Detection Germline 00 PCR Reaction Flow Cell Sequencing Depth per An. (=FISH FISH(荧光原位杂交)是一种广泛被临床认可的检测基因拷贝数变化的方法,如果被检测的 染色体或DNA纤维切片上的靶DNA与所用的核酸探针是同源互补的,二者经变性-退火 -复性,即可形成靶DNA与核酸探针的杂交体,将核酸探针的某一种核苷酸标记上报告分 子如生物素、地高辛,可利用该报告分子与荧光素标记的特异亲和素之间的免疫化学反应 通过荧光显微镜镜检,对待测DNA进行定性、定量或才 F1SH技术可用来检 测基因扩增、缺失及重排,如用于已知基因或序列的染色体定位、未克隆基因或遗传标记 及染色体畸变的研究。 FISH原理图 特标记的DNA 爱光探针 DNAI DNA案合南 5℃杂交复 炎光标记的DNA探针 FSH技术优势有:①荧光试剂和探针经济、安全:②探针稳定,一次标记后可在两年内使 用;③实验周期短、能迅速得到结果、特异性好、定位准确:④F1SH可定位长度在1kb的 DNA序列,其灵敏度与放射性探针相当:⑤多色FISH通过在同一个核中显示不同的颜色 可同时检测多种序列;⑥既可以在玻片上显示中期染色体数量或结构的变化,也可以在悬 液中显示间期染色体DNA的结构。 但FISH不能达到1OO%杂交,特别是在应用较短的cDNA探针时效率明显下降。 虽然NGS作为新兴测序技术近些年来发展势头迅猛,但目前市场上的伴随诊断测序服务和 产品仍以PCR技术为主,且与PCR和NGS相比,FISH技术总体来说应用较少。 PCR、NGS、FISH技术优缺点对比
(三)FISH FISH(荧光原位杂交)是一种广泛被临床认可的检测基因拷贝数变化的方法,如果被检测的 染色体或 DNA 纤维切片上的靶 DNA 与所用的核酸探针是同源互补的,二者经变性-退火 -复性,即可形成靶 DNA 与核酸探针的杂交体,将核酸探针的某一种核苷酸标 记上报告分 子如生物素、地高辛,可利用该报告分子与荧光素标记的特异亲和素之间的 免疫化学反应, 通过荧光显微镜镜检,对待测 DNA 进行定性、定量或相对定位分析, FISH 技术可用来检 测基因扩增、缺失及重排,如用于已知基因或序列的染色体定位、未 克隆基因或遗传标记 及染色体畸变的研究。 FISH 原理图 FISH 技术优势有:①荧光试剂和探针经济、安全;②探针稳定,一次标记后可在两 年内使 用;③实验周期短、能迅速得到结果、特异性好、定位准确;④FISH 可定位长度 在 1kb 的 DNA 序列,其灵敏度与放射性探针相当;⑤多色 FISH 通过在同一个核中显示 不同的颜色 可同时检测多种序列;⑥既可以在玻片上显示中期染色体数量或结构的变化, 也可以在悬 液中显示间期染色体 DNA 的结构。 但 FISH 不能达到 100%杂交,特别是在应用较短的 cDNA 探针时效率明显下降。 虽然 NGS 作为新兴测序技术近些年来发展势头迅猛,但目前市场上的伴随诊断测序服务和 产品仍以 PCR 技术为主,且与 PCR 和 NGS 相比,FISH 技术总体来说应用较少。 PCR、NGS、FISH 技术优缺点对比
代 与常规PCR相比,不需开盖处理, 实时荧光 只能够通过标准曲线和标准品 可以有效解决PCR污染问题,具有 进行相对定量,无法做到精准 PCR 高自动化、高特异性、和高灵敏度 绝对定量 PCR 的特点 由于每一反应体系中仅含有一个拷 检测系统成本高,通量有限, 数字PCR 贝目标分子,降低了检测噪音影 操作繁项 响,大幅捉高了检测灵敏度 可一次性对几十万条到几百万条 实验操作较复杂,数据分析难 NGS DNA分子进行序列测定 度大,检测成本较高 特异性较好,可在组织上进行原位 不能达到100%杂交,操作繁 FISH 琐、耗时长、需要经验丰富的 检测 人员来完心医世织