
基因编辑技术
基因编辑技术

·基因编辑是指对基因组进行定点修饰的一项新技术。利用该技术,可以精确地定 位到基因组的某一位点上,在这位点上剪断靶标DA片段并插入新的基因片段。 此过程既模拟了基因的自然突变,又修改并编辑了原有的基因组,真正达成了 “编辑基因”。 ·目前主要有3种基因编辑技术,分别为: a)人工核酸酶介导的锌指核酸酶(zinc-finger nucleases,ZFN)技术; b)转录激活因子样效应物核酸酶(transcription activator-like effector nucleases,TALEN)技术; c)RNA引导的CRISPR-Cas核酸酶技术(clustered regulatory interspaced short pal indromic repeat/Cas-based RNA-guided DNA endonucleases,CRISPR/Cas )
• 基因编辑是指对基因组进行定点修饰的一项新技术。利用该技术,可以精确地定 位到基因组的某一位点上, 在这位点上剪断靶标 DNA 片段并插入新的基因片段。 此过程既模拟了基因的自然突变, 又修改并编辑了原有的基因组, 真正达成了 “编辑基因” 。 • 目前主要有 3 种基因编辑技术, 分别为: a)人工核酸酶介导的锌指核酸酶(zinc-finger nucleases,ZFN)技术; b)转录激活因子样效应物核酸酶(transcription activator-like effector nucleases,TALEN)技术; c)RNA引导的CRISPR-Cas核酸酶技术(clustered regulatory interspaced short palindromic repeat/Cas-based RNA-guided DNA endonucleases,CRISPR/Cas )

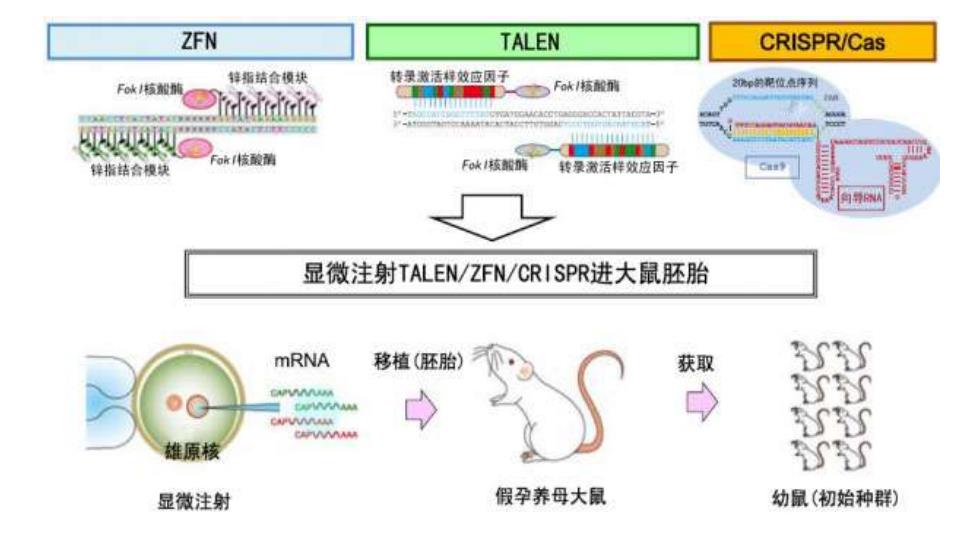
1.ZFN基因编辑技术 ZFN技术是第一代基因组编辑技术,其功能的实现是基于具有独特的DNA 序列识别的锌指蛋白发展起来的。ZFN由特异性识别序列的锌指蛋白(ZFP) 和Fok丨核酸内切酶组成。其中,由ZFP构成的DNA识别域能识别特异位点 并与之结合,而由Fok丨构成的切割域能执行剪切功能,两者结合可使靶位 点的双链DNA断裂。于是,细胞可以通过同源重组(HR)修复机制和非同源末 端连接(NHEJ)修复机制来修复DNA。HR修复有可能会对靶标位点进行恢复修 饰或者插入修饰,而NHEJ修复极易发生插入突变或缺失突变。两者都可造 成移码突变,因此达到基因敲除的目的
1.ZFN基因编辑技术 ZFN 技术是第一代基因组编辑技术,其功能的实现是基于具有独特的DNA 序列识别的锌指蛋白发展起来的。ZFN由特异性识别序列的锌指蛋白(ZFP) 和 FokⅠ核酸内切酶组成。其中,由ZFP构成的DNA识别域能识别特异位点 并与之结合,而由FokⅠ构成的切割域能执行剪切功能,两者结合可使靶位 点的双链DNA断裂。于是,细胞可以通过同源重组(HR)修复机制和非同源末 端连接(NHEJ)修复机制来修复DNA。HR修复有可能会对靶标位点进行恢复修 饰或者插入修饰,而NHEJ修复极易发生插入突变或缺失突变。两者都可造 成移码突变,因此达到基因敲除的目的

2.TLEN基因编辑技术 TLE(Transcription activator-like effectors):一种源于植物 致病菌Kanthomonas sp..的靶向基因操作技术。TALENs包含两个TALEN蛋白, 每个TALEN都是由TALE array与FokI融合而成。其中一个TALEN靶向正义 链上靶标位点,另一个则靶向反义链上的靶标位点.然后Fk〡形成二聚体, 在靶向序列中间的spacer处切割DA,造成双链DNA断裂,随后细胞启动DNA 损伤修复机制。针对不同的TLEN骨架,其最适宜的spacer长度不同,其长 度范围一般为12-20bP。实验结果表明,TALENs在靶向DNA时,第一个碱基 为T时其结合效果更佳
2.TALEN基因编辑技术 TALE(Transcription activator-like effectors):一种源于植物 致病菌Xanthomonas sp.的靶向基因操作技术。TALENs包含两个TALEN蛋白, 每个TALEN都是由TALE array与FokⅠ融合而成。其中一个TALEN靶向正义 链上靶标位点,另一个则靶向反义链上的靶标位点. 然后FokⅠ形成二聚体, 在靶向序列中间的spacer处切割DNA, 造成双链DNA断裂,随后细胞启动DNA 损伤修复机制。针对不同的TALEN骨架,其最适宜的spacer长度不同,其长 度范围一般为12-20 bp。实验结果表明,TALENs在靶向DNA时,第一个碱基 为T时其结合效果更佳

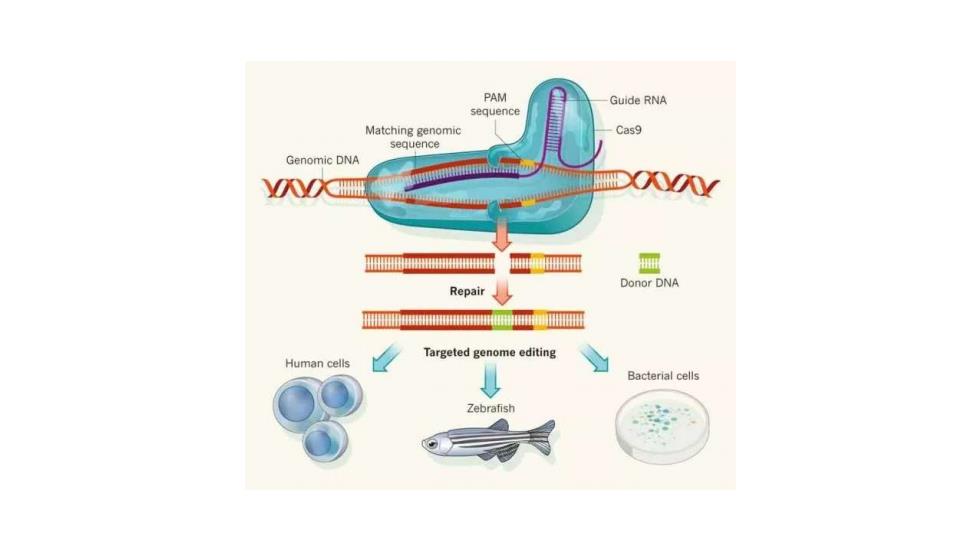
3.CRISPR/Cas9基因组编辑技术 CRISPR/Cas系统由Cas9核酸内切酶与sgRA构成。转录的sgRA折叠成特定的 三维结构后与Cas9蛋白形成复合体,指导Cas9核酸内切酶识别特定靶标位点,在 PAM序列上游处切割DNA造成双链DNA断裂,并启动DNA损伤修复机制。从不同菌种 中分离的CRISPR/Cas系统,其CrRNA(或者是人工构建的SgRNA)靶向序列的长度不 同,PAM序列也可能不同。最新报道,CRISPR-C2c2系统能够根据靶向序列特异地 编辑单链NA
3.CRISPR /Cas9 基因组编辑技术 CRISPR/Cas系统由Cas9核酸内切酶与sgRNA构成。转录的sgRNA折叠成特定的 三维结构后与Cas9蛋白形成复合体,指导Cas9核酸内切酶识别特定靶标位点,在 PAM序列上游处切割DNA造成双链DNA断裂, 并启动DNA损伤修复机制。从不同菌种 中分离的CRISPR/Cas系统,其CrRNA(或者是人工构建的sgRNA)靶向序列的长度不 同,PAM序列也可能不同。最新报道,CRISPR-C2c2系统能够根据靶向序列特异地 编辑单链RNA
PAM Guide RNA sequence Matching genomic Cas9 sequence Genomic DNA 入a DX0X HH 画 Donor DNA Repair 用 Targeted genome editing Human cells Bacterial cells Zebrafish
ZFN TALEN CRISPR/Cas 锌指结合模块 转录藏活样效应因子 FOk核城的 Fak核酸 2光和的泥位点等列 iiririr 于BD 锌指结合模块 Fak核酸酯 Fk核裤 转录激活样效应因子 显微注射TALEN/ZFN/CRISPR进大鼠胚胎 mRNA 移植(胚胎) 获取 S CAVAA 灯 CAIVU 雄原核 显微注射 假孕养母大鼠 幼鼠(初始种群)

1.共同点 结构的相似性:无论是ZFN、TALEN还是CRISPR/,Cas9,都是由DNM识别域和核酸内切酶 两部分组成。其中ZFN技术具有锌指结构域能够识别靶点DNM,而TALENE的DNA识别区域 是重复可变双残基的重复,DA剪切区域都是一种名为FokI的核酸内切酶结构域。 CRISPR的DNA识别区域是crRNAT或向导RNM,Cas9蛋白负责DNA的剪切。当DNA结合域识 别靶点DNA序列后,核酸内切酶或Cas9蛋白将DA剪切,靶DNA双链断裂,再启动DNA损 伤修复机制,实现基因敲除、插入等。 作用过程的相似性:通过DNA识别模块对特异性的DNA位点识别并结合,然后在 相关核酸内切酶的作用下完成特定位点的剪切,从而借助于细胞内固有的HD(同源 定向修复)或NHEJ(非同源末端连接)途径修复过程完成特定序列的插入、删除及基 因融合
1.共同点 结构的相似性:无论是ZFN、TALEN还是CRISPR/Cas9,都是由DNA识别域和核酸内切酶 两部分组成。其中ZFN技术具有锌指结构域能够识别靶点DNA,而TALEN的DNA识别区域 是重复可变双残基的重复,DNA剪切区域都是一种名为Fokl的核酸内切酶结构域。 CRISPR的DNA识别区域是crRNA或向导RNA,Cas9蛋白负责DNA的剪切。当DNA结合域识 别靶点DNA序列后,核酸内切酶或Cas9蛋白将DNA剪切,靶DNA双链断裂,再启动DNA损 伤修复机制,实现基因敲除、插入等。 作用过程的相似性:通过DNA识别模块对特异性的DNA位点识别并结合,然后在 相关核酸内切酶的作用下完成特定位点的剪切,从而借助于细胞内固有的HDR(同源 定向修复)或NHEJ(非同源末端连接)途径修复过程完成特定序列的插入、删除及基 因融合
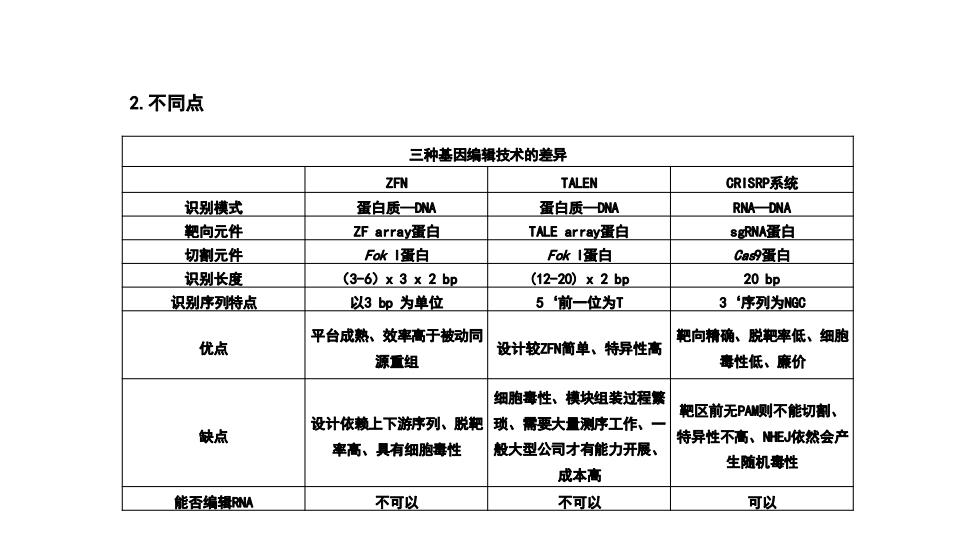
2.不同点 三种基因编辑技术的差异 ZFN TALEN CRISRP系统 识别模式 蛋白质DM 蛋白质DM RNADNA 靶向元件 ZF array蛋自 TALE arrayi蛋自 sNM蛋自 切剂元件 FokI蛋白 F冰I蛋自 Cas9蛋白 识别长度 (3-6)x3x2bp (12-20)x2bp 20 bp 识别序列特点 以3bp为单位 5前一位为T 3“序列为NGC 平台成熟、效率高于被动同 靶向精确、脱靶率低、细胞 优点 设计较ZN简单、特异性高 源重组 毒性低、廉价 细胞毒性、模块组装过程繁 靶区前无PAM侧不能切割、 设计依赖上下游序列、脱靶 琐、需要大量测序工作、一 缺点 率高、具有细胞毒性 般大型公司才有能力开展、 特异性不高、MEJ依然会产 生随机毒性 成本高 能香编辑RNM 不可以 不可以 可以
2.不同点 三种基因编辑技术的差异 ZFN TALEN CRISRP系统 识别模式 蛋白质—DNA 蛋白质—DNA RNA—DNA 靶向元件 ZF array蛋白 TALE array蛋白 sgRNA蛋白 切割元件 Fok I蛋白 Fok I蛋白 Cas9蛋白 识别长度 (3-6)x 3 x 2 bp (12-20) x 2 bp 20 bp 识别序列特点 以3 bp 为单位 5‘前一位为T 3‘序列为NGC 优点 平台成熟、效率高于被动同 源重组 设计较ZFN简单、特异性高 靶向精确、脱靶率低、细胞 毒性低、廉价 缺点 设计依赖上下游序列、脱靶 率高、具有细胞毒性 细胞毒性、模块组装过程繁 琐、需要大量测序工作、一 般大型公司才有能力开展、 成本高 靶区前无PAM则不能切割、 特异性不高、NHEJ依然会产 生随机毒性 能否编辑RNA 不可以 不可以 可以

在这套系统里,Cas9蛋白会在目标DNA序列上切出口子,造成双链NM断裂。 随后,再利用同源重组等方法,在DA修复的过程中对基因组进行编辑。 这一方法虽然有效,但很粗暴。打个比方,这就好像一页书上有错别字, 为了修正书上的错误,要把这整页书给撕下来,再重新粘上一页那样。可 以想象,这样的工作谈不上优雅,也容易产生潜在的问题
在这套系统里,Cas9蛋白会在目标DNA序列上切出口子,造成双链DNA断裂。 随后,再利用同源重组等方法,在DNA修复的过程中对基因组进行编辑。 这一方法虽然有效,但很粗暴。打个比方,这就好像一页书上有错别字, 为了修正书上的错误,要把这整页书给撕下来,再重新粘上一页那样。可 以想象,这样的工作谈不上优雅,也容易产生潜在的问题