
非小细胞肺 癌和EGFR
非小细胞肺 癌和EGFR

疾病概述 ●●●● 肺癌是世界上最常见的恶性肿瘤之一,已成为我国城市人口恶性肿 瘤死亡原因的第1位。 非小细胞肺癌是起源于支气管黏膜、支气管腺体和肺泡上皮的一类 肺恶性肿瘤,显微镜下特点是核异形、细胞较大、胞浆丰富。 根据组织病理学分类可分为腺癌、鳞状细胞癌、腺鳞癌、大细胞癌 及肉瘤样癌等亚型。 在我国,非小细胞肺癌为最常见的肿瘤。 据2013年肺癌相关的调查报道,肺癌新发病及死亡数量为恶性肿瘤 首位,其中,非小细胞肺癌约占所有肺癌的85%,男性发病率与死 亡率均高于女性非小细胞型肺癌包括鳞状细胞癌(鳞癌)、腺癌、 大细胞癌,与小细胞癌相比其癌细胞生长分裂较慢,扩散转移相对 较晚。非小细胞肺癌约占所有肺癌的80%,约75%的患者发现时已处 于中晚期,5年生存率很低
1 疾病概述 肺癌是世界上最常见的恶性肿瘤之一,已成为我国城市人口恶性肿 瘤死亡原因的第1位。 非小细胞肺癌是起源于支气管黏膜、支气管腺体和肺泡上皮的一类 肺恶性肿瘤,显微镜下特点是核异形、细胞较大、胞浆丰富。 根据组织病理学分类可分为腺癌、鳞状细胞癌、腺鳞癌、大细胞癌 及肉瘤样癌等亚型。 在我国,非小细胞肺癌为最常见的肿瘤。 据2013年肺癌相关的调查报道,肺癌新发病及死亡数量为恶性肿瘤 首位,其中,非小细胞肺癌约占所有肺癌的85%,男性发病率与死 亡率均高于女性非小细胞型肺癌包括鳞状细胞癌(鳞癌)、腺癌、 大细胞癌,与小细胞癌相比其癌细胞生长分裂较慢,扩散转移相对 较晚。非小细胞肺癌约占所有肺癌的80%,约75%的患者发现时已处 于中晚期,5年生存率很低
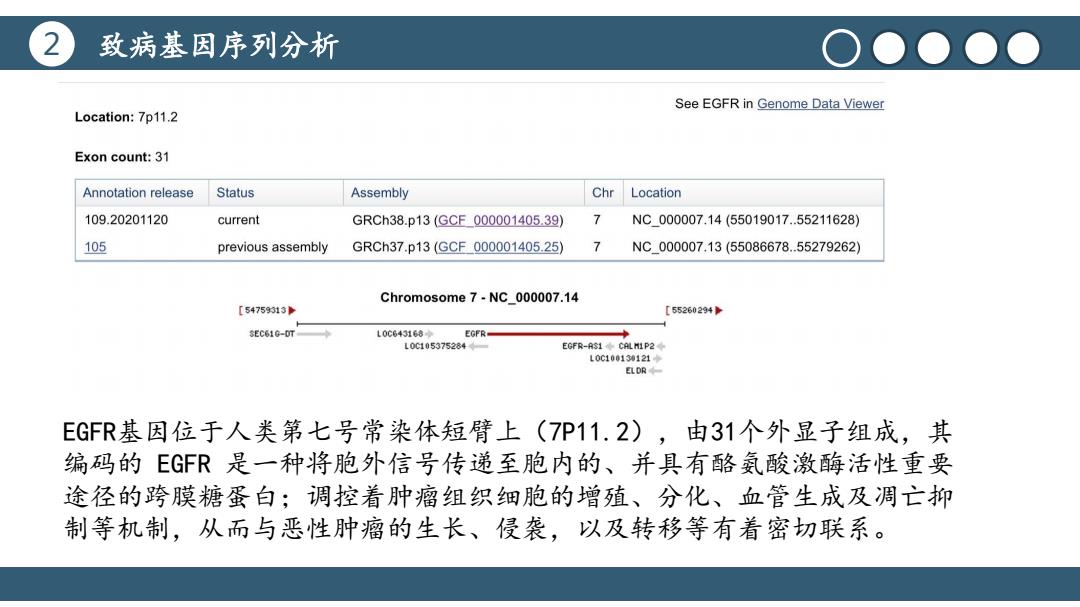
致病基因序列分析 O●●●● See EGFR in Genome Data Viewer Location:7p11.2 Exon count:31 Annotation release Status Assembly Chr Location 109.20201120 current GRCh38.p13(GCF000001405.39) NC_000007.14(55019017.55211628) 105 previous assembly GRCh37.p13(GCF000001405.25) NC000007,13(55086678.55279262) Chromosome 7-NC_000007.14 [54759313 [55260294 SEC610-DT L0c643168 EGFR L0C1053752844 EGFR-A31◆CALM1P2◆ L00100130121◆ ELDR EGFR基因位于人类第七号常染体短臂上(7P11.2),由31个外显子组成,其 编码的EG℉是一种将胞外信号传递至胞内的、并具有酪氨酸激酶活性重要 途径的跨膜糖蛋白;调控着肿瘤组织细胞的增殖、分化、血管生成及调亡抑 制等机制,从而与恶性肿瘤的生长、侵袭,以及转移等有着密切联系
2 致病基因序列分析 标题文本预设 点击此处更换文本点击此处更换文本点 击此处更换文本 标题文本预设 点击此处更换文本点击此处更换文本点 击此处更换文本 标题文本预设 点击此处更换文本点击此处更换文本点 击此处更换文本 EGFR基因位于人类第七号常染体短臂上(7P11.2),由31个外显子组成,其 编码的 EGFR 是一种将胞外信号传递至胞内的、并具有酪氨酸激酶活性重要 途径的跨膜糖蛋白;调控着肿瘤组织细胞的增殖、分化、血管生成及凋亡抑 制等机制,从而与恶性肿瘤的生长、侵袭,以及转移等有着密切联系

致病基因序列分析 O●●●● 104 120M 30M 90H 50M 70M 180M 90M 100M 1O M 120M 130M 140M 159,345973 白号Nc0o0007.14~|Fnd v→Q Q目 入Tools,◆Tracks~是Download,e?, 55H 155020K 55,040K 55,860K s5.880K s5100K 55120K 55140K 551680K $180K S5200K 55228K NCBI Homo sapiens Updated Annotation Release 109.20201120 on GRCh38... EGFR HH.005228.5H +HHP©052192 HH0013468992 HP001333828.1 HH0013469412 =P013338781 1H013468982 HHP.901333827.1 HH0013468972 HHNP0013338261 H2012842 十H十十十P9584411 H2012822 十HH十十一NP9584391 N2812832 1十P958441 00134690021 11P.0013338291 L0105375284 EGFR-ASI L0c10e1301 ■R0017452122 ■047551山 mR9272722 S6H8唱n9ate,NCBI Homo sap1 ens Updated Annotation- 上0× Genes,Ensembl release 102 0g 40× Es600000146648L- 计→州) Es60e000224e57 ■ ☐+BsT6000044241 456000602377 EHsT000042593
2 致病基因序列分析 标题文本预设 点击此处更换文本点击此处更换文本点 击此处更换文本 标题文本预设 点击此处更换文本点击此处更换文本点 击此处更换文本 标题文本预设 点击此处更换文本点击此处更换文本点 击此处更换文本

致病基因序列分析 ●●●● Assembly AGACGTCCGGGCAGCCCCCGGCGCAGCGCGGCCGCAGCAGCCTCCGCCCCCCGCACGGTGTGAGCGCCCG GAGACTTTCTTTCTTGGATGTCTCTTTTTGCTGTTTGAAGAATTTGAGCCAACCAAAATATTAAACCTGT ACGCGGCCGAGGCGGCCGGAGTCCCGAGCTAGCCCCGGCGGCCGCCGCCGCCCAGACCGGACGACAGGCC CTTACACACACACACACACACACACACACACACACACCGGATTGCTGTCCCTGGTTCAAGTGTGCCAAGT ACCTCGTCGGCGTCCGCCCGAGTCCCCGCCTCGCCGCCAACGCCACAACCACCGCGCACGGCCCCCTGAC GTGCAGACAGAACATGAGCGAGTCTGGCTTCGTGACTACCGACCATAAACCCACTTGACAGGGGAAACAT TCCGTCCAGTATTGATCGGGAGAGCCGGAGCGAGCTCTTCGGGGAGCAGCGATGCGACCCTCCGGGACGG GCCTTGGAAGGTTTAATTGCACAATTCCAACCTTGAGCTGCGCGGGTTCCAAGAGCCAGGCCCGTACTTG CCGGGGCAGCGCTCCTGGCGCTGCTGGCTGCGCTCTGCCCGGCGAGTCGGGCTCTGGAGGAAAAGAAAGG CTGTTGATGTCATTGGCTTGGGGAGTTGGGGTTTGGTGCCCAGCGCGGTCGTTGGGGGAGGGGCAAGGCA TAAGGGCGTGTCTCGCCGGCTCCCGCGCCGCCCCCGGATCGCGCCCCGGACCCCGCAGCCCGCCCAACCG TAGAACAGTGGTTCCCAGACCTTGCTGCACATTGGAATTACCTGGGATTAAAAAAAAAAAAATCAAAACA CGCACCGGCGCACCGGCTCGGCGCCCGCGCCCCCGCCCGTCCTTTCCTGTTTCCTTGAGATCAGCTGCGC AAAACCAGTGTCTGGCTCCCGCCCCCAGACATTCTGATTTAATTGGCATGGGGCAAGACCTGGACTTGGG CGCCGACCGGGACCGCGGGAGGAACGGGACGTTTCGTTCTTCGGCCGGGAGAGTCTGGGGCGGGCGGAGG ATTTTTTTTAATGCTCTTCATGTGATCTGTTGGGCAGCCAGATTTGGGGATCACTAGACGGAAGAAGGAT AGGAGACGCGTGGGACACCGGGCTGCAGGCCAGGCGGGGAACGGCCGCCGGGACCTCCGGCGCCCCGAAC TGTTAAAGTCTCCGGAGATGTTACTTGCCAATGCTAAGAGCTCTTTGAGGACATCTGGAATTGTTACAAT CGCTCCCAACTTTCTTCCCTCACTTTCCCCGCCCAGCTGCGCAGGATCGGCGTCAGTGGGCGAAAGCCGG ATTGCCAAATATAGGAAAGAGGGAAAAGGTAGAGTGTGATTCCAATAATAAAGGATTCCGCTTTTCATTG GTGCTGGTGGGCGCCTGGGGCCGGGGTCCCGCACGTGCGCCCCGCGCTGTCTTCCCAGGGCGCGACGGGG AAGGAACTGGTGGAAAGGTTTCTTCTCTGCTGAGCCTGCAGGCCCGTCCTGCCTGCCTGGGGTGCCCGGG TCCTGGCGCGCACCCGAGGGGCGGGCGCTGCCCACCCGCCGAGACTGCACTGTTTAGGGAAGCTGAGGAA AGACGCGGGCCTGCTCCGGAGACTGCTGACTGCCGGTCCTGTTAGTCAGGTGTCAGCCCTGTCTCTGCCG GGAACCCAAAAATACAGCCTCCCCTCGGACCCCGCGGGACAGGCGGCTTTCTGAGAGGACCTCCCCGCCT AAGAGACTCTTCTCTTTATTTTAAATTAAACCCTCAGAGCACCACCAAAGCATCACTTTTCTCCCTCCAT CCGCCCTCCGCGCAGGTCTCAAACTGAAGCCGGCGCCCGCCAGCCTGGCCCCGGCCCCTCTCCAGGTCCC TGGTGTTCTCATTCTTTGATGTTACTTGTTTGAACACCACTATTAGTAGTTGGAGATTTGTTCCTGAGAA AAATATAAATACCACTTAATTTGCCTGTTTGTCCCGCATTCACTCAAAACAGAATGCTCCTGAAGACAAG CGCGATCCTCGTTCCCCAGTGTGGAGTCGCAGCCTCGACCTGGGAGCTGGGAGAACTCGTCTACCACCAC AGAGAGAGTAGGAGAACAGACGCTATTCCATTACAGTAACATAAAAGACTGGATTTTCAGGGGCAAATTA CTGCGGCTCCCGGGGAGGGGTGGTGCTGGCGGCGGTTAGTTTCCTCGTTGGCAAAAGGCAGGTGGGGTCC TTAAAATAGGAGATGAGCTCTTTTAACAGAAATTTGTTTAAGGCCTGTGTCTATCAAATTCAGTGGATTT GACCCGCCCCTTGGGCGCAGACCCCGGCCGCTCGCCTCGCCCGGTGCGCCCTCGTCTTGCCTATCCAAGA TATTCAAGATGCACTTTGTTTAGTGGGAGTTTTGTTTGGTTCTGGGACATGCTAACTTCTAGACTTGCTG GTGCCCCCCACCTCCCGGGGACCCCAGCTCCCTCCTGGGCGCCCGCGCCGAAAGCCCCAGGCTCTCCTTC CTCTTAGAGGTAATGACTGCCAGACACCATTTCATGAGTCCTAATCCCCACATTAAGCATAAGAGGTGCA GATGGCCGCCTCGCGGAGACGTCCGGGTCTGCTCCACCTGCAGCCCTTCGGTCGCGCCTGGGCTTCGCGG CACTCTCCTCCTATGGGGGAAACTGAGGTACGAAGAACTAAAGTGACTTTCCCACAGCTGGTGGGAGGCA TGGAGCGGGACGCGGCTGTCCGGCCACTGCAGGGGGGGATCGCGGGACTCTTGAGCGGAAGCCCCGGAAG GACGGGAAATTCACACCAGGGGCTTCCAACTCCAGATCCCTCTCTCAACTTCCAAACTCCACTGCCTTGT CAGAGCTCATCCTGGCCAACACCATGGTGTTTCAAAATGGGGCTCACAGCAAACTTCTCCTCAAAACCCG CCGAGTTCTGGTTTCAGGAGATCCAAATCAGGTGTGTGCAAATGTCTAATGTCAGAGCTGGCAAGGGGAA AGGGCCCAGGGAGCCGGCTCATGACGATGAGCCTGTCTGAAGCTTCAACGCGGGCTGTCCGGCAGTCTGC ATTCCTGCCGAGTTCCTCAGCCCTCTGTTGGGTCACCTTCCATAGAGGCAGCTTAGTCCTCAGTTCAGTG AGCATGGAGTGGAGACTGCTTGAGGGGTGCTGAGCAAAGCCCTGCCTCTTACAGGATGAAGGTGCTCTCC
2 致病基因序列分析

致病基因序列分析 O●●●● ##Genome-Annotation-Data-END## FEATURES Location/Qualifiers source 1..192612 /organism="Homo sapiens" /mol_type="genomic DNA" /db xref="taxon:9606" /chromosome="7" gene complement(<1..192) /gene="L0C105375284" /note="Derived by automated computational analysis using gene prediction method:Gnomon." /db_xref="GeneID:105375284" /db_×ref="MIM:131550" mRNA j01n(1..349,123270..123421,124289..124472,127590,,127724, 132278..132346,133530..133648,134995,.135136, 136814..136930,137517..137643,137743..137816, 138647..138737,141123..141322,142483..142615, 144717..144807,146264..146421,152159..152197 153967..154108,154905.,155027,155706..155804, 162277..162462,172703..172858,173750..173825, 179701..179847,181300..181397,182172..182339, 182719..182766,183501..183609,186240..192612) /gene="EGFR" /gene_synonym="ERBB;ERBB1;HER1;mENA;NISBD2;PIG61
2 致病基因序列分析 标题文本预设 点击此处更换文本点击此处更换文本点 击此处更换文本 标题文本预设

致病基因序列分析 O●● ●● 突变类型: EGFR基因外显子19突变为最常见的突变类型之一,主要发生在密码子第746~750位 的碱基缺失,而导致由它们所编码的蛋白丢失。 ①E746-A750(1)为外显子19的最常见突变类型,共检测出4例,主要由碱基 序列c.2234至2249共16个碱基缺失,从而导致由密码子第746~750位所编码EGFR蛋 白所需的氨基酸(ELREA)丢失。 ②E746-A750(2)所缺失的15个碱基主要发生在碱基序列c.2235至c.2249,但由 密码子编码的氨基酸丢失与类型1相同。 ③E746-A750(3)所缺失的15个碱基较类型3后移一位,但由此碱基丢失引起的氨 基酸丢失与类型1相同。 ④E746-A751所缺失16个碱基发生在碱基序列c.2236至c.2251,相应的密码子第 746~751位所编码的氨基酸(EL-REAT)丢失。 ⑤L747-P753insS所缺失的18个碱基序列发生在碱基序列c.2240至c.2257,从而导 致密码子第747~753位所编码的氨基酸(LREATSP)丢失,同时由密码子753所编 码的脯氨酸(Pro)由新引入的丝氨酸(Ser)所替代
2 致病基因序列分析 标题文本预设 点击此处更换文本点击此处更换文本点 击此处更换文本 标题文本预设 点击此处更换文本点击此处更换文本点 击此处更换文本 标题文本预设 点击此处更换文本点击此处更换文本点 击此处更换文本 突变类型: EGFR基因外显子19突变为最常见的突变类型之一,主要发生在密码子第746~750位 的碱基缺失,而导致由它们所编码的蛋白丢失。 ①E746- A750(1)为外显子 19 的最常见突变类型,共检测出4 例,主要由碱基 序列c.2234至2249共16个碱基缺失,从而导致由密码子第746~750位所编码EGFR蛋 白所需的氨基酸(ELREA)丢失。 ②E746- A750(2)所缺失的15个碱基主要发生在碱基序列c.2235至 c.2249,但由 密码子编码的氨基酸丢失与类型1相同。 ③E746-A750(3)所缺失的15个碱基较类型3后移一位,但由此碱基丢失引起的氨 基酸丢失与类型1相同。 ④E746-A751所缺失16个碱基发生在碱基序列c.2236至c.2251,相应的密码子第 746 ~751位所编码的氨基酸(EL-REAT)丢失。 ⑤L747-P753insS所缺失的18个碱基序列发生在碱基序列c.2240至c.2257,从而导 致密码子第747 ~753位所编码的氨基酸(LREATSP)丢失,同时由密码子753所编 码的脯氨酸(Pro)由新引入的丝氨酸(Ser)所替代
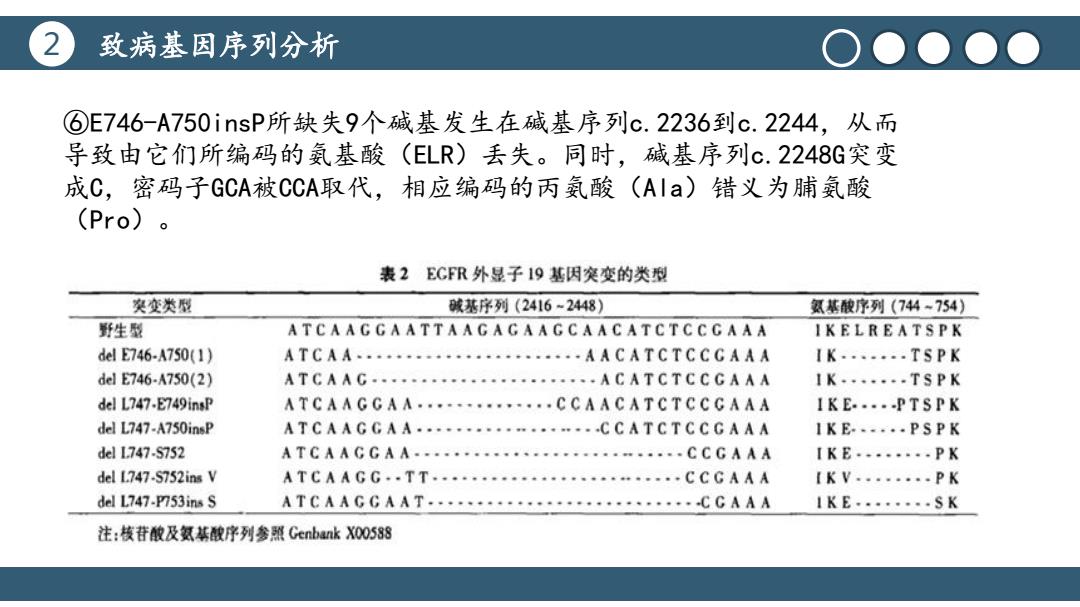
致病基因序列分析 ●)● ●●● ⑥E746-A750insP所缺失9个碱基发生在碱基序列c.2236到c.2244,从而 导致由它们所编码的氨基酸(ELR)丢失。同时,碱基序列c.2248G突变 成C,密码子GCA被CCA取代,相应编码的丙氨酸(AIa)错义为脯氨酸 (Pro)。 表2EGFR外显子19基因突变的类型 突变类型 碱基序列(2416-2448)】 氨基酸序列(744-754)】 野生型 ATCAAGGAATTAACAGAAGCAACATCTCCCAAA IKELREATSPK ddE746-A750(1) ATCAA.···AACATCTCCCAAA IK.......TSPK ddE746-A750(2)】 ATCAAC…··· ·····ACATCTCCCAAA IK.·,,·.TSPK del L747-E749insP ATCAAGCA A..----.--.---..CCAACATCTCCCAAA IKE-----PTSPK del L747-A750insP ATCAAGCAA····· …··CCATCTCCGAAA 1KE..,·,PSPK del1747.S752 ATCAACCA A······.· =·····CCGAAA IKE,·,··,PK del L747-S752ins V ATCAAGG.,TT.,,·,···· IKV.··…PK del L747-P753ins S ATCAACCAAT,·········,· ··CCAAA IK E........SK 注:核苷酸及氨基酸序列参照Genbank XO0538
2 致病基因序列分析 标题文本预设 点击此处更换文本点击此处更换文本点 击此处更换文本 标题文本预设 点击此处更换文本点击此处更换文本点 击此处更换文本 ⑥E746-A750insP所缺失9个碱基发生在碱基序列c.2236到c.2244,从而 导致由它们所编码的氨基酸(ELR)丢失。同时,碱基序列c.2248G突变 成C,密码子GCA被CCA取代,相应编码的丙氨酸(Ala)错义为脯氨酸 (Pro)

致病基因序列分析 O●●●● EGFR基因外显子20突变,即S7681是由碱基序列c.2303G突变为T,第768 位密码子AGC转变成ATC,由它所编码的丝氨酸(Ser)被异亮氨酸(|Ie) 替代。 EGFR基因外显子21突变是EGFR基因突变的主要类型之一,均由碱基点错义 突变引起,共检测出3种突变类型 a.L858R突变占绝大部分,共检测出5例。突变发生在碱基序列c.2573T 错义成G,而第858位的密码子CTG转变成CGG,所编码的亮氨酸(Leu) 被精氨酸(Arg)替代。 b.P848L突变发生在碱基序列c.2543C错义为T,相应的第848位密码子 CCG变为CTG,而由密码子所编码的脯氨酸(Pro)替代成亮氨酸 (Leu)。 c. L861Q突变是发生在碱基序列c.2582T错义为A,以致第861位密码子由 CTG转变为CAG,相应编码的亮氨酸(Leu)被谷氨酰胺(GIn)所替代
2 致病基因序列分析 标题文本预设 点击此处更换文本点击此处更换文本点 击此处更换文本 标题文本预设 点击此处更换文本点击此处更换文本点 击此处更换文本 EGFR基因外显子20突变,即S768I是由碱基序列 c.2303G突变为T,第768 位密码子AGC转变成ATC,由它所编码的丝氨酸(Ser)被异亮氨酸(Ile) 替代。 EGFR基因外显子21突变是EGFR基因突变的主要类型之一,均由碱基点错义 突变引起,共检测出3种突变类型 a. L858R突变占绝大部分,共检测出5例。突变发生在碱基序列c.2573T 错义成G,而第858位的密码子CTG转变成CGG,所编码的亮氨酸(Leu) 被精氨酸(Arg)替代。 b. P848L突变发生在碱基序列c.2543C错义为T,相应的第848位密码子 CCG变为CTG,而由密码子所编码的脯氨酸(Pro)替代成亮氨酸 (Leu)。 c. L861Q突变是发生在碱基序列c.2582T错义为A,以致第861位密码子由 CTG转变为CAG,相应编码的亮氨酸(Leu)被谷氨酰胺(Gln)所替代

3 蛋白质组成和结构分析 O●●●● EGFR基因负责编码并制造一种称为表皮生长因子受体的受体蛋白。 EGFR受体蛋白是一种跨膜蛋白,其分为三部分:蛋白的一端位于细胞外,一部 分位于细胞膜,另一端则位于细胞内。其结构由三部分组成:细胞外配体结合 域、跨膜区、细胞内酪氨酸激酶域。这允许EGFR受体与细胞外的其他蛋白(称 为配体)结合,帮助细胞接收信号并对其环境作出反应。而受体与配体的结合 如同钥匙与锁,也因此它们都有特定的结合“伙伴”。当EGFR与配体结合时, 它会附着于另一个位于附近的EGFR受体并形成复合物(二聚体),从而进入激 活状态,并激活细胞内的信号传导途径
3 蛋白质组成和结构分析 标题文本预设 点击此处更换文本点击此处更换文本点 击此处更换文本 标题文本预设 点击此处更换文本点击此处更换文本点 击此处更换文本 标题文本预设 点击此处更换文本点击此处更换文本点 击此处更换文本 EGFR基因负责编码并制造一种称为表皮生长因子受体的受体蛋白。 EGFR受体蛋白是一种跨膜蛋白,其分为三部分:蛋白的一端位于细胞外,一部 分位于细胞膜,另一端则位于细胞内。其结构由三部分组成:细胞外配体结合 域、跨膜区、细胞内酪氨酸激酶域。这允许EGFR受体与细胞外的其他蛋白(称 为配体)结合,帮助细胞接收信号并对其环境作出反应。而受体与配体的结合 如同钥匙与锁,也因此它们都有特定的结合“伙伴”。当EGFR与配体结合时, 它会附着于另一个位于附近的EGFR受体并形成复合物(二聚体),从而进入激 活状态,并激活细胞内的信号传导途径