
VMD教程 简介: 这个教程为新用户介绍了VMD的用法。老用户也可以用本教程进一步熟悉程序的应 用,以更好地利用VMD。本教程是针对VMD1.8.3设计的,需要约3个小时来完成。 本教程新增的内容可用三个独立的单元讲解。第一个单元主要内容是分子图形表现方法 基础,还会介绍制作形象逼真的图像要了解的知识。另外的两个单元是针对高级用户,介 绍了VMD的脚本。尽管非技术性用户可以略去脚本的阅读,但是我们鼓励每个人都去试一 试着读一下,因为它会提供一些有力而易用的工具,这些工具是简单的图形用户界面所无法 提供的。 本教程以一种有趣的小蛋白质泛素的研究为例来说明VM①的应用。在本文中,一些资 料是在小框中出现的。这些小框中包括教程的补充内容,例如泛素扮演的生物学角色,使用 VMD的一些提示和捷径等等。 如果你有对本教程的评论和问题,请发邮件至tutorial-1@ks.uiuc.edu。邮件列表可以在 http:lwww.ks.uiuc.edu/Training/Tutorials/mailing list/tutorial-l/.中找到。 泛素本教程会用VMD来显示泛素。泛素是一个由76个氨基酸组成 的小蛋白质,在所有的真核生物中普遍存在。在所有真核生物蛋白 质中,泛素是最为保守的蛋白质之一(在昆虫,鱼,牛和人中,前 74个氨基酸是完全一样的)。它己被证明存在于细胞核、细胞质和 细胞表面。它首要的功能是介导蛋白质降解,在降解过程中,作为 细胞内蛋白水解酶识别的标志。 需要的程序: 以下是本教程中需要的程序 VMD:可以从htp:wwks.uiuc.edu/Research/vmd过下载(在所有平台上均可使用)。 绘图程序:要观看从VMD输出的图像,需要专门的程序。VMD有一个内置的绘图程序, 也可以应用外部程序。应用什么程序是由你的操作系统决定的。例如: -Unix/Linux:xmgrace,http://plasma-gate.weizmann.ac.il/Grace/ -Windows:Excel,http://office.microsoft.com/en-us/FX010858001033.aspx (需要购买) -Mac/Multiple Platforms:Mathematica,http://www.wolfram.com/ (需要购买),gnuplot,http://www.gnuplot info/(免费下载) 现在开始学习VMD 你可以在VMD-tutorial-files目录里找到本教程的文件。如图1所示的VMD-tutorial-files的 文件和目录
VMD 教程 简介: 这个教程为新用户介绍了 VMD 的用法。老用户也可以用本教程进一步熟悉程序的应 用,以更好地利用 VMD。本教程是针对 VMD 1.8.3 设计的,需要约 3 个小时来完成。 本教程新增的内容可用三个独立的单元讲解。第一个单元主要内容是分子图形表现方法 基础, 还会介绍制作形象逼真的图像要了解的知识。另外的两个单元是针对高级用户,介 绍了 VMD 的脚本。尽管非技术性用户可以略去脚本的阅读,但是我们鼓励每个人都去试一 试着读一下,因为它会提供一些有力而易用的工具,这些工具是简单的图形用户界面所无法 提供的。 本教程以一种有趣的小蛋白质泛素的研究为例来说明 VMD 的应用。在本文中,一些资 料是在小框中出现的。这些小框中包括教程的补充内容,例如泛素扮演的生物学角色,使用 VMD 的一些提示和捷径等等。 如果你有对本教程的评论和问题,请发邮件至tutorial-l@ks.uiuc.edu。邮件列表可以在 http://www.ks.uiuc.edu/Training/Tutorials/mailing list/tutorial-l/.中找到。 泛素 本教程会用VMD来显示泛素。泛素是一个由76个氨基酸组成 的小蛋白质,在所有的真核生物中普遍存在。在所有真核生物蛋白 质中,泛素是最为保守的蛋白质之一(在昆虫,鱼,牛和人中,前 74个氨基酸是完全一样的)。它已被证明存在于细胞核、细胞质和 细胞表面。它首要的功能是介导蛋白质降解,在降解过程中,作为 细胞内蛋白水解酶识别的标志。 需要的程序: 以下是本教程中需要的程序 VMD: 可以从 http://www.ks.uiuc.edu/Research/vmd/下载(在所有平台上均可使用)。 绘图程序:要观看从 VMD 输出的图像,需要专门的程序。VMD 有一个内置的绘图程序, 也可以应用外部程序。应用什么程序是由你的操作系统决定的。例如: –Unix/Linux: xmgrace, http://plasma-gate.weizmann.ac.il/Grace/ –Windows: Excel, http://office.microsoft.com/en-us/FX010858001033.aspx (需要购买) –Mac/Multiple Platforms: Mathematica, http://www.wolfram.com/ (需要购买); gnuplot, http://www.gnuplot.info/(免费下载) 现在开始学习 VMD 你可以在 VMD-tutorial-files 目录里找到本教程的文件。如图 1 所示的 VMD-tutorial-files 的 文件和目录

1UBQ.pdb coloring.tcl equilibration.dcd example-output mmsd.dat pulling.dcd vmd-tutorial-files rmsd-fullthrottle.tcl rmsd dat rmsd.tcl ubiquitin-equilibrated coor ubiquitin psf 图l:VMD-tutorial-files的目录结构 运行VMD,可以在Unix终端窗口中键入vmd,在Mac OS X的应用文件夹中双击VMD应用 程序图标或者在windows中单击开始一程序一VMD。 1VMD基础 在本单元中你会通过构建一个泛素的较美观的图形来熟悉VMD的基本命令。另外,你 可以学习怎样用VMD来寻找蛋白质结构上的有趣的特点。 1.1导入分子 600 D 0 Fle Molecule Graphics Display Mouse Extensions Help 第一个步骤是导入分子。在教程中提 New Molecule... 供了一个pdb文件IUBQ.pdb,文件中包含 Atoms Frames 了泛素的原子坐标。 Load State. Save State. Render.. I在VMD主窗口的菜单栏中选择File一 I Qult K灯200m◆Lcop口 step 1 speed I H> New Molecule,.如图2(a),屏幕上会显 000 Holecule Fils r 示另外一个窗口,即Molecule File Browser Load files for:New Molecule (b)。 Filename: Determine file type 2 应用Browse.(c)按钮在 Automatically三 Load vmd-tutorial-files中找到文件 /olumetric Data的ets IUBQ.pdb,注意到当你选择这个文 件的时候,就会回到Molecule File oLoad in backaroun oLoad all at ance Browser窗口。为了精确地导入你要 导入的文件一定不要忘了按下Load(d)按钮。 图2导入分子 现在,泛素在你的OpenGL Displayi窗口中显示出来。你可以随时选择Molecule File Browser 窗口
图 1:VMD-tutorial-files 的目录结构 运行VMD, 可以在Unix 终端窗口中键入vmd, 在Mac OS X的应用文件夹中双击VMD应用 程序图标或者在windows中单击开始——程序——VMD。 1 VMD基础 在本单元中你会通过构建一个泛素的较美观的图形来熟悉VMD的基本命令。另外,你 可以学习怎样用VMD来寻找蛋白质结构上的有趣的特点。 1.1 导入分子 第一个步骤是导入分子。在教程中提 供了一个pdb文件1UBQ.pdb,文件中包含 了泛素的原子坐标。 1在VMD主窗口的菜单栏中选择File —— New Molecule,如图2(a),屏幕上会显 示另外一个窗口,即Molecule File Browser (b)。 2 应用Browse.(c)按钮在 vmd-tutorial-files中找到文件 1UBQ.pdb,注意到当你选择这个文 件的时候,就会回到Molecule File Browser窗口。为了精确地导入你要 导入的文件一定不要忘了按下Load(d)按钮。 现在,泛素在你的OpenGL Display窗口中显示出来。你可以随时选择Molecule File Browser 窗口。 图 2 导入分子

Webpdb.如果网络连接可用,VMD可以从蛋白质数据库中下载pdb文件。只 要在Molecule File Browse窗口的File Name中键入四个字母的蛋白质号,再点 一下load就可以了。VMD会自动下载该pdb文件: 坐标文件。文件1UBQ.pdb与泛素的X射线衍射1.8埃分辨率侧的结果 香对应(Senadhi Vijay-.Kumar,,Charles E.Bugg and William J.Cook,J. Mol.Biol (1987)194,531)。注意蛋白质被58个水分子包围,结果中不包含氢 原子。 1.2显示蛋白质 为了观察蛋白质的三维结构, 我们要用到多种鼠标模式。 1在OpenGL Display中,按下 鼠标左键同时移动鼠标。进一 步观察有什么现象。这是鼠标 的旋转模式,通过这种模式, 你可以让这个分子绕一个与 (a) (b) 屏幕平行的轴旋转。图3(a) 图3旋转模式 2如果你按下鼠标右键,重复上一步骤,分子会绕一个与屏幕垂直的轴旋转(b)(对于Mac 用户来说,右键产生的效果与在按下nouse?菜单选项后点击命令按钮是一样的)。 3在VMD主窗口中,看一下Mouse菜单 000 WD O (图4),这里,你可以把鼠标模式从 File Molecule Graphics Display Mouse Extensions Help Rotation更换到Translation或者Scale ID TA D F Molecule Rotate Mode r Translate Mode modes。 Scale Mode Center 4 Translation模式允许你按住鼠标左 个Quey 0 键,在屏幕上移动分子。在Translation Label 模式下,你可以通过按下鼠标中键来改 ■Move √zo0m◆ step ■Force PID 变剪切板。 Move Light ◇Add/Remove Bonds 5 在Scale模式下,你可以按住左键水 平移动鼠标来缩小或放大分子。 图4鼠标模式 需要注意的是:鼠标运动不会改变分子中的原子坐标
Webpdb. 如果网络连接可用,VMD可以从蛋白质数据库中下载pdb文件。只 要在Molecule File Browse窗口的File Name中键入四个字母的蛋白质号,再点 一下load就可以了。VMD会自动下载该pdb文件。 坐标文件。文件1UBQ.pdb与泛素的X射线衍射1.8埃分辨率侧的结果 香对应(Senadhi Vijay-Kumar, Charles E. Bugg and William J. Cook, J. Mol. Biol. (1987) 194, 531)。注意蛋白质被58个水分子包围,结果中不包含氢 原子。 1.2 显示蛋白质 为了观察蛋白质的三维结构, 我们要用到多种鼠标模式。 1 在OpenGL Display中,按下 鼠标左键同时移动鼠标。进一 步观察有什么现象。这是鼠标 的旋转模式,通过这种模式, 你可以让这个分子绕一个与 屏幕平行的轴旋转。图3(a) 2 如果你按下鼠标右键,重复上一步骤,分子会绕一个与屏幕垂直的轴旋转(b)(对于Mac 用户来说,右键产生的效果与在按下mouse菜单选项后点击命令按钮是一样的)。 3 在VMD主窗口中,看一下Mouse菜单 (图4),这里,你可以把鼠标模式从 Rotation 更换到Translation或者Scale modes。 4 Translation模式允许你按住鼠标左 键,在屏幕上移动分子。在Translation 模式下,你可以通过按下鼠标中键来改 变剪切板。 5 在Scale模式下,你可以按住左键水 平移动鼠标来缩小或放大分子。 需要注意的是:鼠标运动不会改变分子中的原子坐标。 图 3 旋转模式 图 4 鼠标模式

鼠标模式。注意每一种鼠标模式都有独特的指针形状,也有独特的快捷键( Rotate,.t:Translate,s:Scale),可以代替菜单使用。(当用快捷键的时候,要 保证OpenGL Display窗口是活动的)。在VMD用户手册中可以获得更多的 信息。 Mouse Center菜单项也很有用,它允许你确定分子绕之旋转的支点。 6 选择Center菜单项,在蛋白质一端选择一个原子,这时指针会显示成一个十字。 7现在,按下工,用鼠标旋转分子,看一看你的分子是怎么绕着你选择的支点运动的。 8选择Display一Reset View菜单项(=快捷键),回到默认界面。 1.3学习应用不同的绘图模式 VMD可以用很多种绘图模式来显示你的分子。这里,我们要进一步学习那些可以帮助你确 定蛋白质中不同结构的绘图模式。 000 Graphical Representations Selec.ed Molecule l选择Graphics-一 Representations 0:1UBQ 菜单项,一个叫做Graphical Create Rep Delete Rep Representations的窗口会出现,见图5 Style Coor Selection (a)中黄色高亮。你可以看到目前显 a) Lines Name a 示的分子的图形显示法。 2在Draw Style标签中(b)我们可 以改变所表示的style(d)和color Selected Atoms (C)。在这一部分我们重点来看 a叫 -① drawing模式。 b)- Draw style Selections\TrajectoryPeriodic Coloring Method Material (c)_ Name Opaque 3每一种绘图方法都有自己的参数 Drawing Method 控制。例如,改变线条的稠密度可 Lines Default 以用Graphical Representations窗 口右侧底部的控制按钮(e)。 4现在,在Drawing Method中选择 Thickness 1 VDW(van der Waals),每一个原子 现在都表示为球形。用这种方式你 →m8Apy 可以更容易地看出蛋白质的体积 分布是怎样的。 图5 Graphical Representations窗▣ 5要观察蛋白质内部的原子排布,用窗口右侧底部的控制按钮改变Sphere Scale到0.5
鼠标模式。注意每一种鼠标模式都有独特的指针形状,也有独特的快捷键((r: Rotate, t: Translate, s:Scale),可以代替菜单使用。(当用快捷键的时候,要 保证OpenGL Display窗口是活动的)。在VMD用户手册中可以获得更多的 信息。 Mouse ——Center菜单项也很有用,它允许你确定分子绕之旋转的支点。 6 选择Center菜单项,在蛋白质一端选择一个原子,这时指针会显示成一个十字。 7 现在,按下r, 用鼠标旋转分子,看一看你的分子是怎么绕着你选择的支点运动的。 8 选择Display ——Reset View菜单项(=快捷键),回到默认界面。 1.3 学习应用不同的绘图模式 VMD可以用很多种绘图模式来显示你的分子。这里,我们要进一步学习那些可以帮助你确 定蛋白质中不同结构的绘图模式。 1 选择Graphics—— Representations 菜单项,一个叫做Graphical Representations的窗口会出现,见图5 (a)中黄色高亮。你可以看到目前显 示的分子的图形显示法。 2 在Draw Style标签中(b)我们可 以改变所表示的style (d) 和color (c)。在这一部分我们重点来看 drawing模式。 3 每一种绘图方法都有自己的参数 控制。例如,改变线条的稠密度可 以用Graphical Representations窗 口右侧底部的控制按钮(e)。 4 现在,在Drawing Method中选择 VDW(van der Waals),每一个原子 现在都表示为球形。用这种方式你 可以更容易地看出蛋白质的体积 分布是怎样的。 5 要观察蛋白质内部的原子排布,用窗口右侧底部的控制按钮改变Sphere Scale到 0.5, 图5 Graphical Representations窗口

Sphere Resolution到l3。注意分辨率越高,分子的显示速度越慢。 6注意Coloring Method一Name菜单项,每一个原子都有其自己的颜色,比如,O是红 色的,N是蓝色的,C是青色的,S是黄色的。 7按下Default键,这个操作允许你回到默认的绘图方式中。 更多显示方法。还有有趣的显示方法是CPK和Licorice。在CPK中, 就像以前化学中的球棒模型,每个原子都用球形表示,每个键都用圆柱 棒表示(球和圆柱形棒的半径和分辨率都可以独立地调节)。Licorice绘 图方法(广泛使用)也用球形表示原子,用圆柱形棒表示键,但是球的 半径不能被独立调节。 前面的显示方式可以让你看到蛋白质大分子的细节。但是,更多的普遍结构属性可以用抽象 的绘图方式来观看。 8在Drawing Method下选择Tube style,观察蛋白骨架。Radiusi设为0.8。 9在tube模式下观察你的蛋白质,你可以分辨出它有多少a螺旋、折叠和无规则卷曲吗? 我们要了解的最后一个绘图模式是NewCartoon。它可以给出一个以二级结构为基础的简化 的蛋白质图像。α螺旋以卷曲的条带状表示,B折叠以固形箭头表示,所有其它的结构以管状 表示。这可能是观察蛋白质分子总体构造的最普遍的方法。 l0选择Drawing Method—NewCartoon 11现在确定蛋白质分子中有多少α螺旋、折叠和无规则卷曲。 泛素的结构。泛素有一个三圈半的c螺旋(残基23到34,其中有三 个是疏水的),一个310-螺旋(残基56到59),还有5个折叠(残基 1到7,10到17,40到45,48到50,64到72),还有7个反向转角。 VMD用STRIDE程序,以一种探索性的法则计算出二级结构
Sphere Resolution 到13。注意分辨率越高,分子的显示速度越慢。 6 注意Coloring Method —— Name菜单项, 每一个原子都有其自己的颜色,比如,O是红 色的,N是蓝色的,C是青色的,S是黄色的。 7 按下Default键,这个操作允许你回到默认的绘图方式中。 . 前面的显示方式可以让你看到蛋白质大分子的细节。但是,更多的普遍结构属性可以用抽象 的绘图方式来观看。 8 在Drawing Method下选择Tube style,观察蛋白骨架。Radius设为0.8。 9 在tube模式下观察你的蛋白质,你可以分辨出它有多少α螺旋、β折叠和无规则卷曲吗? 我们要了解的最后一个绘图模式是NewCartoon。它可以给出一个以二级结构为基础的简化 的蛋白质图像。α螺旋以卷曲的条带状表示,β折叠以固形箭头表示,所有其它的结构以管状 表示。这可能是观察蛋白质分子总体构造的最普遍的方法。 , 10 选择Drawing Method ——NewCartoon. 11 现在确定蛋白质分子中有多少α螺旋、β折叠和无规则卷曲。 泛素的结构。泛素有一个三圈半的α螺旋(残基23到34,其中有三 个是疏水的),一个310-螺旋(残基56到59),还有5个β折叠(残基 1到7,10到17,40到45,48到50,64到72),还有7个反向转角。 VMD用STRIDE程序,以一种探索性的法则计算出二级结构 更多显示方法。还有有趣的显示方法是 CPK 和 Licorice。在 CPK 中, 就像以前化学中的球棒模型,每个原子都用球形表示,每个键都用圆柱 棒表示(球和圆柱形棒的半径和分辨率都可以独立地调节)。Licorice 绘 图方法(广泛使用)也用球形表示原子,用圆柱形棒表示键,但是球的 半径不能被独立调节
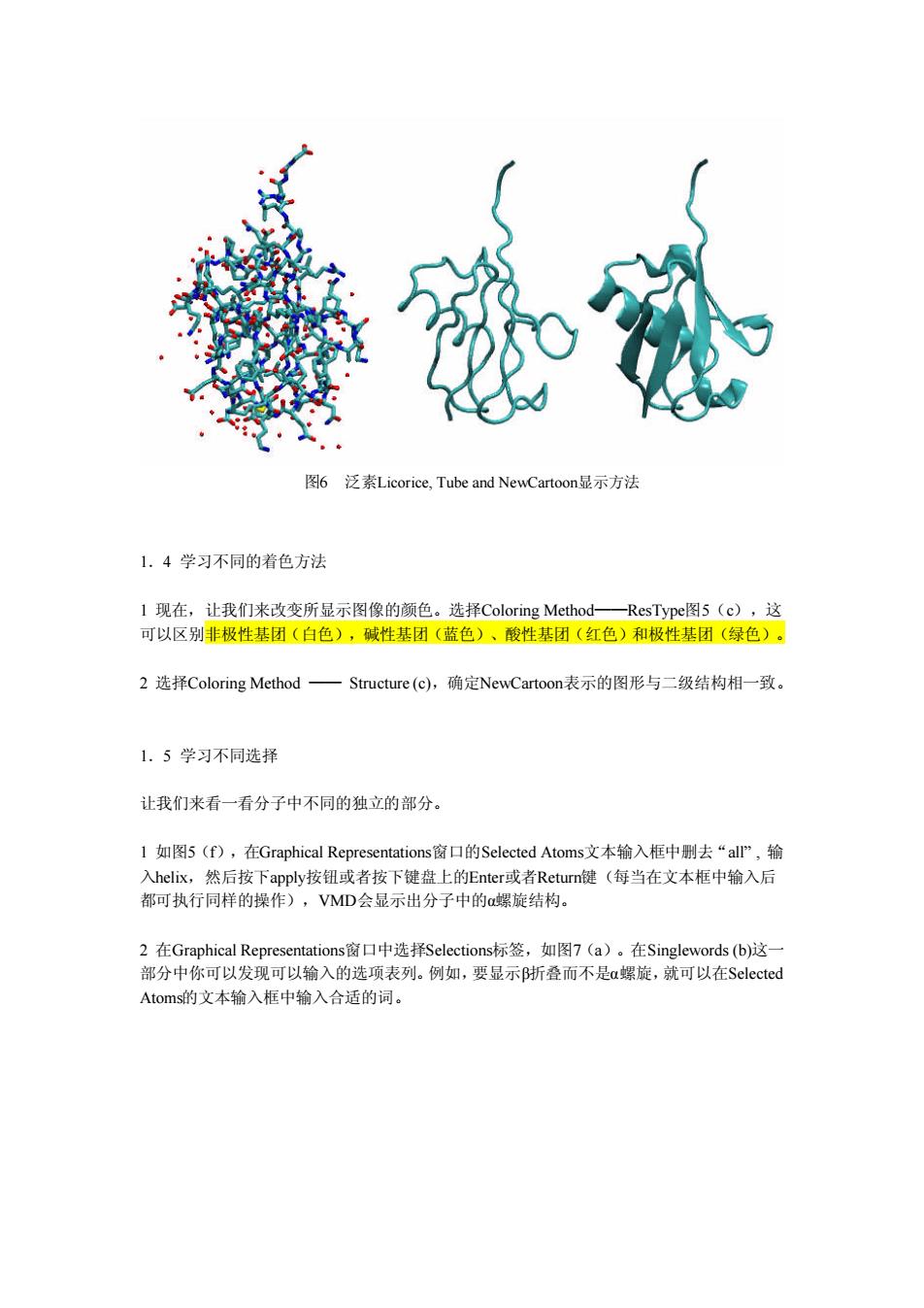
图6泛素Licorice,Tube and NewCartoon.显示方法 1.4学习不同的着色方法 1现在,让我们来改变所显示图像的颜色。选择Coloring Method-一一ResTypel图5(c),这 可以区别非极性基团(白色),碱性基团(蓝色)、酸性基团(红色)和极性基团(绿色)。 2选择Coloring Method一Structure(c),确定NewCartoon表示的图形与二级结构相一致。 1.5学习不同选择 让我们来看一看分子中不同的独立的部分。 1如图5(f),在Graphical Representations窗口的Selected Atoms文本输入框中删去“al”,输 入heliⅸ,然后按下apply按钮或者按下键盘上的Enter或者Return键(每当在文本框中输入后 都可执行同样的操作),VMD会显示出分子中的α螺旋结构。 2在Graphical Representations窗口中选择Selections标签,如图7(a)。在Singlewords(b)这一 部分中你可以发现可以输入的选项表列。例如,要显示折叠而不是a螺旋,就可以在Selected Atoms的文本输入框中输入合适的词
1.4 学习不同的着色方法 1 现在,让我们来改变所显示图像的颜色。选择Coloring Method——ResType图5(c),这 可以区别非极性基团(白色),碱性基团(蓝色)、酸性基团(红色)和极性基团(绿色)。 2 选择Coloring Method —— Structure (c),确定NewCartoon表示的图形与二级结构相一致。 1.5 学习不同选择 让我们来看一看分子中不同的独立的部分。 1 如图5(f),在Graphical Representations窗口的Selected Atoms文本输入框中删去“all”, 输 入helix,然后按下apply按钮或者按下键盘上的Enter或者Return键(每当在文本框中输入后 都可执行同样的操作),VMD会显示出分子中的α螺旋结构。 2 在Graphical Representations窗口中选择Selections标签,如图7(a)。在Singlewords (b)这一 部分中你可以发现可以输入的选项表列。例如,要显示β折叠而不是α螺旋,就可以在Selected Atoms的文本输入框中输入合适的词。 图6 泛素Licorice, Tube and NewCartoon显示方法

000 Graphical Representations 布尔操作组合也可以用于选择时的文本 Selected Molecule 输入。 0:1UBG 3为了看分子除了α螺旋和β折叠的部 Create Rep Delete Rep 分,可以在Selected Atoms中输入:(not Style Color Selection Cartoon Structure helix helix)and(not betasheet) 4 Selections标签(b)的Keyword(c)栏 可根据蛋白质某些部分的特定值来进行 选择。看一看Keyword resname(d)中的 Selected Atoms 可能值。输入(resname LYS)或者 helixd (resname GLY)可以显示蛋白质中所有 Draw stye Selections Trajectory Periodic 的赖氨酸或者甘氨酸。赖氨酸在泛素的 b) Singlewords 构型中扮演重要的角色。 a and or not none backbone Apply sidechain 5现在,把当前显示的Drawing Method protein Reset 改变到CPK模式,把Draw Style中的 Macro definition: Coloring Methodi改到ResID。在屏幕中可 以看到不同的赖氨酸和甘氨酸。每一种 c Keyword Value d 有多少个你能数得清吗? name type index 6在Selected Atoms的文本输入框中输 residue resname 入water。.选择Coloring Method一 resid Name。你可以看到在整个系统中的58 图7 Graphical Representations窗口和Selection标签 个水分子(实际上只有氧被显示出来)。 7为了看一看哪些水分子离蛋白质分子更近一些,可以用within命令。输入waterwithin 3 of protein,这就选择了距离蛋白质3埃之内的所有的水分子。 8最后,在Selected Atoms中键入下列内容: Selection Action protein Shows the Protein resid 1 The first residues (resid 1 76)and (not water) The first and last residues (resid 23 to 34)and (protein) The_helix 前述的选项提供了研究蛋白质或其他分子的有力工具。 1.6多重显示
布尔操作组合也可以用于选择时的文本 输入。 3 为了看分子除了α螺旋和β折叠的部 分,可以在Selected Atoms中输入:(not helix)and(not betasheet) : 4 Selections标签 (b)的Keyword (c)栏 可根据蛋白质某些部分的特定值来进行 选择。看一看Keyword resname (d)中的 可能值。输入(resname LYS)或者 (resname GLY)可以显示蛋白质中所有 的赖氨酸或者甘氨酸。赖氨酸在泛素的 构型中扮演重要的角色。 . 5 现在,把当前显示的Drawing Method 改变到CPK模式,把Draw Style中的 Coloring Method改到ResID。在屏幕中可 以看到不同的赖氨酸和甘氨酸。每一种 有多少个你能数得清吗? 6 在Selected Atoms的文本输入框中输 入water。选择Coloring Method —— Name。你可以看到在整个系统中的58 个水分子(实际上只有氧被显示出来)。 7 为了看一看哪些水分子离蛋白质分子更近一些,可以用within命令。输入waterwithin 3 of protein,这就选择了距离蛋白质3埃之内的所有的水分子。 8 最后,在Selected Atoms中键入下列内容: : Selection Action protein resid 1 (resid 1 76) and (not water) (resid 23 to 34) and (protein) Shows the Protein The first residues The first and last residues The _ helix 前述的选项提供了研究蛋白质或其他分子的有力工具。 1.6 多重显示 图7 Graphical Representations窗口和Selection标签

如图8(a),在Graphical Representations OGraphical Representations 窗口中,用Create Rep按钮可以创建多重 Selected Molecule 显示图像。因此,你可以让分子的不同 0:1BQ 部分显示不同的样式和颜色。 a) Create Rep Delete Rep Style Color Selection NewCartoon Structure protein 1对当前显示,把Drawing Method设为 VDW ResType resname LYS CPK Name water NewCartoon,把Coloring Method设为 VDW ColorlD 1 resid 1 76 and d) Surf Molecule protein Structure. Selected Atoms proteinl 2在Selected Atoms中键入protein. Draw style Selections Trajectory Periodic Coloring Method Material 一c) 3按下Create Rep键(a),现在,用 Molecule Transparent Draw Style菜单项和Selected Atoms.文本 Drawing Method Surf Default 输入框来更改新的图形显示,可以把 Drawing Method设为VDW,Coloring Methodi设为ResType,并键入resname Representation Method Solid Surface LYS,使其成为当前选择。 Probe Radius 1.4 ◆Apply Changes Apply】 △utomatically 4重复前述步骤,产生下列两种新的显 示方法:: 图8泛素的多重显示方法 Drawing Style Coloring Method Selection CPK Name Water VDW ColorIDI Resid 1 76 and name CA 5再次按下Create Rep按钮,创建最后一个图形显示法。选择Drawing Method一Surf Coloring Method一Molecule,在Selected Atoms中键入protein。在Material部分(c)中选择 Transparent菜单项。 6用鼠标你可以选择己创建的不同的显示法,并可以独立地改变其中的任何一种。你也可以 用双击鼠标或者Delete Rep按钮打开或关闭它们。关闭第二个和最后一个图形表示法。在这 一部分的最后,Graphical Representations窗口的显示如图8
如图8(a),在Graphical Representations 窗口中,用Create Rep按钮可以创建多重 显示图像。因此,你可以让分子的不同 部分显示不同的样式和颜色。 1 对当前显示,把Drawing Method 设为 NewCartoon,把Coloring Method 设为 Structure. 2 在 Selected Atoms 中键入 protein. 3 按下Create Rep键(a),现在,用 Draw Style菜单项和Selected Atoms文本 输入框来更改新的图形显示,可以把 Drawing Method设为VDW,Coloring Method设为ResType,并键入resname LYS,使其成为当前选择。 4 重复前述步骤,产生下列两种新的显 示方法:: Drawing Style Coloring Method Selection CPK VDW Name ColorID1 Water Resid 1 76 and name CA , 5 再次按下Create Rep按钮,创建最后一个图形显示法。选择Drawing Method —— Surf, Coloring Method —— Molecule,在Selected Atoms中键入protein。在Material 部分(c)中选择 Transparent菜单项。 6 用鼠标你可以选择已创建的不同的显示法,并可以独立地改变其中的任何一种。你也可以 用双击鼠标或者Delete Rep按钮打开或关闭它们。关闭第二个和最后一个图形表示法。在这 一部分的最后,Graphical Representations窗口的显示如图8 , 图 8 泛素的多重显示方法

1.7 Sequence Viewer Extension (a) 当第一次处理一个蛋白质分子的 时候,快速找出和显示不同的氨基 000 VMD Sequence 1UBO (mol 0) 0 酸是非常有用的。Sequence File Help 23 ILE Viewer Extension可以让你很容易 24 GLU 25 ASN K 地选出和显示氨基酸残基。 26 VAL X 27 LYS b Close Window 28 ALA 29 LYS X 1选择Extensions一Analysis- 30 ILE X 31 GLN 一Sequence Viewer菜单项,一个 ASP (e) 33 LYS x 包含氨基酸(图9e)和它们属性的 34 GLU X 列表(b)和(c)的窗口(图9a) Molecule:0 6 ILE 37 PRO X 会出现在屏幕上。 38PR0 Z00m 39 ASP 0.01 41 OLN 2用鼠标点击列表中不同的氨基 42 ARG X 0.51 43 LEU X 酸残基(ε),观察它们是如何被 44 ILE 1.011.00 45PH匝X 标记为高亮的。另外,高亮的残基 46 ALA X 1.51 47 GLY X 还会在DpenGL Display窗口中以 48Ls8 2.01 49 OLN X 黄色bond drawing方式显示,因此 50 LEU X fit all 51 GLU X 你可以很容易地观察它们。用鼠标 ASP every residue 53 GLY 右键可以解除选择。 54 ARG X LEU 1-letter code SER 3用Zoom滑块控制窗口(f),使 58 ASP 其能够显示所有残基。这在大的蛋 60 61 ILE 白质分子中比较有用。 62 GLN X 63LYs室 B value 4按住shif键时同时按下鼠标,就 150 可以同时选择多个残基。看一下图 中显示的残基48.63,11 和29(e)。 图9 sequence窗口 5观察Graphical Representations窗口,用SequenceViewer Extension,你应该能发现一种新的 显示所选残基的方法。就像你以前做过的那样,你可以修改、隐藏、或者删除这种显示方法。 赖氨酸的相关性。多个泛素链可以被C和N末端肽键连接,也可以通 过ys48,63,11或者29连接(就是你在sequence窗口中选择的)不 同连接形成的链有着与功能相关的不同的属性。 关于残基的信息用柱状彩色标记表示,它们是从STRIDE中得到的。B-value表示的是温度因
1.7 Sequence Viewer Extension 当第一次处理一个蛋白质分子的 时候,快速找出和显示不同的氨基 酸是非常有用的。Sequence Viewer Extension可以让你很容易 地选出和显示氨基酸残基。 1 选择Extensions ——Analysis— — Sequence Viewer菜单项,一个 包含氨基酸(图9e)和它们属性的 列表(b)和(c)的窗口(图9 a) 会出现在屏幕上。 2 用鼠标点击列表中不同的氨基 酸残基(e),观察它们是如何被 标记为高亮的。另外,高亮的残基 还会在OpenGL Display窗口中以 黄色bond drawing方式显示,因此 你可以很容易地观察它们。用鼠标 右键可以解除选择。 3 用Zoom滑块控制窗口(f),使 其能够显示所有残基。这在大的蛋 白质分子中比较有用。 4 按住shift键时同时按下鼠标,就 可以同时选择多个残基。看一下图 中显示的残基48, 63, 11 和29 (e)。. 5 观察Graphical Representations窗口,用SequenceViewer Extension,你应该能发现一种新的 显示所选残基的方法。就像你以前做过的那样,你可以修改、隐藏、或者删除这种显示方法。 赖氨酸的相关性。多个泛素链可以被C和N末端肽键连接,也可以通 过lys48,63,11或者29连接(就是你在sequence窗口中选择的)不 同连接形成的链有着与功能相关的不同的属性。 关于残基的信息用柱状彩色标记表示,它们是从STRIDE中得到的。B-value表示的是温度因 图 9 sequence 窗口

子的变化,struct表示二级结构,在图中,各种颜色所代表的意义都用字母作了标注。 T Turn E Extended conformation sheets) B Isolated bridge Alpha helix G 3-10 helix Pi helix Coil 1.8保存结果 用VMD创建的图形可以与创建的图形显示法,VMD环境设置一起保存。这里提到的VMD 环境设置包括你开启一个新的VMD环境所需要的所有信息,这就不会使你以前的工作成果 丢失。 1在VMD主窗口,选择File一Save State菜单项,写上一个合适的文件名(例如: myfirststate.vmd)保存。 File一Load State菜单项允许你导入一个保存过VMD环境设置,就像保存时一样,虽然 设置好的VMD环境允许你用VMD来处理蛋白质的图像,探索它的性质,但是你通常需要得 到能用于论文和其他各种文件的图像。VMD可以润色图像,产生一个可作它用的图像文件。 如下所述: 2用你己经学过的所有关于VMD的知识,用缩放、旋转和改变分子位置的方法找到蛋白质 分子的合适的视野。打开和关闭不同的显示方法,提高选择的分辨率,调整其他属性。如果 你想得到一个质量很高的分子,注意每一个显示方法的分辨率。 3注意你用Sequence Viewer extension创建的新显示法。如果需要的话,隐藏或者删除它们。 4在润色图像之前,选择Graphics一Colors菜单项,改变背景颜色。选择Display category, Background name和8 white color..。这样背景就变成白色的了。 5选择File一Render菜单项(渲染),一个叫做File Render Controls的窗口会在屏幕上出现。 6可以用不同的文件包来润色分子。如果你用的操作系统是Unix或者Mac OS X,就在Render using菜单中选择TachyonInternal,否则选择Tachyon。 7在Filename文本输入框中键入图像要保存的文件名,例如:picture.tga或者picture.dat(默
子的变化,struct表示二级结构,在图中,各种颜色所代表的意义都用字母作了标注。 . T E B H G I C Turn Extended conformation (_ sheets) Isolated bridge Alpha helix 3-10 helix Pi helix Coil 1.8 保存结果 用VMD创建的图形可以与创建的图形显示法,VMD环境设置一起保存。这里提到的VMD 环境设置包括你开启一个新的VMD环境所需要的所有信息,这就不会使你以前的工作成果 丢失。 1 在VMD主窗口,选择File ——Save State菜单项,写上一个合适的文件名(例如: myfirststate.vmd)保存。 File —— Load State菜单项允许你导入一个保存过VMD环境设置,就像保存时一样,虽然 设置好的VMD环境允许你用VMD来处理蛋白质的图像,探索它的性质,但是你通常需要得 到能用于论文和其他各种文件的图像。VMD可以润色图像,产生一个可作它用的图像文件。 如下所述: 2 用你已经学过的所有关于VMD的知识,用缩放、旋转和改变分子位置的方法找到蛋白质 分子的合适的视野。打开和关闭不同的显示方法,提高选择的分辨率,调整其他属性。如果 你想得到一个质量很高的分子,注意每一个显示方法的分辨率。 3 注意你用Sequence Viewer extension创建的新显示法。如果需要的话,隐藏或者删除它们。 4 在润色图像之前,选择Graphics —— Colors菜单项,改变背景颜色。选择Display category, Background name和 8 white color.。这样背景就变成白色的了。 5 选择File—— Render菜单项(渲染),一个叫做File Render Controls的窗口会在屏幕上出现。 6 可以用不同的文件包来润色分子。如果你用的操作系统是Unix或者Mac OS X,就在Render using菜单中选择TachyonInternal,否则选择Tachyon。 7 在Filename文本输入框中键入图像要保存的文件名,例如:picture.tga 或者picture.dat (默