
宿主通过粪便中的microRNA塑造肠道菌群 一、导读 宿主肠道拥有复杂的微生物群,其初始源自母代(体),并通 过摄食及与其他环境因素的接触而继续发展。人类幼儿在大约3岁 时,其肠道菌群会渐趋于成人的菌群组成特点,变得相当稳定。许 多因素都会影响肠道菌群,诸如宿主的遗传,饮食及病理状态等。 同一物种其菌群组成趋于相同,但不同物种间菌群大相径庭。在交 互肠道菌群移植实验中,移植后的无菌斑马鱼及无菌小鼠的肠道菌 群与其同类更为相似,这表明宿主拥有让特定菌群选择性定殖的机 制。因为肠道菌群在宿主的代谢、免疫及疾病中都扮演重要角色, 故阐明宿主调控菌群的机制与方式显得尤为重要。 miRNA是一类小的非编码RNA,大约长度为18-23nt,在细胞 核中合成,细胞质中发挥功能。然而,越来越多的证据表明miRNA 也出现在胞外并随体液而流动散播。先前的研究已经发现粪便中的 miRNA可以作为肠道肿瘤的分子标志物。但正常的肠道中是否拥有 miRNA还未探明。本文在肠内容物及粪便中发现miRNA,并且阐 明其在调控宿主菌群的角色。 二、论文 原名:The Host Shapes the Gut Microbiota via Fecal MicroRNA 译名:宿主通过粪便中的microRNA塑造肠道菌群 期T刊:Cell Host&Microbe IF:14.946 发表时间:2016年1月
宿主通过粪便中的 microRNA 塑造肠道菌群 一、导读 宿主肠道拥有复杂的微生物群,其初始源自母代(体),并通 过摄食及与其他环境因素的接触而继续发展。人类幼儿在大约 3 岁 时,其肠道菌群会渐趋于成人的菌群组成特点,变得相当稳定。许 多因素都会影响肠道菌群,诸如宿主的遗传,饮食及病理状态等。 同一物种其菌群组成趋于相同,但不同物种间菌群大相径庭。在交 互肠道菌群移植实验中,移植后的无菌斑马鱼及无菌小鼠的肠道菌 群与其同类更为相似,这表明宿主拥有让特定菌群选择性定殖的机 制。因为肠道菌群在宿主的代谢、免疫及疾病中都扮演重要角色, 故阐明宿主调控菌群的机制与方式显得尤为重要。 miRNA 是一类小的非编码 RNA,大约长度为 18-23nt,在细胞 核中合成,细胞质中发挥功能。然而,越来越多的证据表明 miRNA 也出现在胞外并随体液而流动散播。先前的研究已经发现粪便中的 miRNA 可以作为肠道肿瘤的分子标志物。但正常的肠道中是否拥有 miRNA 还未探明。本文在肠内容物及粪便中发现 miRNA,并且阐 明其在调控宿主菌群的角色。 二、论文 原名:The Host Shapes the Gut Microbiota via Fecal MicroRNA 译名:宿主通过粪便中的 microRNA 塑造肠道菌群 期刊:Cell Host&Microbe IF:14.946 发表时间:2016 年 1 月
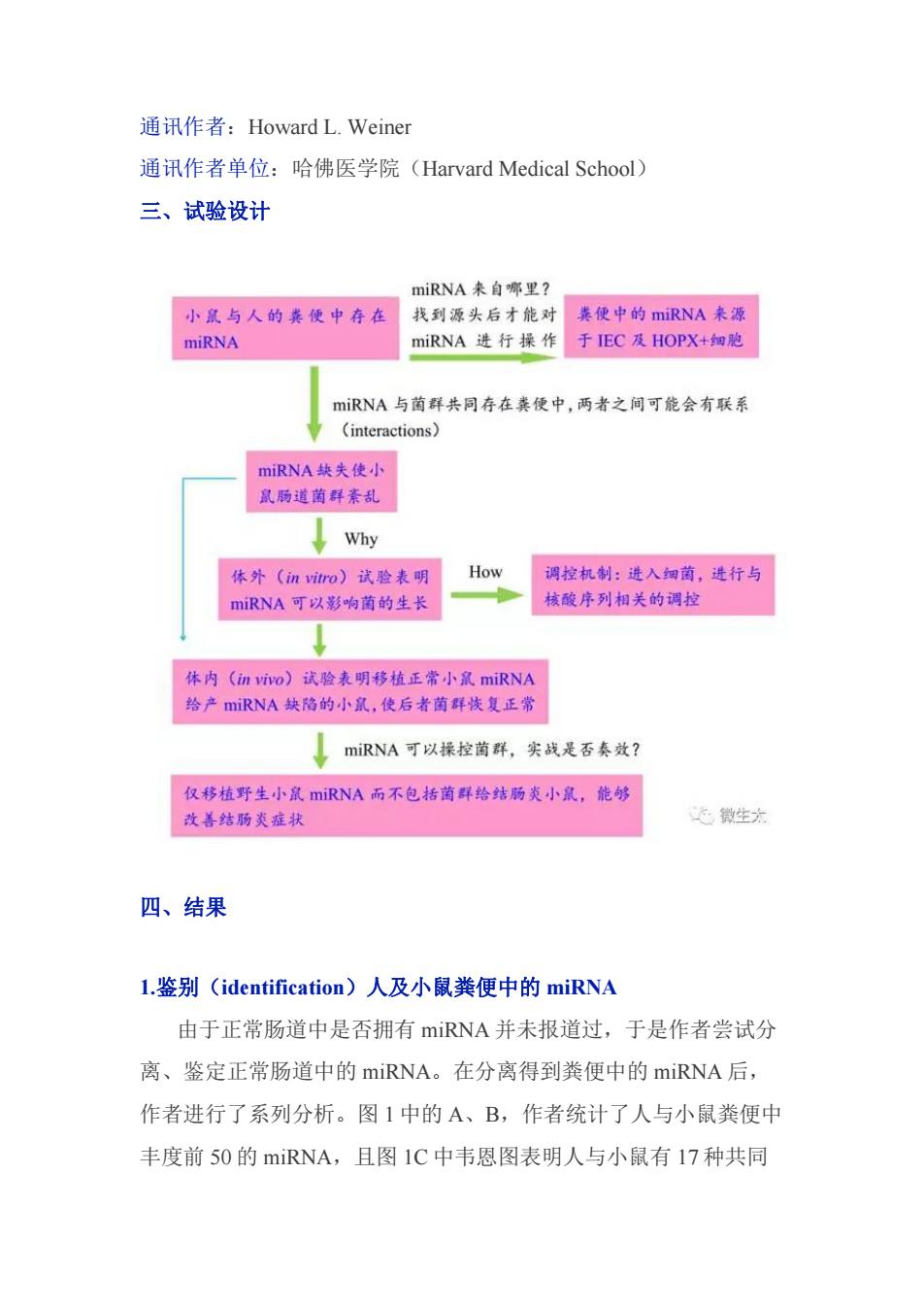
通讯作者:Howard L.Weiner 通讯作者单位:哈佛医学院(Harvard Medical School) 三、试验设计 nirna来自哪里? 小鼠与人的英便中存在 我到源头后才能对美便中的mRNA来源 miRNA miRNA进行操作 于IEC及HOPX+细胞 miRNA与菌群共同存在粪便中,两者之间可能会有联系 (interactions) DIRNA块失使小: 鼠肠道菌群素乱 ↓ Why 体外(in vitro)试验表明 How 调控机制:进入细菌,进行与 miRNA可以影响菌的生长 核酸序列相关的调拉 体内(miO)试验表明移植正常小鼠miRNA 给产miRNA缺格的小,使后者菌群恢复正常 miRNA可以操控菌群,实战是否奏效? 仅移植野生小鼠miRNA而不包括菌群给结肠炎小鼠,能够 改善结肠炎症状 四、结果 l.鉴别(identification)人及小鼠粪便中的miRNA 由于正常肠道中是否拥有miRNA并未报道过,于是作者尝试分 离、鉴定正常肠道中的miRNA。在分离得到粪便中的miRNA后, 作者进行了系列分析。图1中的A、B,作者统计了人与小鼠粪便中 丰度前50的miRNA,且图1C中韦恩图表明人与小鼠有17种共同
通讯作者:Howard L. Weiner 通讯作者单位:哈佛医学院(Harvard Medical School) 三、试验设计 四、结果 1.鉴别(identification)人及小鼠粪便中的 miRNA 由于正常肠道中是否拥有 miRNA 并未报道过,于是作者尝试分 离、鉴定正常肠道中的 miRNA。在分离得到粪便中的 miRNA 后, 作者进行了系列分析。图 1 中的 A、B,作者统计了人与小鼠粪便中 丰度前 50 的 miRNA,且图 1C 中韦恩图表明人与小鼠有 17 种共同
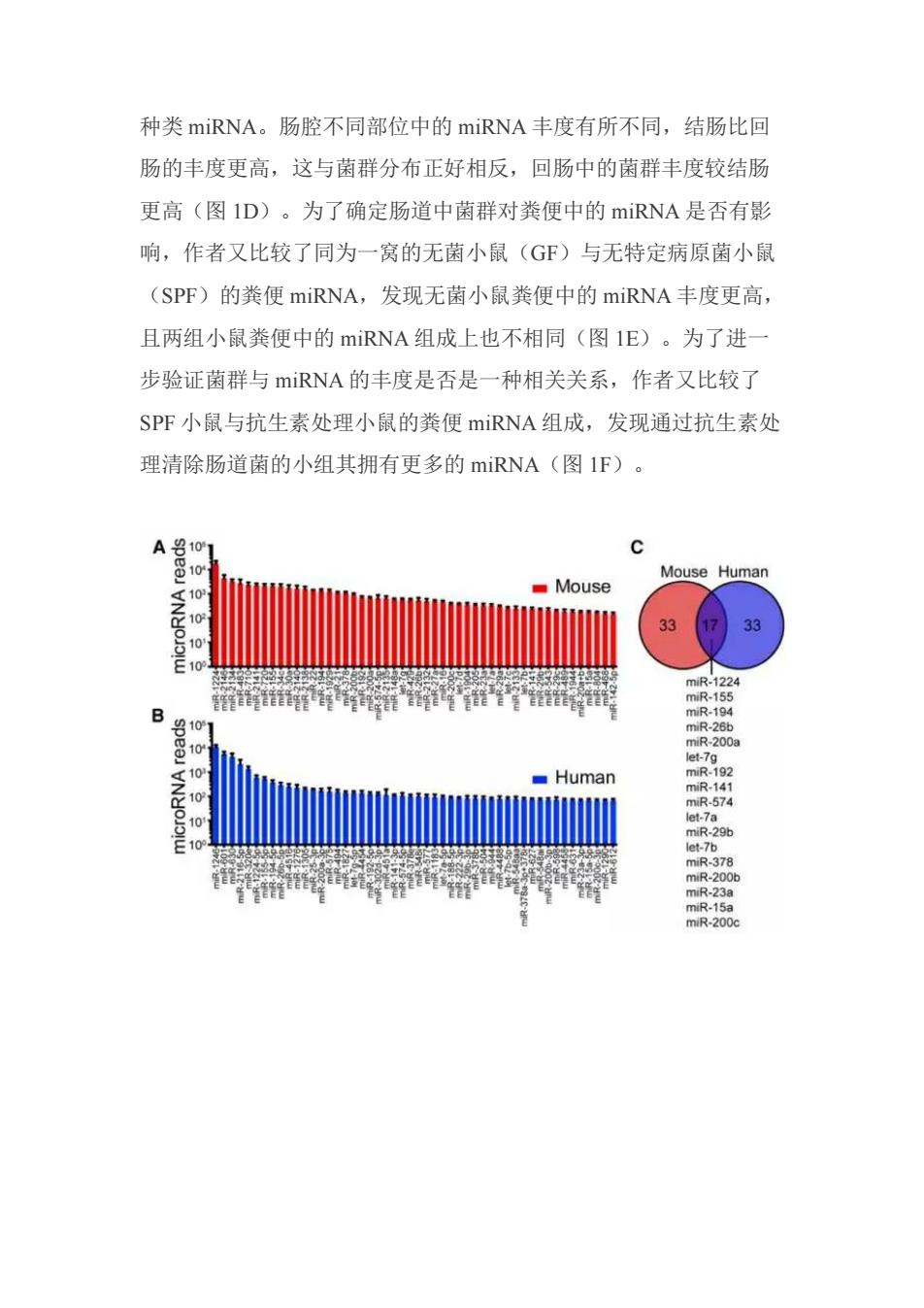
种类miRNA。肠腔不同部位中的miRNA丰度有所不同,结肠比回 肠的丰度更高,这与菌群分布正好相反,回肠中的菌群丰度较结肠 更高(图1D)。为了确定肠道中菌群对粪便中的miRNA是否有影 响,作者又比较了同为一窝的无菌小鼠(GF)与无特定病原菌小鼠 (SPF)的粪便miRNA,发现无菌小鼠粪便中的miRNA丰度更高, 且两组小鼠粪便中的miRNA组成上也不相同(图1E)。为了进一 步验证菌群与miRNA的丰度是否是一种相关关系,作者又比较了 SPF小鼠与抗生素处理小鼠的粪便miRNA组成,发现通过抗生素处 理清除肠道菌的小组其拥有更多的miRNA(图IF)。 A名1 ouse Humar Mouse 33 Human
种类 miRNA。肠腔不同部位中的 miRNA 丰度有所不同,结肠比回 肠的丰度更高,这与菌群分布正好相反,回肠中的菌群丰度较结肠 更高(图 1D)。为了确定肠道中菌群对粪便中的 miRNA 是否有影 响,作者又比较了同为一窝的无菌小鼠(GF)与无特定病原菌小鼠 (SPF)的粪便 miRNA,发现无菌小鼠粪便中的 miRNA 丰度更高, 且两组小鼠粪便中的 miRNA 组成上也不相同(图 1E)。为了进一 步验证菌群与 miRNA 的丰度是否是一种相关关系,作者又比较了 SPF 小鼠与抗生素处理小鼠的粪便 miRNA 组成,发现通过抗生素处 理清除肠道菌的小组其拥有更多的 miRNA(图 1F)

D E lleal vs Colonic content GF vs Colonized feces ABX vs SPF Colonic content 10 2 Log2 [Fo 图I鉴别(identification)粪便及肠腔内容物中的miRNA 注:D-F上侧为火山图,统计差异及差异显著性。横轴为L0g2 (X/X数量的倍数),纵轴为P值。图中小点表示某一种miRNA: 下侧为PCA分析,分析组成的相似性或差异性。 2.肠上皮细胞及表达Hopx阳性细胞是miRNA的两大主要来源 粪便中mRNA的来源先前未被报道过,因为有研究表明肠上皮 细胞(IECs)分泌外泌体(exosome),而作者发现miRNA存在于 外泌体中,他们就设计实验来验证miRNA是否源自ECs。Dicer酶 是在miRNA成熟过程中的必须酶,它参与miRNA的剪切。故作者 培育了IECs细胞无Dicer酶表达的小鼠(Dicer1△IEC),与IECs细 胞表达Dicer酶的小鼠(Dicer1fl/H)并比较两组小鼠粪便中的 miRNA的丰度,图2A中火山图表明IECs细胞无Dicer酶的小鼠, 其粪便中的miRNA减少了,所以可以得出IECs是miRNA的来源。 同样的方法,发现Hopx表达阳性细胞也是miRNA的来源(图 2B)。且在两种无Dicer酶的细胞中miRNA变化是不同的,故 Hopx+细胞与ECs负责产生不同的miRNA。同时作者又用另一种方
图 1 鉴别(identification)粪便及肠腔内容物中的 miRNA 注:D-F 上侧为火山图,统计差异及差异显著性。横轴为 Log2 (X/X 数量的倍数),纵轴为 P 值。图中小点表示某一种 miRNA; 下侧为 PCA 分析,分析组成的相似性或差异性。 2.肠上皮细胞及表达 Hopx 阳性细胞是 miRNA 的两大主要来源 粪便中 miRNA 的来源先前未被报道过,因为有研究表明肠上皮 细胞(IECs)分泌外泌体(exosome),而作者发现 miRNA 存在于 外泌体中,他们就设计实验来验证 miRNA 是否源自 IECs。Dicer 酶 是在 miRNA 成熟过程中的必须酶,它参与 miRNA 的剪切。故作者 培育了 IECs 细胞无 Dicer 酶表达的小鼠(Dicer1∆IEC),与 IECs 细 胞表达 Dicer 酶的小鼠(Dicer1fl/fl)并比较两组小鼠粪便中的 miRNA 的丰度,图 2A 中火山图表明 IECs 细胞无 Dicer 酶的小鼠, 其粪便中的 miRNA 减少了,所以可以得出 IECs 是 miRNA 的来源。 同样的方法,发现 Hopx 表达阳性细胞也是 miRNA 的来源(图 2B)。且在两种无 Dicer 酶的细胞中 miRNA 变化是不同的,故 Hopx+细胞与 IECs 负责产生不同的 miRNA。同时作者又用另一种方
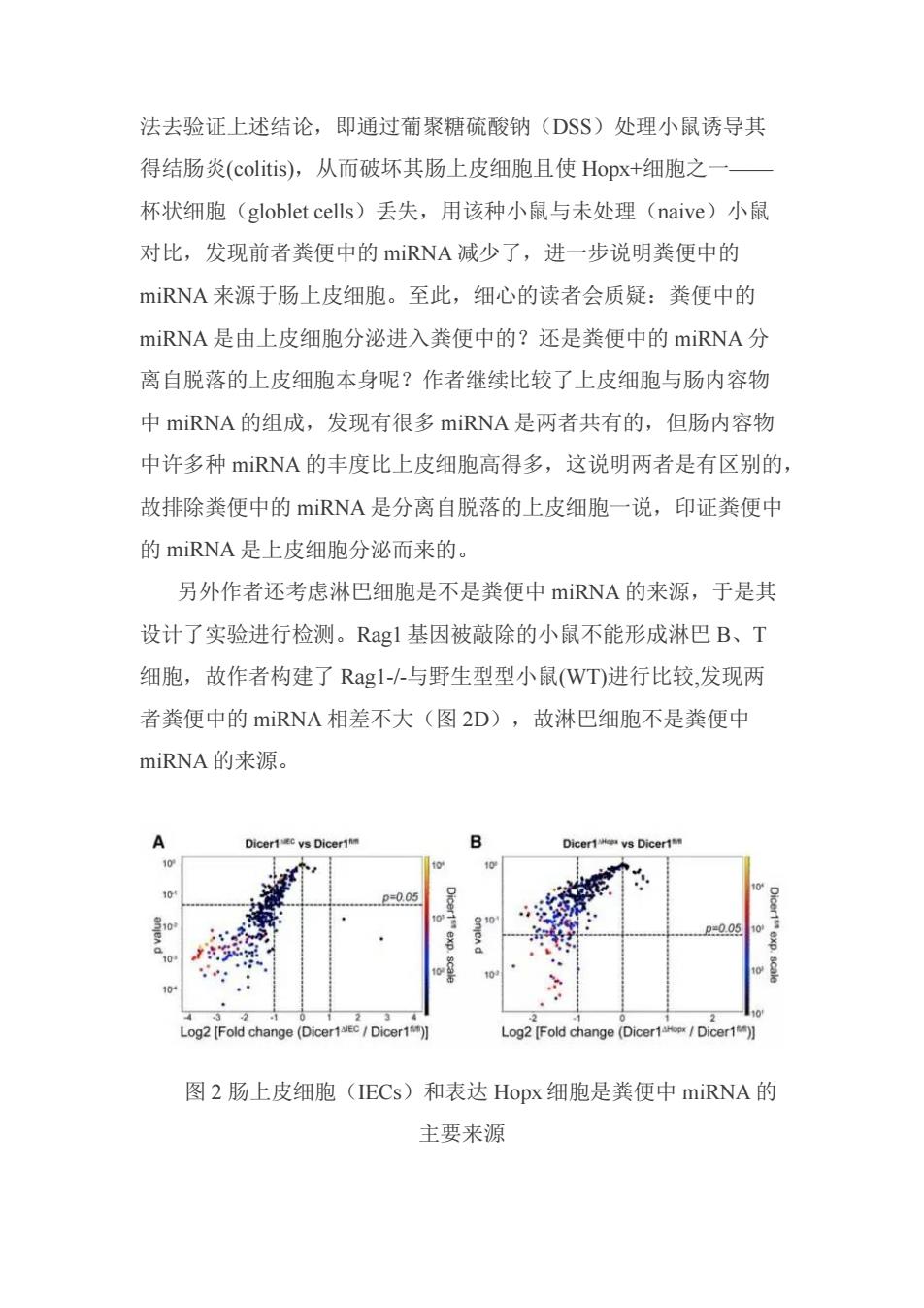
法去验证上述结论,即通过葡聚糖硫酸钠(DSS)处理小鼠诱导其 得结肠炎(colitis),从而破坏其肠上皮细胞且使Hopx+细胞之一 杯状细胞(globlet cells)丢失,用该种小鼠与未处理(naive)小鼠 对比,发现前者粪便中的mRNA减少了,进一步说明粪便中的 miRNA来源于肠上皮细胞。至此,细心的读者会质疑:粪便中的 miRNA是由上皮细胞分泌进入粪便中的?还是粪便中的miRNA分 离自脱落的上皮细胞本身呢?作者继续比较了上皮细胞与肠内容物 中miRNA的组成,发现有很多miRNA是两者共有的,但肠内容物 中许多种miRNA的丰度比上皮细胞高得多,这说明两者是有区别的, 故排除粪便中的miRNA是分离自脱落的上皮细胞一说,印证粪便中 的miRNA是上皮细胞分泌而来的。 另外作者还考虑淋巴细胞是不是粪便中miRNA的来源,于是其 设计了实验进行检测。Rgl基因被敲除的小鼠不能形成淋巴B、T 细胞,故作者构建了Ragl-与野生型型小鼠(WT进行比较,发现两 者粪便中的miRNA相差不大(图2D),故淋巴细胞不是粪便中 miRNA的来源。 Log2 IFold change(/Dicer1) Log2 [Fold change (Dicer1/Dicer1) 图2肠上皮细胞(IECs)和表达Hopx细胞是粪便中miRNA的 主要来源
法去验证上述结论,即通过葡聚糖硫酸钠(DSS)处理小鼠诱导其 得结肠炎(colitis),从而破坏其肠上皮细胞且使 Hopx+细胞之一—— 杯状细胞(globlet cells)丢失,用该种小鼠与未处理(naive)小鼠 对比,发现前者粪便中的 miRNA 减少了,进一步说明粪便中的 miRNA 来源于肠上皮细胞。至此,细心的读者会质疑:粪便中的 miRNA 是由上皮细胞分泌进入粪便中的?还是粪便中的 miRNA 分 离自脱落的上皮细胞本身呢?作者继续比较了上皮细胞与肠内容物 中 miRNA 的组成,发现有很多 miRNA 是两者共有的,但肠内容物 中许多种 miRNA 的丰度比上皮细胞高得多,这说明两者是有区别的, 故排除粪便中的 miRNA 是分离自脱落的上皮细胞一说,印证粪便中 的 miRNA 是上皮细胞分泌而来的。 另外作者还考虑淋巴细胞是不是粪便中 miRNA 的来源,于是其 设计了实验进行检测。Rag1 基因被敲除的小鼠不能形成淋巴 B、T 细胞,故作者构建了 Rag1-/-与野生型型小鼠(WT)进行比较,发现两 者粪便中的 miRNA 相差不大(图 2D),故淋巴细胞不是粪便中 miRNA 的来源。 图 2 肠上皮细胞(IECs)和表达 Hopx 细胞是粪便中 miRNA 的 主要来源

3.肠上皮细胞产miRNA缺陷使(同类型)小鼠肠道菌群组成相异度 增大 为了明确mRNA是否影响肠道菌群,作者通过高通量测序肠道 菌群的16 S rRNA基因的V4区并进行生物信息学分析。通过比较肠 上皮细胞Dicer酶缺陷小鼠与Dicer酶表达小鼠,发现两组小鼠之间 菌群组成的几个菌门(Phyla)发生了改变,如厚壁菌门 (Firmicutes)、变形菌门(Proteobacterial)。在科(family)水平 上,缺陷小鼠拟杆菌科(Bacteroidaceae)、螺杆菌科 (Helicobacteraceae)增多,普雷沃氏菌科(Prevotellaceae)、紫单胞 菌科(Porphyromonadaceae)、毛螺菌科(Lachnospiraceae)及疣微 菌科(Ruminococcaceae)减少。(图3A)PCoA分析显示,与正常 小鼠(WT)相比,缺陷小鼠组内小鼠之间肠道菌群差异较大(图 3B),同时图3C、3D中对所有菌科Unifrac分析也印证了上述现象。 (不加权Unifrac衡量系统发生相似性而加权Unifrac衡量丰度)。 进一步地,他们将上一步所有菌科限制到具体某一菌科,对野生型 小鼠(WT)中最多的3种菌科(Prevotellaceae Porphyromonadaceae、.Lachnospiraceae)进行距离分析(distance metric),图3E-3G显示缺陷小鼠与野生型小鼠之间存在明显,且 在Prevotellaceae、Porphyromonadaceae两个菌科上差异尤为明显。 然而,在Prevotellaceae方面却没有出现显著的beta多样性差异(补 充图2B-2D),这表明缺陷小鼠间的beta多样性要不是因为物种组 成上的差异、要不是因为特定细菌丰度上的差异造就的,为了确定 是哪一种原因造成的,作者又利用基于Bray-Curtis算法计算整体 OTUs,对属(Genus)水平的beta多样性进行分析(图3H),但由 于缺陷小鼠之间beta多样性太高,并没有找到缺陷小鼠与野生型小
3.肠上皮细胞产 miRNA 缺陷使(同类型)小鼠肠道菌群组成相异度 增大 为了明确 miRNA 是否影响肠道菌群,作者通过高通量测序肠道 菌群的 16S rRNA 基因的 V4 区并进行生物信息学分析。通过比较肠 上皮细胞 Dicer 酶缺陷小鼠与 Dicer 酶表达小鼠,发现两组小鼠之间 菌群组成的几个菌门(Phyla)发生了改变,如厚壁菌门 (Firmicutes)、变形菌门(Proteobacterial)。在科(family)水平 上,缺陷小鼠拟杆菌科(Bacteroidaceae)、螺杆菌科 (Helicobacteraceae)增多,普雷沃氏菌科(Prevotellaceae)、紫单胞 菌科(Porphyromonadaceae)、毛螺菌科(Lachnospiraceae)及疣微 菌科(Ruminococcaceae)减少。(图 3A)PCoA 分析显示,与正常 小鼠(WT)相比,缺陷小鼠组内小鼠之间肠道菌群差异较大(图 3B),同时图 3C、3D 中对所有菌科 Unifrac 分析也印证了上述现象。 (不加权 Unifrac 衡量系统发生相似性而加权 Unifrac 衡量丰度)。 进一步地,他们将上一步所有菌科限制到具体某一菌科,对野生型 小鼠(WT)中最多的 3 种菌科(Prevotellaceae、 Porphyromonadaceae、Lachnospiraceae)进行距离分析(distance metric),图 3E-3G 显示缺陷小鼠与野生型小鼠之间存在明显,且 在 Prevotellaceae、Porphyromonadaceae 两个菌科上差异尤为明显。 然而,在 Prevotellaceae 方面却没有出现显著的 beta 多样性差异(补 充图 2B-2D),这表明缺陷小鼠间的 beta 多样性要不是因为物种组 成上的差异、要不是因为特定细菌丰度上的差异造就的,为了确定 是哪一种原因造成的,作者又利用基于 Bray-Curtis 算法计算整体 OTUs,对属(Genus)水平的 beta 多样性进行分析(图 3H),但由 于缺陷小鼠之间 beta 多样性太高,并没有找到缺陷小鼠与野生型小

鼠之间的主要差异OTU,仅仅找到缺陷小鼠较低的10个OTUs及2 个较高OTUs。 P237 ● ● PC1(50.4%) PC3(13.4%) Dicer1Dicer1 微生大 0 p=0.0005 p00001 p=0.0010 02 02 family
鼠之间的主要差异 OTU,仅仅找到缺陷小鼠较低的 10 个 OTUs 及 2 个较高 OTUs

G 0.2 0.06 aiegen8 图3肠上皮细胞产iRNA缺陷使(同一环境)小鼠肠道菌群组成相 异度增大 注:已有理论是同样坏境下,宿主肠道菌群组成会更为接近。缺少 miRNA的存在,小鼠肠道菌群失去固有发展,两者关系好似指挥官 之于士兵、磁铁之于铁。C-H为箱线图,箱子中的一条线为中位数, 代表平均水平,箱子的宽度一定程度反应数据的波动程度。 Unifrac度量的标准是根据构建的进化树枝的长度计量两个不同 环境样品之间的差异,差异通过0-1距离值表示,进化树上最早分 化的树枝之间的距离为l,即差异最大。不加劝的(unweighted) Unifrac的计算是基于两样本间菌群组成差异,而加权的(weighted) Unifrac还将物种丰度考虑进去。 4.宿主miRNA影响肠道细菌生长 iRNA会使肠道菌群差异增大,能不能影响肠道细菌生长呢? 作者打算在体外(in vitro)验证一下。他们从肠道菌群中筛选了两 株菌,一株厌氧菌Fusobacterium nucleatum(Fn)及兼性厌氧菌 Escherichia coli(E.coli)准备培养用。作者想到细菌中是否存在 miRNA的核酸序列靶点,故将两株菌、节段丝状细菌(SFB)以及
图 3 肠上皮细胞产 miRNA 缺陷使(同一环境)小鼠肠道菌群组成相 异度增大 注:已有理论是同样坏境下,宿主肠道菌群组成会更为接近。缺少 miRNA 的存在,小鼠肠道菌群失去固有发展,两者关系好似指挥官 之于士兵、磁铁之于铁。C-H 为箱线图,箱子中的一条线为中位数, 代表平均水平,箱子的宽度一定程度反应数据的波动程度。 Unifrac 度量的标准是根据构建的进化树枝的长度计量两个不同 环境样品之间的差异,差异通过 0-1 距离值表示,进化树上最早分 化的树枝之间的距离为 1,即差异最大。不加劝的(unweighted) Unifrac 的计算是基于两样本间菌群组成差异,而加权的(weighted) Unifrac 还将物种丰度考虑进去。 4.宿主 miRNA 影响肠道细菌生长 miRNA 会使肠道菌群差异增大,能不能影响肠道细菌生长呢? 作者打算在体外(In vitro)验证一下。他们从肠道菌群中筛选了两 株菌,一株厌氧菌 Fusobacterium nucleatum (Fn)及兼性厌氧菌 Escherichia coli (E. coli)准备培养用。作者想到细菌中是否存在 miRNA 的核酸序列靶点,故将两株菌、节段丝状细菌(SFB)以及
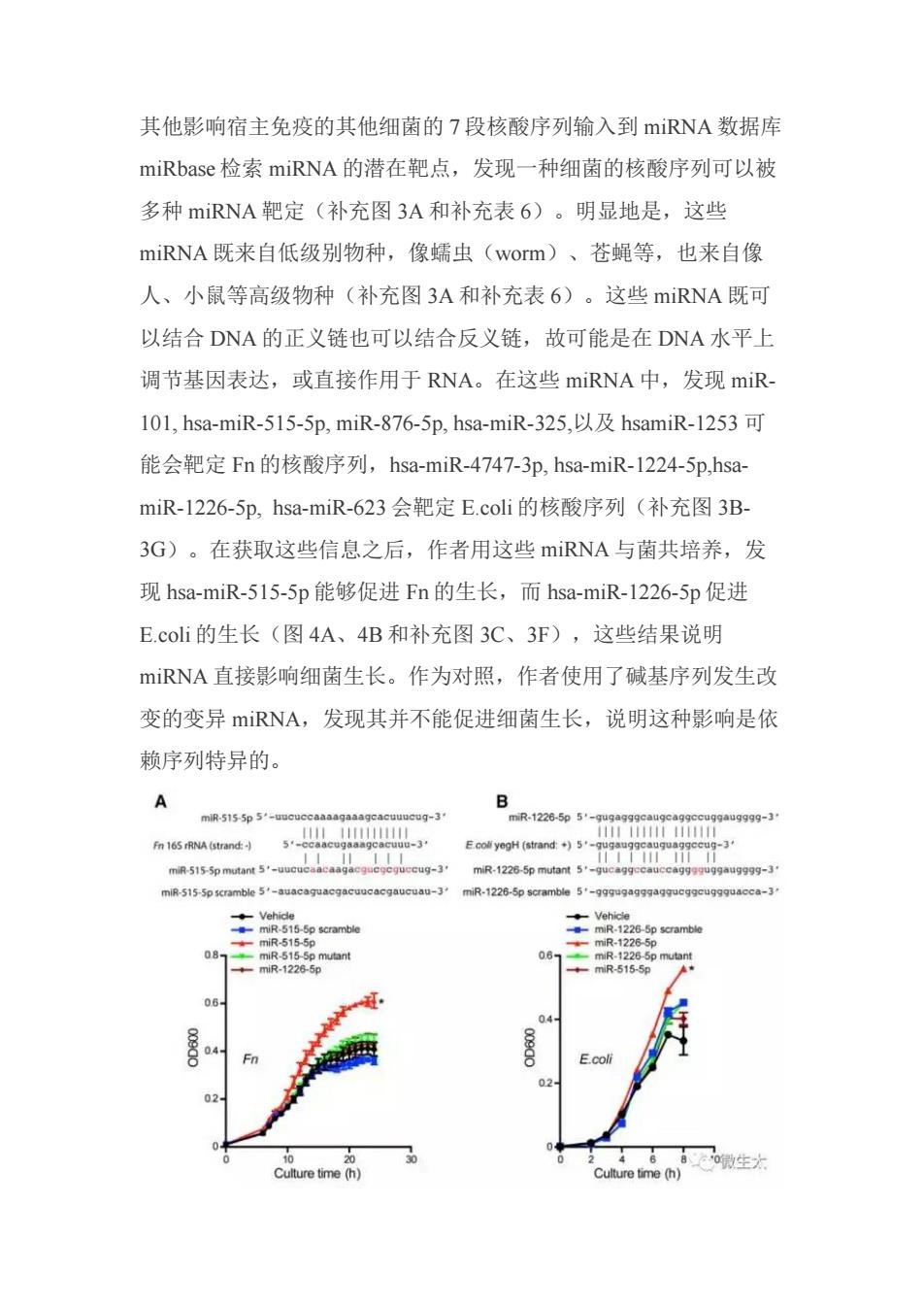
其他影响宿主免疫的其他细菌的7段核酸序列输入到miRNA数据库 miRbase检索miRNA的潜在靶点,发现一种细菌的核酸序列可以被 多种miRNA靶定(补充图3A和补充表6)。明显地是,这些 miRNA既来自低级别物种,像蠕虫(worm)、苍蝇等,也来自像 人、小鼠等高级物种(补充图3A和补充表6)。这些miRNA既可 以结合DNA的正义链也可以结合反义链,故可能是在DNA水平上 调节基因表达,或直接作用于RNA。在这些miRNA中,发现miR 101,hsa-miR-515-5p,miR-876-5p,hsa-miR-325,以及hsamiR-1253可 能会靶定Fn的核酸序列,hsa-miR-4747-3p,hsa-miR-1224-5p,hsa miR-1226-5p,hsa-miR-623会靶定E.coli的核酸序列(补充图3B- 3G)。在获取这些信息之后,作者用这些mRNA与菌共培养,发 现hsa-miR-515-5p能够促进Fn的生长,而hsa-miR-1226-5p促进 E.coli的生长(图4A、4B和补充图3C、3F),这些结果说明 iRNA直接影响细菌生长。作为对照,作者使用了碱基序列发生改 变的变异iRNA,发现其并不能促进细菌生长,说明这种影响是依 赖序列特异的。 A mR5155p5- EoolyogH (strand: 3 0-3+mR.1226.5 mR5 生 ure time (h】
其他影响宿主免疫的其他细菌的 7 段核酸序列输入到 miRNA 数据库 miRbase 检索 miRNA 的潜在靶点,发现一种细菌的核酸序列可以被 多种 miRNA 靶定(补充图 3A 和补充表 6)。明显地是,这些 miRNA 既来自低级别物种,像蠕虫(worm)、苍蝇等,也来自像 人、小鼠等高级物种(补充图 3A 和补充表 6)。这些 miRNA 既可 以结合 DNA 的正义链也可以结合反义链,故可能是在 DNA 水平上 调节基因表达,或直接作用于 RNA。在这些 miRNA 中,发现 miR- 101, hsa-miR-515-5p, miR-876-5p, hsa-miR-325,以及 hsamiR-1253 可 能会靶定 Fn 的核酸序列,hsa-miR-4747-3p, hsa-miR-1224-5p,hsa- miR-1226-5p, hsa-miR-623 会靶定 E.coli 的核酸序列(补充图 3B- 3G)。在获取这些信息之后,作者用这些 miRNA 与菌共培养,发 现 hsa-miR-515-5p 能够促进 Fn 的生长,而 hsa-miR-1226-5p 促进 E.coli 的生长(图 4A、4B 和补充图 3C、3F),这些结果说明 miRNA 直接影响细菌生长。作为对照,作者使用了碱基序列发生改 变的变异 miRNA,发现其并不能促进细菌生长,说明这种影响是依 赖序列特异的

图4宿主miRNA直接影响肠道细菌生长 5.宿主miRNA进入细菌调控细菌基因转录 先前报道miRNA能进入线粒体调控线粒体基因表达,而线粒体 进化起源于细菌是大家认可的理论,这些为miRNA调控细菌提供了 理论基础。为了弄清楚iRNA是如何影响细菌生长的,作者考虑到 上述的理论,假设miRNA也能进入细菌,因此用结合荧光(Cy3) 的miRNA培养表达绿色荧光蛋白(GFP)的大肠杆菌,发现 miRNAs进入细菌并于细菌核酸共定位,在Fn中也是如此(图SA 5D,补充图4B-4E,补充视频1、2)。他们进一步发现miRNA在 细菌中出现聚集情况(图5E、5F),从时间与空间上都提供 miRNA与细菌互相作用的证据。到此并不清楚,miRNA具体的作 用机制,考虑到miRNA可能会调控细菌的基因表达来调控菌的生长 作者用hsa-miR-515-5p培养Fn,并用Q-PCR统计16 S rRNA转录的 数量,结果发现Fn中16 S rRNA/23 S rRNA的比例增大了(图5G)。 相似的是,在大肠杆菌中miR-1226-5p使yegH增多(图5H)。综 上可以看出miRNA可以通过调控细菌的基因转录而影响细胞生长
图 4 宿主 miRNA 直接影响肠道细菌生长 5.宿主 miRNA 进入细菌调控细菌基因转录 先前报道 miRNA 能进入线粒体调控线粒体基因表达,而线粒体 进化起源于细菌是大家认可的理论,这些为 miRNA 调控细菌提供了 理论基础。为了弄清楚 miRNA 是如何影响细菌生长的,作者考虑到 上述的理论,假设 miRNA 也能进入细菌,因此用结合荧光(Cy3) 的 miRNA 培养表达绿色荧光蛋白(GFP)的大肠杆菌,发现 miRNAs 进入细菌并于细菌核酸共定位,在 Fn 中也是如此(图 5A- 5D,补充图 4B-4E,补充视频 1、2)。他们进一步发现 miRNA 在 细菌中出现聚集情况(图 5E、5F),从时间与空间上都提供 miRNA 与细菌互相作用的证据。到此并不清楚,miRNA 具体的作 用机制,考虑到 miRNA 可能会调控细菌的基因表达来调控菌的生长, 作者用 hsa-miR-515-5p 培养 Fn,并用 Q-PCR 统计 16S rRNA 转录的 数量,结果发现 Fn 中 16S rRNA/23S rRNA 的比例增大了(图 5G)。 相似的是,在大肠杆菌中 miR-1226-5p 使 yegH 增多(图 5H)。综 上可以看出 miRNA 可以通过调控细菌的基因转录而影响细胞生长