
CRISPR-Cas9基因编辑
CRISPR-Cas9基因编辑

基因组编辑技术 又称基因组定点修饰技术,通过在某些物种基因组中进行靶向特异 性的突变,从而解答并提出更多精确的生物学问题。 方法包括: •锌指核酸酶(Zinc-finger nuclease,ZFN)技术 转录激活因子样效应物核酸酶(Transcription activator-like effector nuclease,TALEN)技术 Cas9/gRNA系统(CRISPR-cas9技术)
又称基因组定点修饰技术,通过在某些物种基因组中进行靶向特异 性的突变,从而解答并提出更多精确的生物学问题。 方法包括: •锌指核酸酶(Zinc-finger nuclease,ZFN)技术 •转录激活因子样效应物核酸酶(Transcription activator-like effector nuclease,TALEN)技术 •Cas9/gRNA 系统(CRISPR-cas9技术) 基因组编辑技术
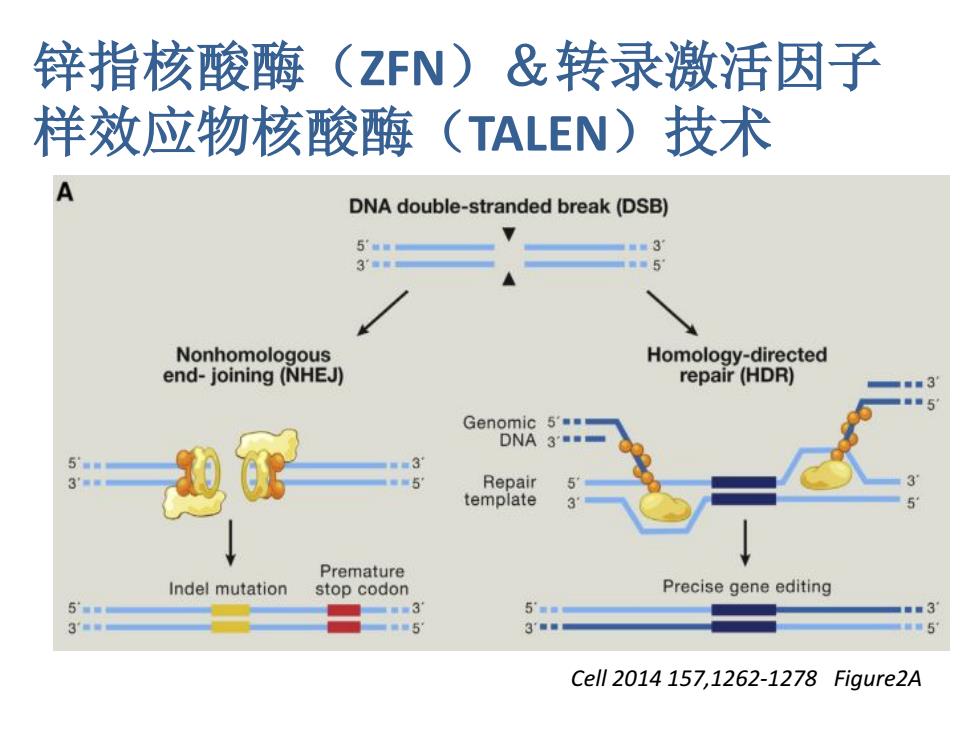
锌指核酸酶(ZFN)&转录激活因子 样效应物核酸酶(TALEN): 技术 DNA double-stranded break(DSB) 5 3 3 =5 Nonhomologous Homology-directed end-joining (NHEJ) repair (HDR) 3 Genomic 5 5 =w3 3= 5 Repair S template Premature Indel mutation stop codon Precise gene editing 年31 5' =每■31 量5 3■ 5 Cell2014157,1262-1278 Figure.2A
Cell 2014 157,1262-1278 Figure2B 不论是TALEN技术还是ZFN技术,其定向打靶都依赖于 DNA序列特异性结合蛋白模块的合成,将蛋白模块与限制性内切 酶 (FokⅠ) 的DNA切割域融合而得到。由蛋白特异结合域定位, DNA切割域剪切。 锌指核酸酶(ZFN)&转录激活因子 样效应物核酸酶(TALEN)技术 Cell 2014 157,1262-1278 Figure2A
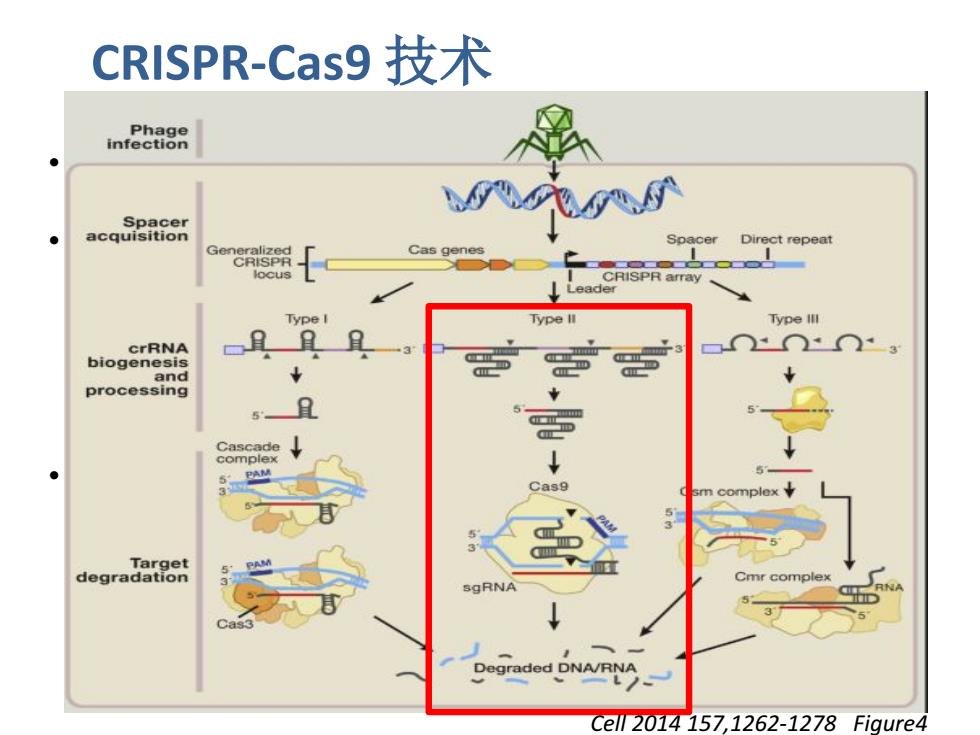
CRISPR-Cas9: 技术 Phage infection Spacer acquisition locus CRISPR crRNA biogenesis and processing Cas Target degradation Cmr complex ▣N Degraded DNA/RNA Ce2014157,1262-1278 Figure4
CRISPR-Cas9 技术 • 原理:规律性重复短回文序列簇(clustered regularly interspaced short palindromic repeats,CRISPRs) • CRISPR-Cas系统是细菌和古细菌在不断进化的过程中获得一 种适应性免疫防御机制,研究者根据 CRISPR-Cas 系统的特 性对此系统进行改造产生了第 3 代人工核酸内切酶技 术——CRISPR-Cas9技术。该技术通过小片段的 RNA 介导对 入侵的核酸进行靶向定位并通过Cas酶对核酸进行酶切、 降 解。 • CRISPR-Cas9 技术是一种多物种适应性的,位点特异性的有 效基因组编码工具。 Cell 2014 157,1262-1278 Figure4
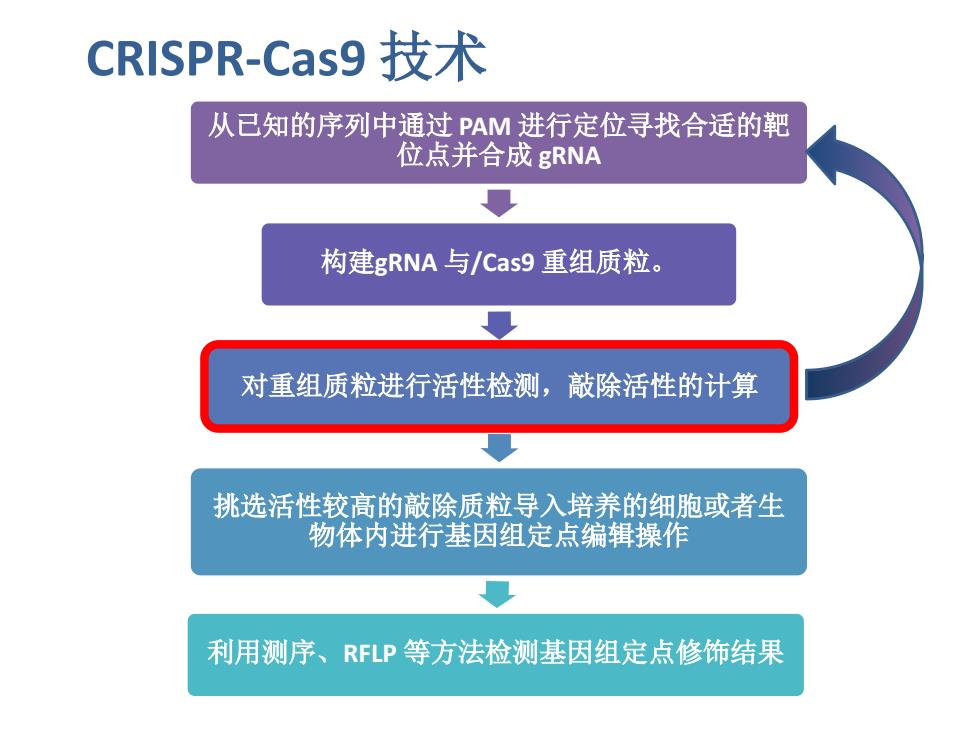
CRISPR-Cas9技术 从已知的序列中通过PAM进行定位寻找合适的靶 位点并合成gRNA 构建gRNA与/Cas9重组质粒。 对重组质粒进行活性检测,敲除活性的计算 挑选活性较高的敲除质粒导入培养的细胞或者生 物体内进行基因组定点编辑操作 利用测序、RFLP等方法检测基因组定点修饰结果
CRISPR-Cas9 技术 从已知的序列中通过 PAM 进行定位寻找合适的靶 位点并合成 gRNA 构建gRNA 与/Cas9 重组质粒。 对重组质粒进行活性检测,敲除活性的计算 挑选活性较高的敲除质粒导入培养的细胞或者生 物体内进行基因组定点编辑操作 利用测序、RFLP 等方法检测基因组定点修饰结果

SgRNA的设计 ·选择PAM(NGG)5端的一段碱基序列20nt作为原间隔序列,即 敲除的靶位点。(PAM,Protospacer adjacent motifs原间隔序列 临近基序。一般形式为NGG,其作用是将间隔序列定位于入侵 的噬菌体或质粒的DNA序列中) 原则: ·选择的序列必须在全基因组中进行比对,原间隔序列必须唯一, 否则会对其他基因进行敲除而出现错误的实验结果。 ·优先选择DSB位点的侧翼存在重复序列(如果DSB的侧翼存在重 复序列,在进行非同源末端重组时能够精确的介导断裂位点碱基 的缺失)。 ·PAM的5端尽量存在酶切位点。(有利于后期的活性检测)
sgRNA 的设计 • 选择 PAM(NGG)5‘端的一段碱基序列20nt作为原间隔序列,即 敲除的靶位点。(PAM,Protospacer adjacent motifs 原间隔序列 临近基序。一般形式为 NGG,其作用是将间隔序列定位于入侵 的噬菌体或质粒的 DNA 序列中) 原则: • 选择的序列必须在全基因组中进行比对,原间隔序列必须唯一, 否则会对其他基因进行敲除而出现错误的实验结果。 • 优先选择 DSB 位点的侧翼存在重复序列(如果 DSB 的侧翼存在重 复序列,在进行非同源末端重组时能够精确的介导断裂位点碱基 的缺失)。 • PAM 的5‘端尽量存在酶切位点。(有利于后期的活性检测)

构建重组质粒 构建方案: 1.一是将gRNA与Cas9连入同一个质粒载体中并以双启动子启动,载 体中携带荧光报告基因(用于流式细胞仪筛选)或抗性基因(用于 药物筛选) 2. 二是将gRNA与Cas9分别连载不同的载体上,两个载体携带有不同 的荧光报告基因或抗性基因 载体选择: 慢病毒载体 以HIV-1的主要功能元件为基础构建起来的转基因和基 因治疗载体。区别于腺病毒载体,可实现外源基因在目标细胞内稳定的 传代表达
构建重组质粒 构建方案 : 1. 一是将gRNA 与 Cas9 连入同一个质粒载体中并以双启动子启动,载 体中携带荧光报告基因(用于流式细胞仪筛选)或抗性基因(用于 药物筛选) 。 2. 二是将 gRNA与 Cas9 分别连载不同的载体上,两个载体携带有不同 的荧光报告基因或抗性基因 载体选择: 慢病毒载体 以HIV-1的主要功能元件为基础构建起来的转基因和基 因治疗载体。区别于腺病毒载体,可实现外源基因在目标细胞内稳定的 传代表达

gRNA的活性检测 限制性内切酶法 当Cas9/SERNA靶点位置中间序列存在限制性内切酶切位点时,如果 通过Cas9/SgRNA发生突变,这个位点将可能被破坏,而不能被内切酶 酶切。可采用电泳的方法估计突变效率,以突变效率的高低来衡量 sgRNA的活性。 非配对内切酶法 T7核酸内切酶I(T7 endonuclease I,T7E1)能够识别不完全配 对DNA并对其进行切割,如果通过Cas9/SgRNA发生突变,将基因组 DNA做PCR,将相对应的PCR产物与野生型DNA的PCR产物等量混合, 并退火杂交,将产生非配对DNA片段,将能被非配对内切酶T7E1剪切。 用电泳的方法估计突变效率,以突变效率的高低来衡量sgRNA的活性。 SSA活性检测 一个终止子插入luciferase(GFP)的编码区中央,luciferase(GFP) 就会失去活性。为检测Cas9/SgRNA剪切活性,将一个Cas9/SgRNA的靶 点位置序列插在终止子后。在Cas9/SgRNA的作用下,靶点位置产生 DSB,细胞通过同源重组方式修复DNA,形成一个有活性的luciferase。. 通过与对照组的比值变化就可反应Cas9/SgRNA剪切的活性水平
gRNA的活性检测 •限制性内切酶法 当 Cas9/sgRNA靶点位置中间序列存在限制性内切酶切位点时,如果 通过 Cas9/sgRNA 发生突变,这个位点将可能被破坏,而不能被 内切酶 酶切。可采用电泳的方法估计突变效率, 以突变效率的高低来衡量 sgRNA 的活性。 •非配对内切酶法 T7 核 酸 内 切 酶 I(T7 endonuclease I,T7E1) 能够识别不完全配 对 DNA 并对其进行切割,如果通过 Cas9/sgRNA 发生突变,将基因组 DNA 做 PCR,将相对应的 PCR 产物与野生型 DNA的 PCR 产物等量混合, 并退火杂交,将产生非配对DNA 片段,将能被非配对内切酶 T7E1 剪切。 用电泳的方法估计突变效率,以突变效率的高低来衡量 sgRNA 的活性。 •SSA 活性检测 一个终止子插入 luciferase(GFP)的编码区中央,luciferase(GFP) 就会失去活性。为检测 Cas9/sgRNA 剪切活性,将一个 Cas9/sgRNA的靶 点位置序列插在终止子后。在Cas9/ sgRNA 的作用下,靶点位置产生 DSB,细胞通过同源重组方式修复 DNA,形成一个有活性的luciferase。 通过与对照组的比值变化就可反应 Cas9/sgRNA 剪切的活性水平
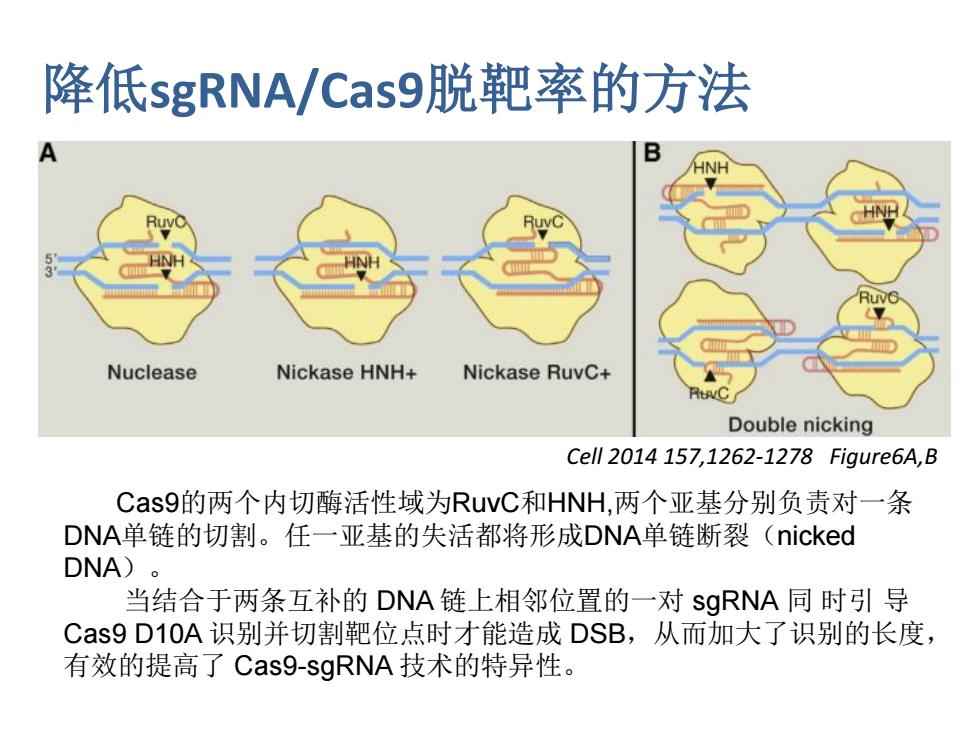
降低SgRNA/Cas9脱靶率的方法 Ruv Nuclease Nickase HNH+ Nickase RuvC+ Double nicking Cel∥2014157,1262-1278 Figure6A,B Cas9的两个内切酶活性域为RuvC和HNH,两个亚基分别负责对一条 DNA单链的切割。任一亚基的失活都将形成DNA单链断裂(nicked DNA)。 当结合于两条互补的DNA链上相邻位置的一对sgRNA同时引导 Cas9D1OA识别并切割靶位点时才能造成DSB,从而加大了识别的长度, 有效的提高了Cas9-sgRNA技术的特异性
降低sgRNA/Cas9脱靶率的方法 Cas9的两个内切酶活性域为RuvC和HNH,两个亚基分别负责对一条 DNA单链的切割。任一亚基的失活都将形成DNA单链断裂(nicked DNA)。 当结合于两条互补的 DNA 链上相邻位置的一对 sgRNA 同 时引 导 Cas9 D10A 识别并切割靶位点时才能造成 DSB,从而加大了识别的长度, 有效的提高了 Cas9-sgRNA 技术的特异性。 Cell 2014 157,1262-1278 Figure6A,B

重组质粒导入细胞/生物体 CBh SgRNA Cas9 Cas9 Cell2014157,1262-1278 Figure6C,D,E C、将编码Cas9和sgRNA的表达质粒直接转染至细胞系中。 D、将纯化的Cas9和体外表达的sgRNA显微注射至受精卵,快速得到转 基因动物模型。 E、将编码CRISPR组分的高效价的病毒载体转导至组织或细胞,完成特 定时间,组织的基因编辑
重组质粒导入细胞/生物体 C、将编码Cas9和sgRNA的表达质粒直接转染至细胞系中。 D、将纯化的Cas9和体外表达的sgRNA显微注射至受精卵,快速得到转 基因动物模型。 E、将编码CRISPR组分的高效价的病毒载体转导至组织或细胞,完成特 定时间,组织的基因编辑。 Cell 2014 157,1262-1278 Figure6C,D,E