
第七章动物基因组学与生物信息学 口第一节基因组学概念 口第二节基因组图谱 ▣第三节生物信息学 ▣第四节基因组的特征和应用 ▣第五节基因组对生物学研究的影响
1 第七章 动物基因组学与生物信息学 第一节基因组学概念 第二节基因组图谱 第三节生物信息学 第四节基因组的特征和应用 第五节基因组对生物学研究的影响

第一节、基因组学概念 基因组(genome)是德国遗传学家H.Winkler在 1920年将gene(基因)和chromosome(染色体)两 个词缩合而创造的一个新词,意思是指染色体上的全部 基因。几十年来,随着分子生物学的发展,其含义扩展 为在个体水平代表一个个体所有遗传性状的总和,在细 胞水平代表一个细胞所有不同染色体(单倍体)的总和, 在分子水平代表一个物种所有DNA分子的总和。 2
2 基因组(genome)是德国遗传学家H. Winkler在 1920年将gene(基因)和chromosome(染色体)两 个词缩合而创造的一个新词,意思是指染色体上的全部 基因。几十年来,随着分子生物学的发展,其含义扩展 为在个体水平代表一个个体所有遗传性状的总和,在细 胞水平代表一个细胞所有不同染色体(单倍体)的总和, 在分子水平代表一个物种所有DNA分子的总和。 第一节、基因组学概念

第二节 基因组图谱 、基本概念 o基因组作图主要分两个范畴:遗传作图(genetics mapping)和物理作图(physical mapping)。 o作图需要界标(landmark)或遗传标记(genetics marker)。常用的遗传标记(血型、蛋白质多态型、等位基因、 特定的DNA序列等)在早期构建遗传图时常用作制图的界标。 现在主要采用以下的界标: 限制性片段长度多态性(RFLP) 微卫星DNA多态性界标 单核苷酸多态性(SNP)界标 非多态的短单一序列作为界标 3
3 第二节 基因组图谱 一、基本概念 ○ 基因组作图主要分两个范畴:遗传作图(genetics mapping)和物理作图(physical mapping)。 ○ 作图需要界标(landmark)或遗传标记(genetics marker)。常用的遗传标记(血型、蛋白质多态型、等位基因、 特定的DNA序列等)在早期构建遗传图时常用作制图的界标。 现在主要采用以下的界标: 限制性片段长度多态性(RFLP) 微卫星DNA多态性界标 单核苷酸多态性(SNP)界标 非多态的短单一序列作为界标

二、遗传图 一)基本概念 应用遗传学技术构建能显示基因以及其它序列特征在基因组 上位置的图。 方法是以多态的遗传标记作为界标,计算细胞减数分裂过程 中遗传标记之间发生重组的频率,来确定两个遗传标记在染色 体上的相对位置。 遗传标记之间的相对距离即图距以厘摩(cM,厘摩尔根 centi-Morgan)为单位。当两个遗传标记之间的重组值为 1%时,图距即为1cM。 4
4 二、遗传图 (一)基本概念 应用遗传学技术构建能显示基因以及其它序列特征在基因组 上位置的图。 方法是以多态的遗传标记作为界标,计算细胞减数分裂过程 中遗传标记之间发生重组的频率,来确定两个遗传标记在染色 体上的相对位置。 遗传标记之间的相对距离即图距以厘摩(cM,厘摩尔根, centi-Morgan)为单位。当两个遗传标记之间的重组值为 1%时,图距即为1cM

经典遗传图的作图最常用的是三点测交法。 现代遗传图的概念是于1980年提出的,就是将单纯的表型多 态性界标改变为以DNA序列的多态作为作图界标。 各种遗传界标可在国际互联网上可以查阅 (http/www.gdb.org)。 当用DNA序列多态作为界标的遗传图时,一但确定该DNA界 标与某一基因的具体位置,便可分离克隆这个基因。 人类第一张以RFLP为界标的遗传图发表于1987年。 5
5 经典遗传图的作图最常用的是三点测交法。 现代遗传图的概念是于1980年提出的,就是将单纯的表型多 态性界标改变为以DNA序列的多态作为作图界标。 各种遗传界标可在国际互联网上可以查阅 (http://www.gdb.org)。 当用DNA序列多态作为界标的遗传图时,一但确定该DNA界 标与某一基因的具体位置,便可分离克隆这个基因。 人类第一张以RFLP为界标的遗传图发表于1987年

(二)遗传图的局限性 分辨率有限高等真核生物子代数量有限,只有少数的减数分裂 事件可供研究,连锁分析的分辨率受很大限制 人类基因组测序要求每100kb有一个标记,1996年发表的人类 遗传图达到每0.6Mb一个标记(1Mb=1000kb) 精确度较低假设交换是随机发生的,但由于交换热点的存在使 某一区段的交换频率远高于其它区段,无法绘制精确的遗传图。 6
6 (二)遗传图的局限性 分辨率有限 高等真核生物子代数量有限,只有少数的减数分裂 事件可供研究,连锁分析的分辨率受很大限制 人类基因组测序要求每100kb有一个标记,1996年发表的人类 遗传图达到每0.6Mb一个标记(1Mb=1000kb) 精确度较低 假设交换是随机发生的,但由于交换热点的存在使 某一区段的交换频率远高于其它区段,无法绘制精确的遗传图

三、物理图 (一))基本概念 应用分子生物学技术来直接分析DNA分子,从而构建能显示包括基 因在内的序列特征的位置图。 以特定的DNA序列为界标直接排列在基因组DNA分子上,这些特定的 DNA序列可以是多态的(如RFLP),但主要是非多态的(如STS、 STR、EST)和特定的基因序列等。 物理图中界标之间的距离单位依作图方法而定,辐射育种作图单位为厘 镭(cR),限制性作图单位元以物理长度,即碱基对数目(bp、kb) 来表示
7 三、物理图 (一)基本概念 应用分子生物学技术来直接分析DNA分子,从而构建能显示包括基 因在内的序列特征的位置图。 以特定的DNA序列为界标直接排列在基因组DNA分子上,这些特定的 DNA序列可以是多态的(如RFLP),但主要是非多态的(如STS、 STR、EST)和特定的基因序列等。 物理图中界标之间的距离单位依作图方法而定,辐射育种作图单位为厘 镭(cR),限制性作图单位元以物理长度,即碱基对数目(bp、kb) 来表示

(二)作图的基本方法 1,限制性作图 将限制性酶切位点标定在DNA分子的相对 位置上。 限制性内切酶有工型、Ⅱ型、Ⅲ型,I型、Ⅲ型酶位点特 殊性不强,Ⅱ型专一性很强。 6bp识别序列的限制性内切酶适用于50kb以下的DNA 分子作图。 限制性作图方法是:比较一个DNA分子被两种酶切割产生 的两套片段。 8
8 (二)作图的基本方法 1.限制性作图——将限制性酶切位点标定在DNA分子的相对 位置上。 限制性内切酶有Ⅰ型、Ⅱ型、Ⅲ型,Ⅰ型、Ⅲ型酶位点特 殊性不强,Ⅱ型专一性很强。 6 bp识别序列的限制性内切酶适用于50 kb以下的DNA 分子作图。 限制性作图方法是:比较一个DNA分子被两种酶切割产生 的两套片段
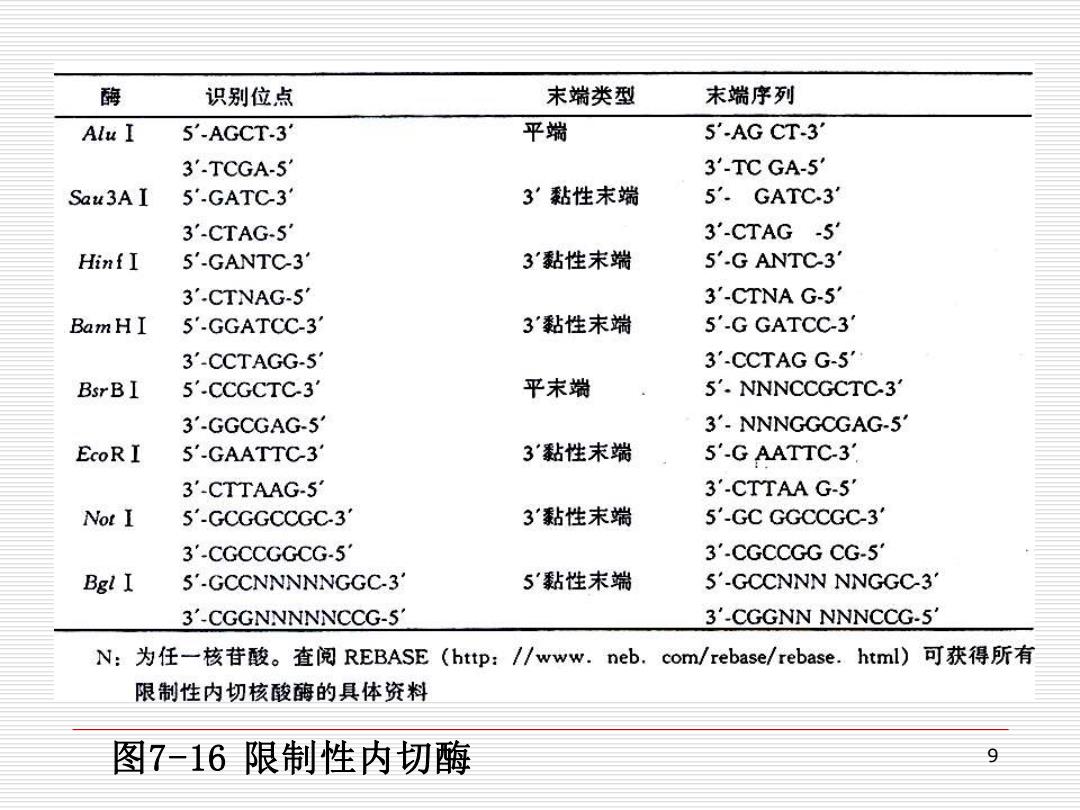
酶 识别位点 末端类型 末端序列 Alu I 5'-AGCT-3' 平端 5'-AG CT-3' 3'-TCGA-5' 3'-TC GA-5' Sau3A I 5'-GATC-3' 3'黏性末端 5'GATC-3' 3'-CTAG-5' 3'-CTAG-5' Hinf I 5'-GANTC-3' 3'黏性末端 5'-G ANTC-3' 3CTNAG-5' 3'-CTNA G-5' BamHI 5'-GGATCC-3' 3'黏性末端 5'-G GATCC-3' 3'-CCTAGG-5 3'-CCTAG G-5' BsrBI 5'-CCGCTC-3' 平末端 5'NNNCCGCTC3' 3-GGCGAG-5' 3-NNNGGCGAG-5' EcoR I 5'-GAATTC-3' 3'黏性末端 5'-G AATTC-3 3'-CTTAAG-5' 3'-CTTAA G-5' Not I 5-GCGGCCGC-3 3黏性末端 5'-GC GGCCGC-3' 3'-CGCCGGCG-5 3'-CGCCGG CG-5' BglI 5'-GCCNNNNNGGC-3' 5黏性末端 5'-GCCNNN NNGGC-3' 3'-CGGNNNNNCCG-5' 3'-CGGNN NNNCCG-5' N:为任一核苷酸。查阅REBASE(http://www.neb.com/rebase/rebase.html)可获得所有 限制性内切核酸酶的具体资料 图7-16限制性内切酶
图 7 -16 限制性内切酶 9

3,序列标记位点(sequence tagged site,STS)作图 通过PCR或分子杂交将小段DNA顺序定位在基因组的DNA区段中。 是目前用于构建最为详尽的大基因组物理图的主流技术。 OSTS作图原理 STS是一段短的DNA序列(100一500)bp,每个基因组只有 一个拷贝。 当两个片段含有同一STS时,可确认这两个片段重叠。 两个不同的STS出现在同一片段的机会取决于它们在基因组中的 位置,彼此接近,同时出现在同片段的机会就大,反之则小。 两个标记间的图距根据分离频率来计算。 10
10 3.序列标记位点(sequence tagged site, STS)作图—— 通过PCR或分子杂交将小段DNA顺序定位在基因组的DNA区段中。 是目前用于构建最为详尽的大基因组物理图的主流技术。 ○ STS作图原理 STS是一段短的DNA序列(100—500)bp,每个基因组只有 一个拷贝。 当两个片段含有同一STS时,可确认这两个片段重叠。 两个不同的STS出现在同一片段的机会取决于它们在基因组中的 位置,彼此接近,同时出现在同一片段的机会就大,反之则小。 两个标记间的图距根据分离频率来计算