
第一节:测序技术概述 0 、测序技术概述 ·第一代测序技术 ·由Frederick Sanger在1977年提出的双脱氧终止法 读长长(1000bp) ·高准确性、低通量、高成本 ·第二代测序技术 也叫下一代测序(Next Generation Sequencing, NGS) 读长短(小于500bp,通常低于100bp) 高准确性、高通量、高灵敏度、低成本 ·第三代测序技术 ·单分子测序技术 读长超长(可达70kb) 超高灵敏度(单分子)、准确性相对低、通量相对低、成本相对高 2
测序技术概述 第一节:测序技术概述 2 • 第一代测序技术 • 由Frederick Sanger在1977年提出的双脱氧终止法 • 读长长(~1000bp) • 高准确性、低通量、高成本 • 第二代测序技术 • 也叫下一代测序(Next Generation Sequencing,NGS) • 读长短(小于500bp,通常低于100bp) • 高准确性、高通量、 高灵敏度、低成本 • 第三代测序技术 • 单分子测序技术 • 读长超长(可达70kb) • 超高灵敏度(单分子)、准确性相对低、通量相对低、成本相对高

第二节:第二代测序原理 00八 第二代测序概述 ·问题:如何提高测序通量? 将DNA序列分割为多个小片段,每个片段单独扩增(放大信 号)后并行化测序,再通过算法拼接出原始序列 如何单独扩增? ·乳液PCR(Emulsion PCR) 固相桥式扩增(Solid--Phase bridge amplification) 固相模板移位(Solid-Phase Template Walking) DNA纳米球(DNA nanoball) 如何并行测序? ·基于连接的测序 (Sequencing By Ligation,SBL) 基于合成的测序(Sequencing By Synthesis,SBs) 3
第二代测序概述 第二节:第二代测序原理 3 • 问题:如何提高测序通量? • 将DNA序列分割为多个小片段,每个片段单独扩增(放大信 号)后并行化测序,再通过算法拼接出原始序列 • 如何单独扩增? • 乳液PCR(Emulsion PCR) • 固相桥式扩增(Solid-Phase bridge amplification) • 固相模板移位( Solid-Phase Template Walking) • DNA纳米球(DNA nanoball) • 如何并行测序? • 基于连接的测序(Sequencing By Ligation, SBL) • 基于合成的测序(Sequencing By Synthesis, SBS)

第二节:第二代测序原理 000八 乳液PCR(Emulsion PCR) a Emulsion PCR (454(Roche).SOLiD (Thermo Fisher).( GeneReader (Qlagen).lon Torrent (Thermo Fisher)) Emulsion On-bead amplification Final product Micelle droplets are loaded lemplates hybridize to bead-bound primers and are amplifed: 100-200 mitlion beads with with primer.template. after amplifcation,the complement strand disassoclates. thousands of bound template dNTPs and polymerate leaving bead-bound ssDNA templates 。 将单链DNA文库固定在带有引物的磁珠上,用扩增试剂乳化得 到油包水的乳液体系,使得每个液滴仅包含一个带模板的磁珠 (或者不包含) PC反应在每个液滴内独立进行,扩增完成后,磁珠表面可得到 相当数量(多达1,000,000) 的模板拷贝用于后续测序 使用该种扩增方式的有Rhoche454、Thermo Fisher SOLiD Thermo Fisher lon Torrent Qiagen GeneReader 4
乳液PCR(Emulsion PCR) 第二节:第二代测序原理 4 • 将单链DNA文库固定在带有引物的磁珠上,用扩增试剂乳化得 到油包水的乳液体系,使得每个液滴仅包含一个带模板的磁珠 (或者不包含) • PCR反应在每个液滴内独立进行,扩增完成后,磁珠表面可得到 相当数量(多达1,000,000)的模板拷贝用于后续测序 • 使用该种扩增方式的有Rhoche 454、Thermo Fisher SOLiD Thermo Fisher Ion Torrent、Qiagen GeneReader

第二节:第二代测序原理 000八 固相桥式扩增(Solid-Phase bridge amplification) b Solid-phase bridge amplification 用amina) Template binding Free templates hybridize with slide-bound adapters Patterned flow cell Microwells on几wcell direct cluster generation increasing cluster density Bridge amplification Cluster generation Distal ends of hybridized templates After several rounds of interact with nearby primers where amplification.100-200 million amplifcation can take place clonal clusters are formed 将3'和5'引物固定在载玻片上,模板稀释后与之随机结合, 形成两头均连接在载玻片上的桥状结构,扩增时使用模板结合 处附近的其他引物扩增。扩增完成后,载玻片表面可形成以模 板链为中心的模板拷贝簇(cluster)),这些簇将被用于后续测 序 由于簇和簇之间有可能重叠,现在开发了pattern flow cell技术 这是在载玻片的基础上,用细管隔出一个个微小的小室,保证 每个模板在单独的小室内扩增,而不和其他模板的扩增重叠。 使用该种扩增方式的有llumina 5
固相桥式扩增(Solid-Phase bridge amplification) 第二节:第二代测序原理 5 • 将3’和5’引物固定在载玻片上,模板稀释后与之随机结合, 形成两头均连接在载玻片上的桥状结构,扩增时使用模板结合 处附近的其他引物扩增。扩增完成后,载玻片表面可形成以模 板链为中心的模板拷贝簇(cluster),这些簇将被用于后续测 序 • 由于簇和簇之间有可能重叠,现在开发了pattern flow cell技术, 这是在载玻片的基础上,用细管隔出一个个微小的小室,保证 每个模板在单独的小室内扩增,而不和其他模板的扩增重叠。 • 使用该种扩增方式的有Illumina
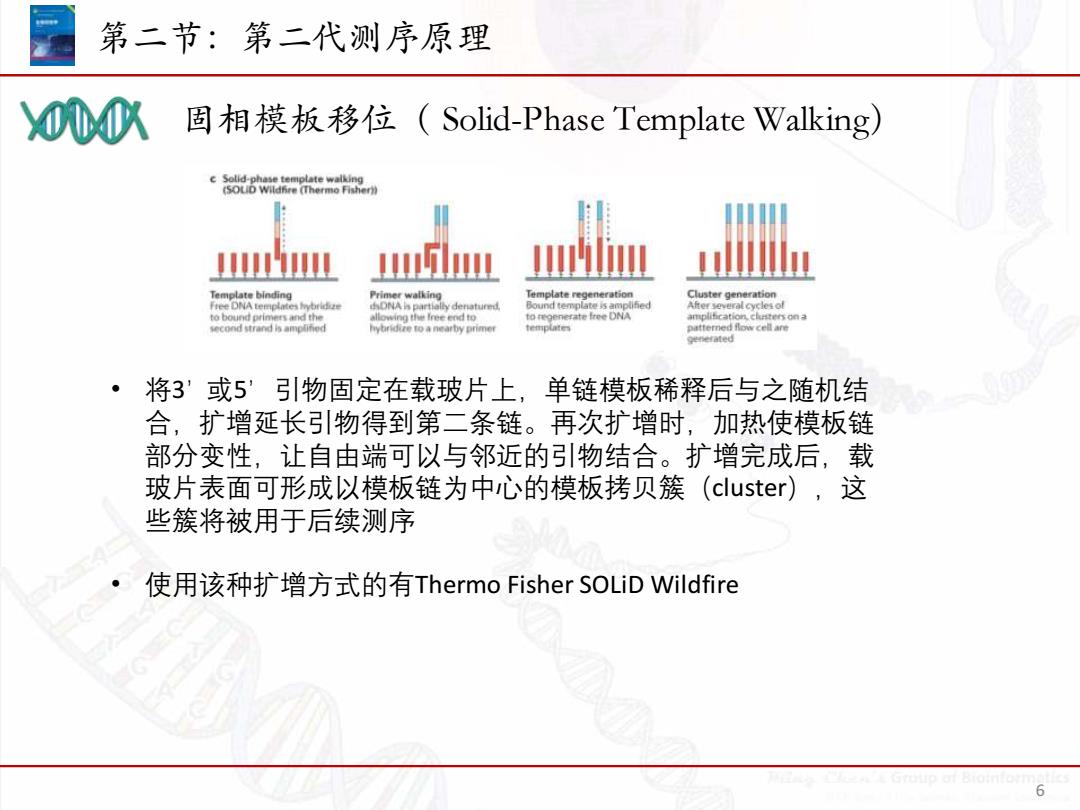
第二节:第二代测序原理 000八 固相模板移位(Solid-Phase Template Walking) c Solid-phase template walking 5 OUD wild锅re (Thermo月her? Template binding Primer walking Template regeneration Cluster generation dhDNA partially denatured Bound template is amplified After several cycles of tob0ndp时lmt5dthe allowing the Iree end to to regenerate free DNA amplifcation cluasters on a second strand is amplifed hybridize to a nearby primer templates patterned flow cell are generated ·将3'或5'引物固定在载玻片上,单链模板稀释后与之随机结 合,扩增延长引物得到第二条链。再次扩增时,加热使模板链 部分变性,让自由端可以与邻近的引物结合。扩增完成后,载 玻片表面可形成以模板链为中心的模板拷贝簇(cluster),这 些簇将被用于后续测序 使用该种扩增方式的有Thermo Fisher SOLiD Wildfire 6
固相模板移位( Solid-Phase Template Walking) 第二节:第二代测序原理 6 • 将3’或5’引物固定在载玻片上,单链模板稀释后与之随机结 合,扩增延长引物得到第二条链。再次扩增时,加热使模板链 部分变性,让自由端可以与邻近的引物结合。扩增完成后,载 玻片表面可形成以模板链为中心的模板拷贝簇(cluster),这 些簇将被用于后续测序 • 使用该种扩增方式的有Thermo Fisher SOLiD Wildfire

第二节:第二代测序原理 000八 DNA纳米球(DNA nanoball) d In-solution DNA nanoball generation (Complete Genomics (BGl)) Rolling circle amplifcation Circalar templates are amplifed to generated long Cleavage concatamers.called DNA nanoballk intermolecular Circular DNA nteractions keep the nanoballs cohesive and templates separate in solution are cleaved downstream of the adapter sequence Adapter ligation lterative ligation One set of adapters Three additional rounds of ligation. end of a DNA circularization and template.followed cleavage generate a Hybridization by template circular template with DNA nanobalis are four d由Jerent adapters immobllized on a patterned flow cell Nature Reviewsi Genetics DNA模板通过迭代的连接、环化、切割,形成一个带有4个接头 的环形模板。环形模板通过滚环扩增形成一条带有多个序列拷 贝的超长DNA,在溶液环境中会自然卷曲成纳米球结构,再将 DNA纳米球固定到patternd flow cell_上以用于后续测序 使用该种扩增方式的有华大基因的Complete Genomics
DNA纳米球(DNA nanoball) 第二节:第二代测序原理 7 • DNA模板通过迭代的连接、环化、切割,形成一个带有4个接头 的环形模板。环形模板通过滚环扩增形成一条带有多个序列拷 贝的超长DNA,在溶液环境中会自然卷曲成纳米球结构,再将 DNA纳米球固定到patternd flow cell上以用于后续测序 • 使用该种扩增方式的有华大基因的Complete Genomics

第二节:第二代测序原理 O0八基于连接的测序(Sequencing By Ligation,SBL) ·DNA模板通过迭代的连接、环化、 切割,形成一个带有4个接头的 环形模板。环形模板通过滚环 扩增形成一条带有多个序列拷 贝的超长DNA,在溶液环境中会 自然卷曲成纳米球结构,再将 DNA纳米球固定到patternd flow cel上以用于后续测序 使用该种扩增方式的有华大基 因的Complete Genomics 8
基于连接的测序(Sequencing By Ligation, SBL) 第二节:第二代测序原理 8 • DNA模板通过迭代的连接、环化、 切割,形成一个带有4个接头的 环形模板。环形模板通过滚环 扩增形成一条带有多个序列拷 贝的超长DNA,在溶液环境中会 自然卷曲成纳米球结构,再将 DNA纳米球固定到patternd flow cell上以用于后续测序 • 使用该种扩增方式的有华大基 因的Complete Genomics
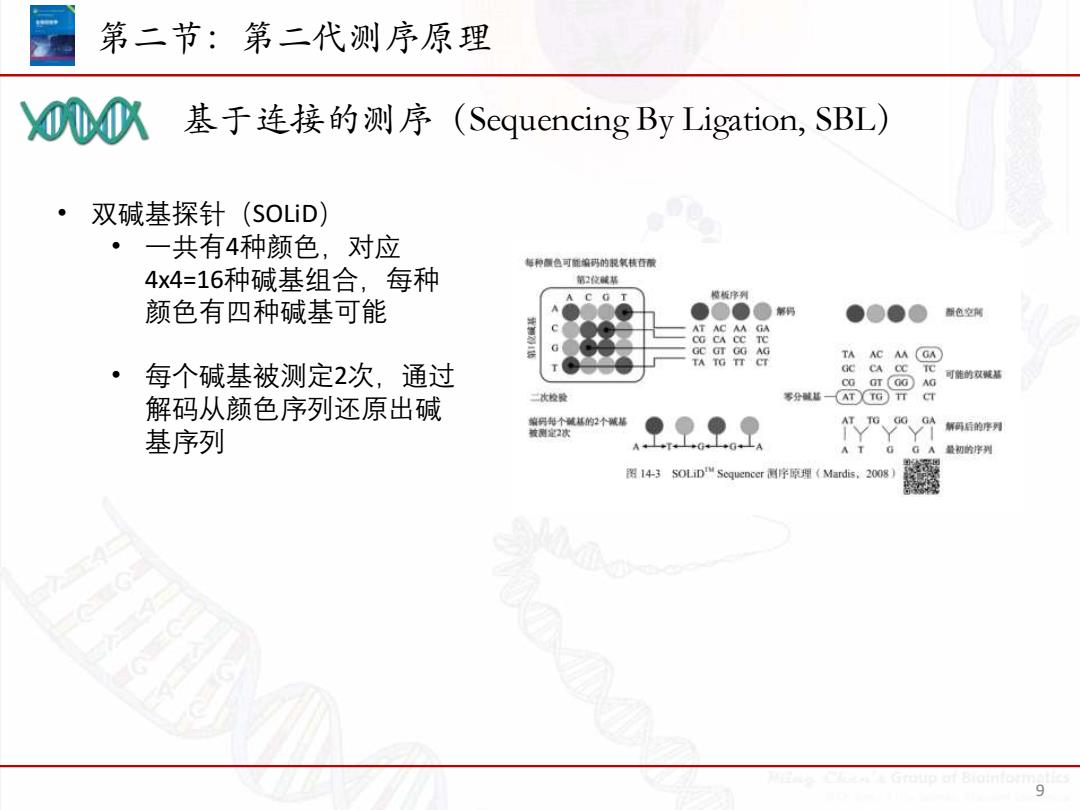
第二节:第二代测序原理 个八基于连接的测序(Sequencing By Ligation,SBL) ·双碱基探针(SOLiD) 一共有4种颜色,对应 每种衡色可新编码的权氧核香而 4x4=16种碱基组合,每种 第2位碱某 榄板序列 颜色有四种碱基可能 ●●● 解码 服色空展 CG 9 TA TG TA AC A GA ·每个碱基被测定2次,通过 Gc CA cc C 可陆的双碱基 AG 解码从颜色序列还原出碱 次检贸 零分藏超一(AT(TGT行 cT 编丹纯个碱框的2个城招 被测定2次 ●● ATG、 解码后的序列 基序列 最初的序列 图143 SOLiDM Sequencer测序原理(Mardis,2008) 9
基于连接的测序(Sequencing By Ligation, SBL) 第二节:第二代测序原理 9 • 双碱基探针(SOLiD) • 一共有4种颜色,对应 4x4=16种碱基组合,每种 颜色有四种碱基可能 • 每个碱基被测定2次,通过 解码从颜色序列还原出碱 基序列
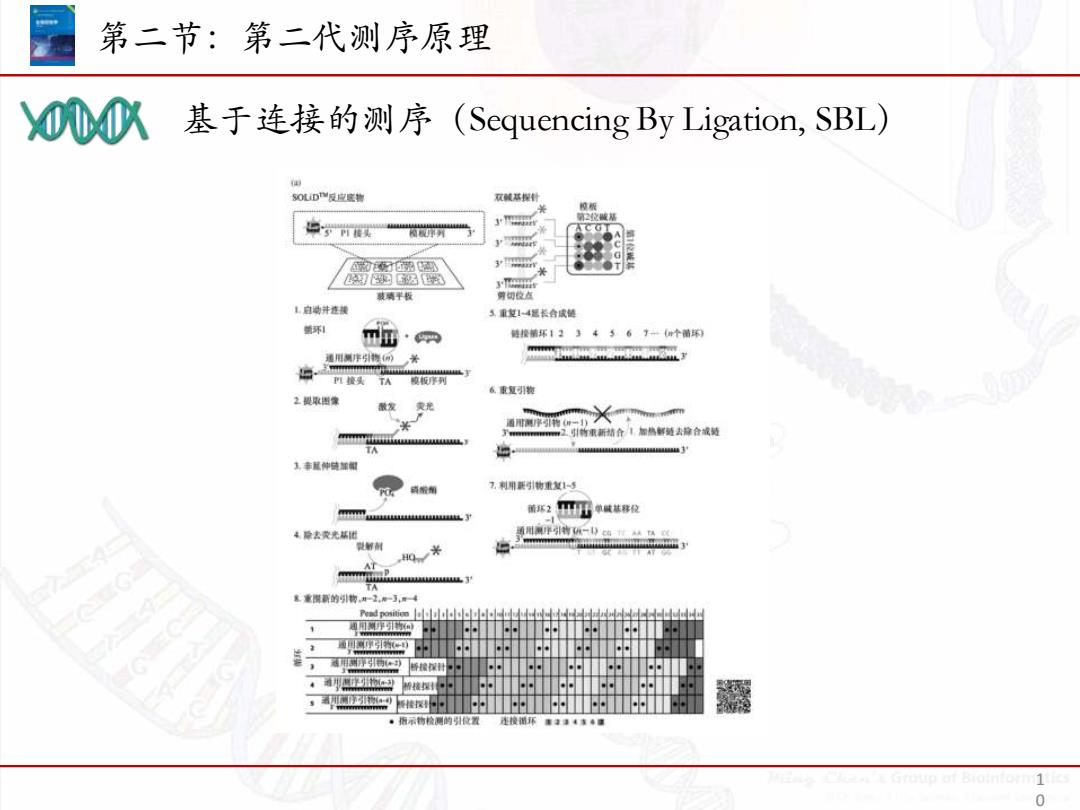
第二节:第二代测序原理 OOM以基于连接的测序(Sequencing By Ligation,SBL) 5SOD型反应底物 双碱基程针 2位 点学m实 塑码 扩 3 吸璃平极 前切位点 1白动并选提 1重复1-4起长合成链 环利 m币·网 链接第环123456子一(你个请环闲 通用测非引梅网)关 酸板序列 板重复可引物 2曼取图象 酸发 更龙 通测序物(一) 2可到的重新结合1图热解鼓去拿合成鼓 TA 1东机钟情加阳 7利用要写1物重复1-3 葡环2山于单碱核移纹 4原去发光某世 复解 0米 AT miR TA 重围新的月物,对-2,一J,年4 Pead position lelslullelslelul 湖事 用序 桥接星 连接环 0
基于连接的测序(Sequencing By Ligation, SBL) 第二节:第二代测序原理 1 0

第二节:第二代测序原理 0例0M以 基于连接的测序(Sequencing By Ligation,SBL) ,单碱基探针(Complete Genomics) b Complete Genomics 。 荧光指示探针序列中指定位 BG动 置的碱基,该碱基的位置可 在生产探针时指定。 ·利用DNA纳米球扩增时插入 Single-base-encoded probes 的接头,使用与接头互补的 A probe with a single known base and degenerate bases hybridizes to a template and is imaged 锚点片段与之在3'端或5'端结 合,再将锚点片段与指示第 一个碱基的探针连接,即可 Reset 检测从锚点开始的第一个碱 After each imaging step,both the probe and anchor are removed 基。成像完成后,除去锚点 Probe with known base at nel 片段和探针 。 重复此步骤,使用第2、第3… 第5个碱基已知的探针,得到 Paired-end sequencing Sequencing is performed for both the left and right sides of 锚点开始的第1-5个碱基 the adapter 1
基于连接的测序(Sequencing By Ligation, SBL) 第二节:第二代测序原理 1 1 • 单碱基探针(Complete Genomics) • 荧光指示探针序列中指定位 置的碱基,该碱基的位置可 在生产探针时指定。 • 利用DNA纳米球扩增时插入 的接头,使用与接头互补的 锚点片段与之在3’端或5’端结 合,再将锚点片段与指示第 一个碱基的探针连接,即可 检测从锚点开始的第一个碱 基。成像完成后,除去锚点 片段和探针 • 重复此步骤,使用第2、第3… 第5个碱基已知的探针,得到 锚点开始的第1-5个碱基