
第一节:Linux操作系统 00八一、Linux简介 Linux源于古老的UNIX系统,其最初的版本是1991年Linus在Minix的基 础上开发的。 Liux最基本的优点就是经济,因为Liux的内核是完全免费的,其用户或厂 商能够自行地搭配其他应用程序的特性。 Liux的发行版有很多,根据软件包管理方式的不同可以分为两类: ()使用RPM方式,包括Red Hat、Fedora、SuSE; (2)使用dpkg方式,包括Debian、Ubuntu等。 选择哪一种Liux发行版并没有太大影响。 2
一、Linux简介 第一节:Linux 操作系统 2 Linux 源于古老的 UNIX 系统,其最初的版本是 1991 年 Linus 在 Minix 的基 础上开发的。 Linux 最基本的优点就是经济,因为 Linux 的内核是完全免费的,其用户或厂 商能够自行地搭配其他应用程序的特性。 Linux 的发行版有很多,根据软件包管理方式的不同可以分为两类: (1)使用 RPM 方式,包括 Red Hat、 Fedora、 SuSE; (2)使用 dpkg 方式,包括 Debian、 Ubuntu 等。 选择哪一种 Linux 发行版并没有太大影响
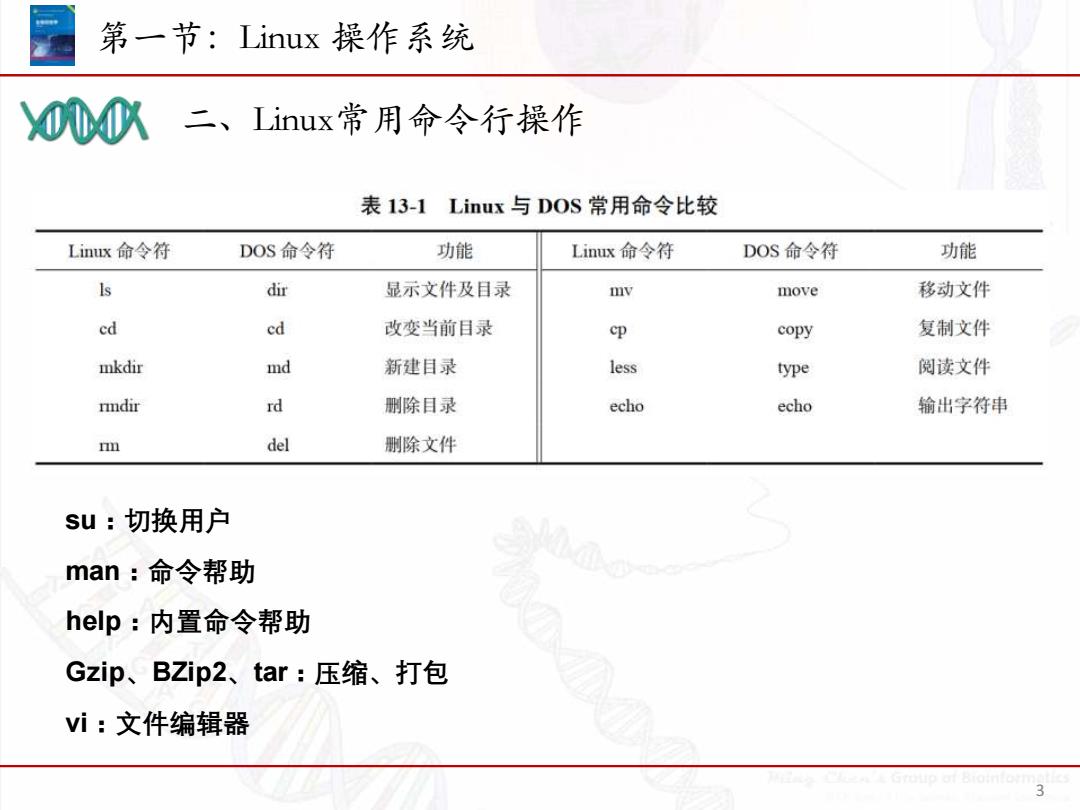
第一节:Linux操作系统 0个队二、Linux常用命令行操作 表13-1 Linux与DOS常用命令比较 Linux命令符 DOS命令符 功能 Linux命令符 DOS命令符 功能 Is dir 显示文件及目录 mv move 移动文件 ed ed 改变当前目录 cp copy 复制文件 mkdi缸 md 新建目录 less type 阅读文件 rmdir rd 删除目录 echo echo 输出字符串 n del 删除文件 su:切换用户 man:命令帮助 help:内置命令帮助 Gzip、BZip2、tar:压缩、打包 vi:文件编辑器 3
二、Linux常用命令行操作 第一节:Linux 操作系统 3 su:切换用户 man:命令帮助 help:内置命令帮助 Gzip、BZip2、tar:压缩、打包 vi:文件编辑器

第一节:Linux操作系统 OO似三、在Linux下使用SAMtools (一)SAMtools的下载与安装 1、先配置好国内镜像源,然后执行apt-get update; 2、下载SAMtools:并解压: wget https://nchc.dI.sourceforge.net/project/samtools/samtools/1.3.1/samtools-1.3.1.tar.bz2 wget https://nchc.dI.sourceforge.net/project/samtools/samtools/1.3.2/htslib-1.3.2.tar.bz2 tar-ixvf samtools-1.3.1.tar.bz2 tar-jxvf htslib-1.3.2.tar.bz2 (二)SAMtools的编译与运行 1、进入文件夹并编辑配置、编译、试运行、安装: cd samtools-1.3.1 ./configure -enable-plugins -enable-libcurl-with-plugin-path=../htslib-1.3.2 make all /samtools make install
三、在 Linux 下使用 SAMtools 第一节:Linux 操作系统 4 (一) SAMtools 的下载与安装 1、先配置好国内镜像源,然后执行 apt-get update; 2、下载SAMtools并解压: wget https://nchc.dl.sourceforge.net/project/samtools/samtools/1.3.1/samtools-1.3.1.tar.bz2 wget https://nchc.dl.sourceforge.net/project/samtools/samtools/1.3.2/htslib-1.3.2.tar.bz2 tar-jxvf samtools-1.3.1.tar.bz2 tar-jxvf htslib-1.3.2.tar.bz2 (二) SAMtools 的编译与运行 1、进入文件夹并编辑配置、编译、试运行、安装: cd samtools-1.3.1 ./configure --enable-plugins --enable-libcurl --with-plugin-path=../htslib-1.3.2 make all ./samtools make install

第一节:inux操作系统 00八 四、使用conda管理软件和安装环境 1.为什么我们需要conda 有必要使用一个工具对不同版本的包和安装环境进行管理,方便切换并减少错误的发生。 2.conda的安装 下载conda: wget https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86 64.sh 依据安装界面提示完成安装: bash Miniconda3-latest-Linux-x86 64.sh 3.conda的使用 conda创建环境:conda create-n env name list of packages 配置一个用于RNA-seq分析的环境,需要安装3.7版本的Python和3.6版本的R: conda create-n RNAseg r-base =3.6 python 3.7 激活RNAseg环境:conda activate RNAseq 退出环境:conda deactivate 在环境配置好后,安装其他软件包:conda install package name 卸载软件包:conda remove package name 5
四、使用conda管理软件和安装环境 第一节:Linux 操作系统 5 1.为什么我们需要conda 有必要使用一个工具对不同版本的包和安装环境进行管理,方便切换并减少错误的发生。 2. conda的安装 下载conda: wget https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh 依据安装界面提示完成安装: bash Miniconda3-latest-Linux-x86_64.sh 3. conda的使用 conda 创建环境:conda create -n env_name list of packages 配置一个用于 RNA-seq 分析的环境,需要安装 3.7 版本的Python 和 3.6 版本的 R : conda create -n RNAseq r-base = 3.6 python = 3.7 激活RNAseq环境:conda activate RNAseq 退出环境:conda deactivate 在环境配置好后,安装其他软件包:conda install package_name 卸载软件包:conda remove package_name

第一节:Linux操作系统 阶似五、容器化技术介绍 目前最流行的Linux:容器解决方案是Docker,它提供了简单易用的容器使用 接口。 Liux容器不是一个完整的操作系统,而是与系统其他部分隔离开的一系列 进程。运行这些进程所需的所有文件都由另一个镜像提供,对于容器里面 的进程来说,它接触到的各种资源都是虚拟的,从而实现了与底层系统的 隔离。 对于生物信息学来说,Docker3主要有两个用途: ①快速部署生物信息学软件; ②开发生物信息学软件时提供测试环境和打包服务。 Docker CE的安装可以参考官方文档 https://docs.docker.com/engine/install. 6
五、容器化技术介绍 第一节:Linux 操作系统 6 目前最流行的Linux容器解决方案是Docker,它提供了简单易用的容器使用 接口。 Linux容器不是一个完整的操作系统,而是与系统其他部分隔离开的一系列 进程。运行这些进程所需的所有文件都由另一个镜像提供,对于容器里面 的进程来说,它接触到的各种资源都是虚拟的,从而实现了与底层系统的 隔离。 对于生物信息学来说, Docker主要有两个用途: ①快速部署生物信息学软件; ②开发生物信息学软件时提供测试环境和打包服务。 Docker CE 的安装可以参考官方文档 https://docs.docker.com/engine/install

第二节:生物信息学中的编程语言 00-、Perl 1、Perl简介 The Perl Programming Language www.perl.org HOME LEARN DOCUMENTATION CPAN COMMUNITY GET INVOLVED DOWNLOAD ABOUT PERL Current Perl version Flexible Powerful 5.18.1-download now That's why we love Perl 5 Find out more Leam Get started DOWNLOAD PERL Documentation Community Perl 5 is a highly capable,feature-rich programming language with over 25 years Events of development.More about why we love Perl... Tip Database interfaces Learning Perl 5 The Perl Community DBIx:Class provides With free online books,over 25,000 Perl has an active world wide an Object Relational extension modules,and a large community with over 300 local Mapper (ORM)to developer community,there are groups,mailing lists and databases (e.g.Oracle, many ways to leam Perl 5. support/discussion websites SQL Server,MySQL, Postgress,Access etc) Documentation Contribute to Perl Sponsor Core documentation,FAQs and Perl is being actively developed. translations. There are many ways to get involved. phyber
一、Perl 第二节:生物信息学中的编程语言 7 1、Perl简介

第二节:生物信息学中的编程语言 O0\-、Perl 2、Perl基础 数据类型: 字符串($string)、数组(@aray)、哈希表(%hash) 正则表达式: 表13-2字符串比较运算符 m// 运算符 返回值 左边和右边相等,则返回真 ne 左边和右边不相等,则返回真 子程序: cmp 左边小于、等于、大于右边,分别返回一1,0,1 Sub SUBNAME t 左边小于右边,则返回真 gt 左边大于右边,则返回真 le 左边小于或等于右边,则返回真 code; ge 左边大于或等于右边,则返回真 8
一、Perl 第二节:生物信息学中的编程语言 8 2、Perl基础 数据类型: 字符串($string)、数组(@array)、哈希表(%hash) 正则表达式: m//; 子程序: Sub SUBNAME { code; }

第二节:生物信息学中的编程语言 00八一、Perl 2、Perl基础 创建包及包函数: Package package1 sub subroutine1 sub subroutine2 调用包: require'/path/package1.pl';package1:subroutine1(; 内置函数: abs、sqrt、exp 文件处理: open(FH,"path../filename"); 模块: LWP:Simple LWP::UserAgent DBI 9
一、Perl 第二节:生物信息学中的编程语言 9 2、Perl基础 创建包及包函数: Package package1;sub subroutine1;sub subroutine2; 调用包: require'/path/package1. pl’;package1::subroutine1(); 内置函数: abs、sqrt、exp 文件处理: open(FH,”path../filename”); 模块: LWP::Simple LWP::UserAgent DBI

第二节:生物信息学中的编程语言 表13-3 BioPerl模块说明 00八一、Perl 应用 模块 1.序列操作 3、BioPerli概况 对序列进行统计 SeqStats.SeqWord 确定限制性内切核酸酶位点 Bio::Restriction 识别氨基酸裂解位点 Sigcleave 多样性序列功能 OddCodes.SeqPattern BioPerl是Perl的扩充, 转换坐标系 Coordinate::Pair,RelSegment 专门用于生物信息 2.搜索相似序列 的工具与函数集。 运行远程BLAST using RemoteBlast.pm 分析BLAST和FASTA结果 Search.SearchIO 分析BLAST结果 BPlite.BPpsilite,and BPbl2seq BioPerl在生物信息学 分析团M结果 HMMER:Results,SearchIO 的使用加速了生物 运行本地BLAST StandAloneBlast 3.序列比对 SimpleAlign 信息学、基因组学 4在基因组DNA寻找基因和其他结构 Genscan.Sim4.Grail,Genemark.ESTScan,MZEF,EPCR 及其他生命科学研 5.开发机器可读性的序列注释 究的发展。 表征序列注释 SeqFeature.RichSeq.Location 表征序列注释 Annotation::Collection 表示大的序列 LargeSeq 表示改变的序列 LiveSeq 表示相关联的序列一突变体,多态性 Allele,SeqDiff 在序列注释中加人特征数据 SeqWithQualiry 序列如红表征—生成和分析 SegIO:game.SegIO::bsml 使用GFF表示序列 Bio:DB:GFF 0
一、Perl 第二节:生物信息学中的编程语言 1 0 3、BioPerl概况 BioPerl是Perl的扩充, 专门用于生物信息 的工具与函数集。 BioPerl在生物信息学 的使用加速了生物 信息学、基因组学 及其他生命科学研 究的发展

第二节:生物信息学中的编程语言 00八一、Pel 3、BioPerl概况 BioPerl继承了Perl强大的正则表示式比对和字符串操作能力。 BioPerl继承了Perl能容错的特点。 续表 应用 模块 6.操作序列簇 Cluster.ClusterIO 7.在BioPe中表示非序列数据:结构。树和图谱 使用3D结构对象并读取PDB文件 Structurel,Structure::IO 树对象和系统发生树 Tree::Tree,TreelO.PAML 图谱对象操作遗传图谱 Map:MapI,MapIO 文献对象搜索文献数据库 Biblio 图形对象以图形代表序列对象 Graphics 1
一、Perl 第二节:生物信息学中的编程语言 1 1 3、BioPerl概况 BioPerl 继承了 Perl 强大的正则表示式比对和字符串操作能力。 BioPerl 继承了 Perl 能容错的特点