
生物大分子波谱学原理 吴季辉 蛋白质核磁脉冲序列分析 阮科 kruan@ustc.edu.cn
蛋白质核磁脉冲序列分析 阮科 kruan@ustc.edu.cn 生物大分子波谱学原理 吴季辉

生物大分子波谐学原理 吴季辉 脉冲序列基本原则 1、平衡态密度算符:Y2,如15N标记蛋白,YHHz+YNN2 2、Delay时期,Hamilton算符为o2+osS2+,S,(两核体系), 多核体系包括所有的化学位移算符和可能的创耦合算符 3、短的硬脉冲,一般只考虑脉冲所起的作用(90,180),忽 略化学位移和演化 4、大多成形脉冲可以近似为90/180脉冲 5、l-lh今?lwM2-lwzS今? BB lyS2 A→AcosB+CsinB
脉冲序列基本原则 1、平衡态密度算符:SgI IZ,如15N标记蛋白,gHHZ+gNNZ 2、Delay时期,Hamilton算符为wI IZ + wSSZ + pJIzSz (两核体系), 多核体系包括所有的化学位移算符和可能的J耦合算符 3、短的硬脉冲,一般只考虑脉冲所起的作用(90,180…),忽 略化学位移和J演化 4、大多成形脉冲可以近似为90/180脉冲 5、I x/y/z—I x/y/z→? Ix/y/z—I x/y/zSx/y/z→? 生物大分子波谱学原理 吴季辉 Ix Iy I z Ix IySz I zSz

生物大分子波谱学原理 吴季辉 脉冲序列分析基本思路 1、 乘积算符 基本单元: 自旋▣波,INEPT,COSY,TOCSY,NOESY t1 t2 t1 2、 相干传递途径 从0开始,-1接收 自由进动时相干阶不变,只有脉冲才能改变相干阶 相干传递途径只画出想要的途径 3、相位循环 脉冲相位变化φ,则相干阶△p经历的相位变化为-△p,即接收相位变化。 若相位循环为360/N,△p为想要的相干阶变化,则△p+nN(n=.-3,-2- 1,0,1,2,3.)也被选择,其它被抑制
脉冲序列分析基本思路 1、乘积算符 基本单元:自旋回波,INEPT, COSY, TOCSY,NOESY 2、相干传递途径 从0开始,-1接收 自由进动时相干阶不变,只有脉冲才能改变相干阶 相干传递途径只画出想要的途径 3、相位循环 脉冲相位变化f,则相干阶Dp经历的相位变化为-fDp,即接收相位变化。 若相位循环为360/N,Dp为想要的相干阶变化,则Dp+nN(n=…-3,-2- 1,0,1,2,3…)也被选择,其它被抑制 生物大分子波谱学原理 吴季辉 t1 t2 t1 -t2

生物大分子波谱学原理 吴季辉 脉冲序列分析基本思路 4、梯度场 可作为z向的脉冲处理 Gz-180-Gz:完善180度脉冲 乘积算符z向后,+强梯度:除去所有的非z向相干阶 梯度选择GzYs-RevINEPT-Gz/Y: 5、间接维正交检波 States,TPPl,States-TPPI
脉冲序列分析基本思路 4、梯度场 可作为z向的脉冲处理 Gz – 180 – Gz:完善180度脉冲 乘积算符z向后,+强梯度:除去所有的非z向相干阶 梯度选择Gz/gS – RevINEPT – Gz/gI: 5、间接维正交检波 States, TPPI,States-TPPI 生物大分子波谱学原理 吴季辉
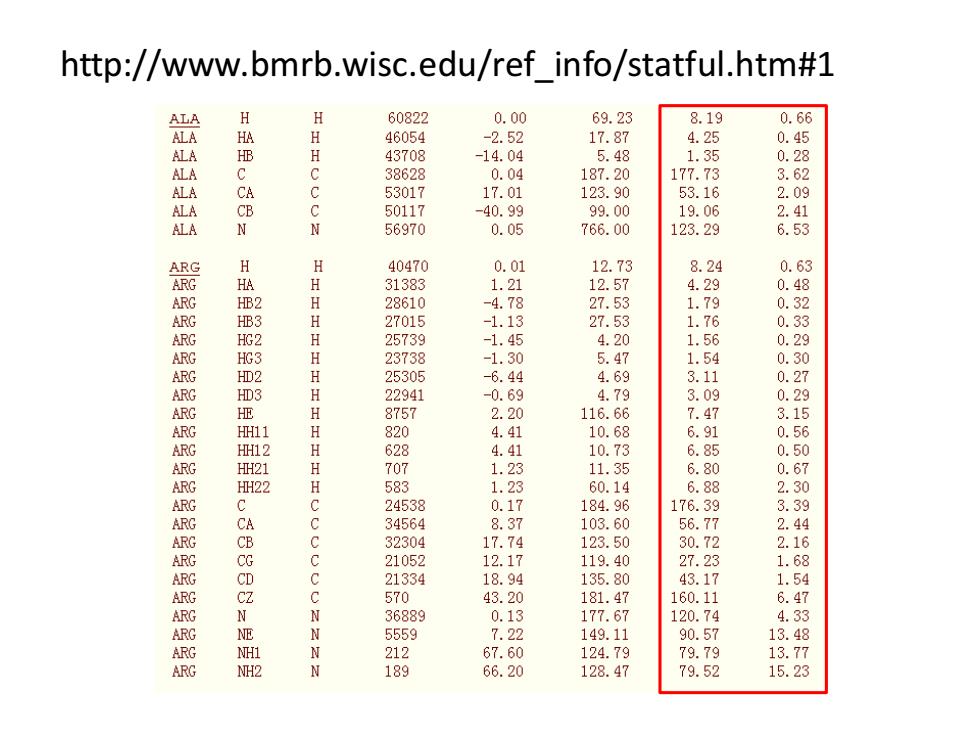
http://www.bmrb.wisc.edu/ref info/statful.htm#1 ALA H 60822 0.00 69.23 8.19 0.66 HA 46054 -2.52 17.87 4.25 0. 43708 -14.04 5.48 1. 0. o88z 38628 0.04 187.20 17 73 3 53017 17.01 123.90 3.1 2 0g 50117 -40.99 99.00 56970 0.05 766.00 123.29 6.53 40470 0.01 12.73 8.24 0.63 31383 12.57 28610 27.53 HB3 27015 2 .53 1. 3 HG2 25739 -1. 45 .2 91966 HG3 23738 -1. 5 7 1 1 0. 0 HD2 25305 .69 . 0.2 22941 0 4. 9 8757 0 11 37 3 HH11 820 4. 68 HH12 HH21 HH22 583 411 354 8 24538 . 4331 01102 5666 3.3 34564 37 103 56. 4 32304 123 5 30. 9915908972337 2. 21052 12 119 1. 68 21334 .94 135. 570 181 47 160 6 36889 177. 120 5559 11 212 189 1489
http://www.bmrb.wisc.edu/ref_info/statful.htm#1
生物大分子波谐学原理 吴季辉 第六章 同核核磁实验方法 COSY型实验 多量子谱 TOCSY NOESY ROESY
第六章 同核核磁实验方法 COSY型实验 多量子谱 TOCSY NOESY ROESY 生物大分子波谱学原理 吴季辉

生物大分子波谱学原理 吴季辉 同核核磁谱 通常所谓同核核磁谱指的是H同核谱,因为一般有机物富 含氢,而H的天然丰度达99.98%,而且H的旋磁比最大,因而 核磁信号最强。其他核的核磁信号的检测相对来讲都比较困难, 尤其是蛋白质中有意义的如13C,15N的天然丰度分别只有1.11% 和0.36%,相对H的检测灵敏度(S)只有0.0159和0.00104,因此要 检测蛋白质中的13C及15N一般要有同位素标记才行,而且还需长 时间累加。cnye(yaB/2 一 般同核谱可研究的蛋白质分子量的上限在1万左右,当然还要 求蛋白质在溶液中有足够的溶解度,化学位移的分布比较分散。 蛋白质中质子的化学位移基本上是random coil的化学位移加上不 同构象的影响。如B折叠多的蛋白质同α螺旋多的蛋白质相比, 化学位移更为分散,谱峰也更容易解析;同种氨基酸残基多的 蛋白质,即使分子量较小,由于化学位移可能会比较密集,谱 峰解析可能比稍大一些的蛋白质还要困难
同核核磁谱 生物大分子波谱学原理 吴季辉

生物大分子波谐学原理 吴季辉 同核核磁谱 在记录二维谱或三维谱等之前均需要记录一维氢谱,通常蛋 白质溶于水或适当的缓冲液,加5-10%的D,O作为锁场介质。 维氢谱可用于检查样品是否有足够浓度,通常16一32次采样要 能得到足够信噪比的谱图,甲基由于快速旋转呈现窄峰,而酰 胺质子由于化学交换及14N的标量弛豫呈现宽峰; 维氢谱还可用于检查样品的共振峰宽是否合理,蛋白质在 高浓度下容易聚集从而增宽谱线,严重时甚至H谱上看不到明 显的峰,样品在这种条件下显然无法进行核磁研究;一维氢谱 还可用于判别样品的化学位移分布情况,可判别样品是否变性, 同时也对研究工作的难度提供一些预测。 蛋白质的一维氢谱一般不应该有非常尖的峰,若出现这种峰 往往是残留的小分子杂质,当然蛋白质的末端及1oop区由于分子 运动较快也会出现一些较尖的峰
同核核磁谱 在记录二维谱或三维谱等之前均需要记录一维氢谱,通常蛋 白质溶于水或适当的缓冲液,加5-10%的D2O作为锁场介质。一 维氢谱可用于检查样品是否有足够浓度,通常16-32次采样要 能得到足够信噪比的谱图,甲基由于快速旋转呈现窄峰,而酰 胺质子由于化学交换及14N的标量弛豫呈现宽峰; 一维氢谱还可用于检查样品的共振峰宽是否合理,蛋白质在 高浓度下容易聚集从而增宽谱线,严重时甚至1H谱上看不到明 显的峰,样品在这种条件下显然无法进行核磁研究;一维氢谱 还可用于判别样品的化学位移分布情况,可判别样品是否变性, 同时也对研究工作的难度提供一些预测。 蛋白质的一维氢谱一般不应该有非常尖的峰,若出现这种峰, 往往是残留的小分子杂质,当然蛋白质的末端及loop区由于分子 运动较快也会出现一些较尖的峰。 生物大分子波谱学原理 吴季辉
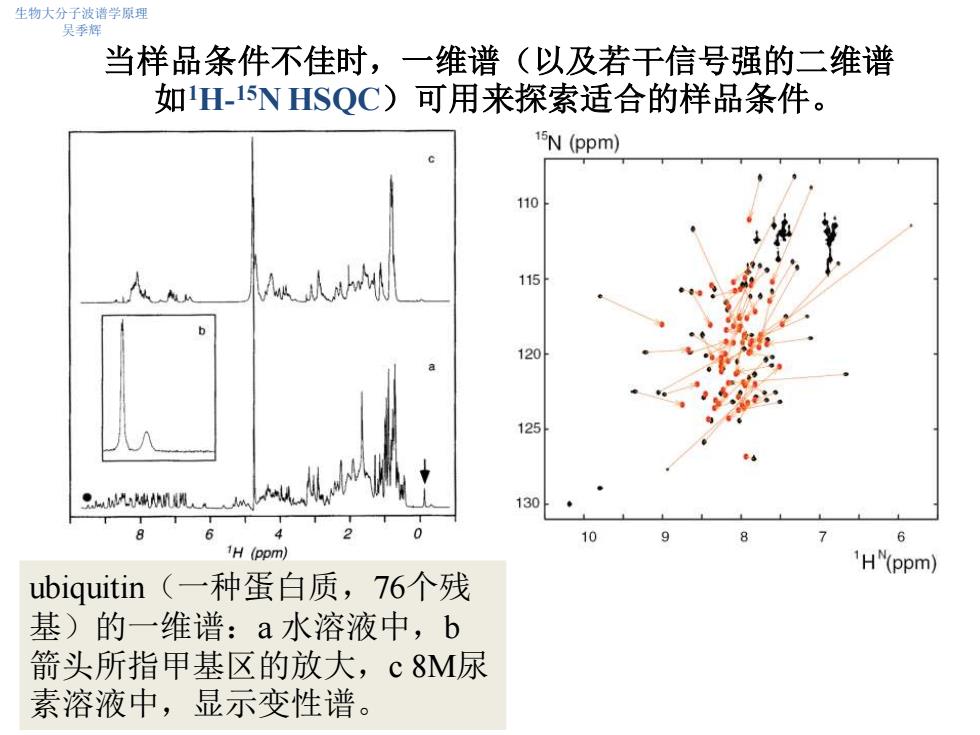
生物大分子波谱学原理 吴季辉 当样品条件不佳时,一维谱(以及若干信号强的二维谱 如H-15 N HSOC)可用来探索适合的样品条件。 15N (ppm) 110 115 120 125 130 9 6 10 7 6 1H (ppm) H"(ppm) ubiquitin(一种蛋白质,76个残 基)的一维谱:a水溶液中,b 箭头所指甲基区的放大,c8M尿 素溶液中,显示变性谱
当样品条件不佳时,一维谱(以及若干信号强的二维谱 如1H- 15N HSQC)可用来探索适合的样品条件。 生物大分子波谱学原理 吴季辉 ubiquitin(一种蛋白质,76个残 基)的一维谱:a 水溶液中,b 箭头所指甲基区的放大,c 8M尿 素溶液中,显示变性谱

生物大分子波谱学原理 吴季辉 A Strategy for Elucidating the Structure of Peptides/Proteins Using 2D Proton Homonuclear NMR Experiments Obtain primary sequence of peptide/protein Collect 2D NMR spectra H-H DQF-COSY H-IH TOCSY,short and long mixing times H-H NOESY,short and long mixing times Assign spin systems use TOCSY and DQF-COSY spectra and,if necessary,NOESY spectra Assign sequential amino acids use NOESY HN-HN and HN-Ha regions Obtain structural information from NMR spectra NOESY cross peak intensities H班 DQF-COSY 3JHN-Ha φ PE COSY 3JHa-H邱 Z H20/D20 exch. HN exchange rates h-bonds Generate structure Use software that "folds"primary sequence on basis of structural info Visualize structures using molecular graphics Refine structure,if needed,with additional data
生物大分子波谱学原理 吴季辉