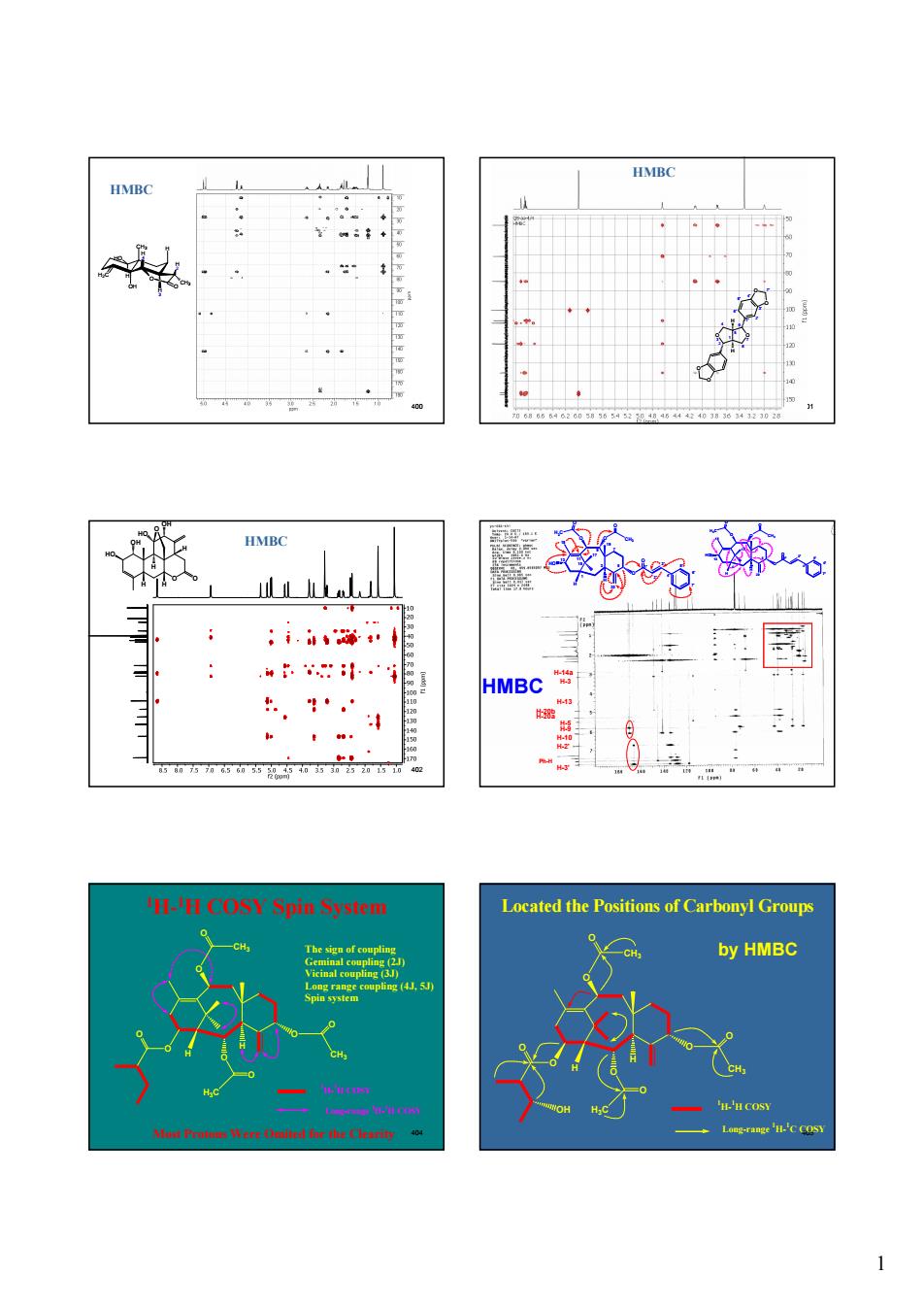
HMBC L HMBC F:l H-H COSY Spin System Located the Positions of Carbonyl Groups by HMBC CH CH H.C
1 400 H2C CH3 OH H O O H 4 H 3 H HO H 2 CH3 HMBC 401 2 O 3 1 4 5 8 O 7 6 1' 6' 2' 5' 3' 4' O O 7' O O H H HMBC 402 O O HO O OH OH HO H H H H HMBC 403 H-10 H-9 H-5 H-20a H-20b H-3' Ph-H H-2' H-13 H-3 H-14a O O H H O HO O 1 3 5 7 9 13 11 15 17 18 19 20 1' 2' 3' CH3 O H3C O 4' 5' 6' 7' O O H H O HO O 1 13 5 17 18 20 1' 3' CH3 O H3C O 4' 5' 6' 7' HMBC Mo 404 st Protons Were Omited for the Clearity 1H-1H COSY Spin System O O O O O H H 1 H- 1 H COSY Long-range 1 H- 1 H COSY CH3 O CH3 O H3C O The sign of coupling Geminal coupling (2J) Vicinal coupling (3J) Long range coupling (4J, 5J) Spin system 405 Located the Positions of Carbonyl Groups by HMBC O O O O O H H 1 H- 1 H COSY Long-range 1 H- 1 C COSY CH3 O CH3 O H3C O OH
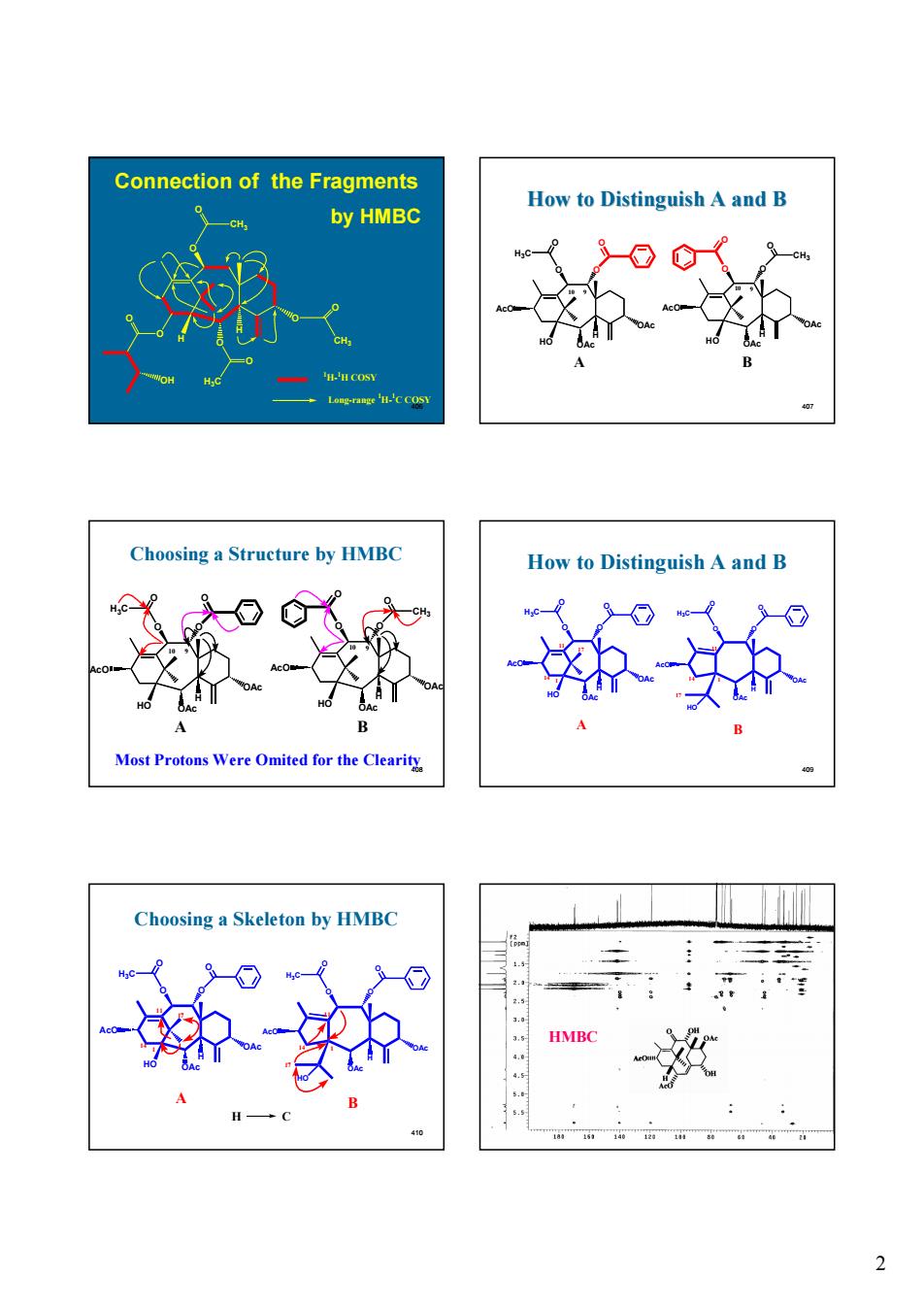
Connection of the Fragments How to Distinguish A and B -CH. by HMBC HHCOSY →CCo四 Choosing a Structure by HMBC How to Distinguish A and B 森疯 领流 Most Protons Were Omited for the Clearity Choosing a Skeleton by HMBC 滋如 洲
2 406 Connection of the Fragments by HMBC O O O O O H H 1 H- 1 H COSY Long-range 1 H- 1 C COSY CH3 O CH3 O H3C O OH 407 OAc OAc O H HO AcO O CH3 O O OAc OAc O H HO AcO O H3C O O A B 10 9 10 9 How to Distinguish A and B 408 Choosing a Structure by HMBC Most Protons Were Omited for the Clearity OAc OAc O H HO AcO O CH3 O O OAc OAc O H HO AcO O H3C O O A B 10 9 10 9 409 OAc OAc O H HO AcO O H3C O O OAc OAc O H AcO O H3C O O HO A B 1 11 14 1 11 14 17 17 How to Distinguish A and B 410 Choosing a Skeleton by HMBC H C OAc OAc O H HO AcO O H3C O O OAc OAc O H AcO O H3C O O HO A B 1 11 14 1 11 14 17 17 411 HMBC
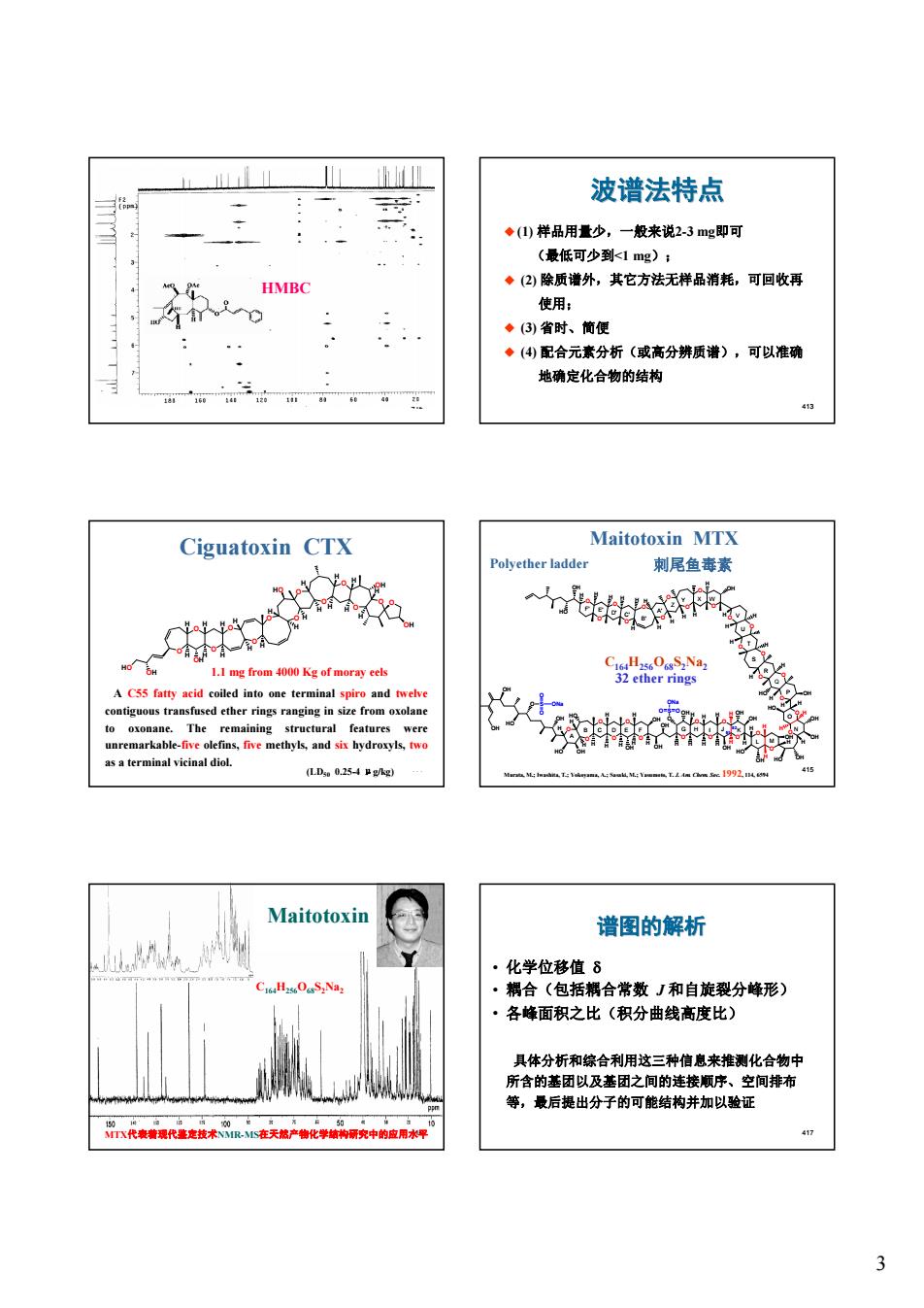
h 波谱法特点 ◆)样品用量少,一般来说2-3m吧即可 (最低可少到k1mg); HMBC ◆(2)除质谱外,其它方法无样品消耗,可回收再 使用: ·)省时、简便 ·(④配合元素分析(或高分辨质谱),可以准确 地确定化合物的结构 Ciguatoxin CTX Maitotoxin MTX Polyether ladder 刺尾鱼毒素 Maitotoxin 谱图的解析 ·化学位移值8 .Na ·耦合(包括耦合常数J和自旋裂分峰形) ·各峰面积之比(积分曲线高度比) 组体公折有偏合利用三纯信息来推化 所含的基团以及基团之间的连接顺序 等,最后提出分子的可能结构并加以验证 在天然产物化学中的用水甲
3 412 HMBC 413 波谱法特点 u(1) 样品用量少,一般来说2-3 mg即可 (最低可少到<1 mg); u (2) 除质谱外,其它方法无样品消耗,可回收再 使用; u (3) 省时、简便 u (4) 配合元素分析(或高分辨质谱),可以准确 地确定化合物的结构 414 Ciguatoxin CTX A C55 fatty acid coiled into one terminal spiro and twelve contiguous transfused ether rings ranging in size from oxolane to oxonane. The remaining structural features were unremarkable-five olefins, five methyls, and six hydroxyls, two as a terminal vicinal diol. O O O O O O O O O O O O O H H H H H HO H H H H OH H OH H H H H H H H H H H OH HO OH (LD50 0.25-4 μg/kg) 1.1 mg from 4000 Kg of moray eels 415 Maitotoxin MTX 刺尾鱼毒素 Murata, M.; Iwashita, T.; Yokoyama, A.; Sasaki, M.; Yasumoto, T. J. Am. Chem. Soc. 1992, 114, 6594 C164H256O68S2Na2 32 ether rings Polyether ladder O O O O O O O O O O O O O OH O O O OH O O O O HO OH O OH S O O ONa HO OH OH OH OH OH O S O ONa HO OH O H H H H H H H HO H H H H H H H H OH OHH H H H H H OH H OH HO OH H H OH H H OH OH H H HO H H H H H O O O O O O O OH H H H H H H H H H HO H A B C D E F G H I J K L M N P Q R S T U V Y X W 51 52 O O O O O HO OH H H H H H H O Z A' B' D C' ' F E' ' 416 Maitotoxin MTX代表着现代鉴定技术NMR-MS在天然产物化学结构研究中的应用水平 C164H256O68S2Na2 417 谱图的解析 • 化学位移值 d • 耦合(包括耦合常数 J 和自旋裂分峰形) • 各峰面积之比(积分曲线高度比) 具体分析和综合利用这三种信息来推测化合物中 所含的基团以及基团之间的连接顺序、空间排布 等,最后提出分子的可能结构并加以验证

H-NMR谱图解析时的注意事项 H-NMR谱图解析时的注意事项 ◆注意区分杂质峰、溶剂峰和转边带等非样品 浙泼氢在清液中会发生交换反应当交换反应速度得 ◆注意分子中活浚氢产生的信号,OH、NH、SH等 活泼氢的核磁共振信号比较特殊,在解析时应注 意。 ◆活泼氢多数能形成氢健,其化半位移值不圆 能与它蛋烈储合而使交换速度大大降低,此时可以来到 定,随测定条件在一定区城内变动: 根据分饰的个数可以区分伯、仲、叔, H-NMR谱图解析时的注意事项 核磁共振谱图综合解析 识别氢谱与碳中的剂峰与杂质峰。 品浓度、测量度等,吸收峰位发生变化的是活 氢:另一种方法是利用意水交换反应。具体敏法为,先 碳原子的级最。H1Q 以及活氧等。 核磁共振谱图综合解析 综合解析谱图的一般程序 ,确定拼的度 2,确定分于式。确定分子式的方法有。 ()质请法测定来知物的分子量。结合元素分析结果 可以计第出化合物的分子 到春相 分辨质请中的分子高于饰和+1,+2同位素蜂的 相对丰度比,查Beynon表来推算分子式
4 418 1H-NMR 谱图解析时的注意事项 u 注意区分杂质峰、溶剂峰和旋转边带等非样品 峰。 u 注意分子中活泼氢产生的信号。OH、NH、SH 等 活泼氢的核磁共振信号比较特殊,在解析时应注 意。 u活泼氢多数能形成氢键,其化学位移值不固 定,随测定条件在一定区域内变动; 419 1H-NMR 谱图解析时的注意事项 活泼氢在溶液中会发生交换反应。当交换反应速度很 快时,体系中存在的多种活泼氢(如样品中既含羧基,又含 胺基、羟基或者含有几个不同化学环境的羟基,样品和溶 剂中含活泼氢等)在核磁共振谱图上只显示一个平均的活泼 氢信号,而且它们与相邻含氢基团的谱峰不再产生耦合裂 分现象。如果使用二甲基亚砜(CH3)2SO 为溶剂,因羟基 能与它强烈缔合而使交换速度大大降低,此时可以观察到 样品中不同羟基的信号以及羟基与邻碳上的质子耦合裂分 的信息。根据裂分峰的个数可以区分伯、仲、叔醇。 420 1H-NMR 谱图解析时的注意事项 • 正是因为活泼氢有以上特点,通过实验可以将它们与其 他氢的信号区别开来。一种方法是改变实验条件,如样 品浓度、测量温度等,吸收峰位置发生变化的就是活泼 氢;另一种方法是利用重水交换反应。具体做法为,先 测绘正常的氢谱,然后在样品溶液中滴加 1-2 滴重水并 振荡,再测绘一氢谱。由于活泼氢与重水中的氘快速交 换,原来由活泼氢产生的吸收峰消失。 421 核磁共振谱图综合解析 • 识别氢谱与碳谱中的溶剂峰与杂质峰。 • 初步分析谱图,找出特征峰并确定各谱线的大致归属。 – 分析一维1H 谱,根据谱图中化学位移值、耦合常数 值、峰形和峰面积找出一些特征峰,获得一些最明显的 结论。 – 对照13C 质子噪声去偶谱以及各个 DEPT 碳谱,确定各 碳原子的级数。HMQC, HSQC – 按照化学位移分区的规律,大致确定各谱线所属的区 域,如在饱和区还是在不饱和区,是否含杂原子、羰基 以及活泼氢等。 422 核磁共振谱图综合解析 • 借助二维核磁共振谱对图谱作进一步的指认。 – 解析1H -1H COSY 谱,从一维谱中已经确定的氢谱线出 发找到与之相关的其它谱线。 – 解析13C -1H COSY(或 HMQC、HSQC)谱,同样从 已知的氢谱线出发找到各相关的碳谱线,以此推断出这 些碳谱线的归属。 – 解析13C -1H 远程相关谱(COLOC 或 HMBC),从已 确定的碳谱线出发,找到与之相关的各氢谱线或从已知 的氢谱线出发找到各相关的碳谱线,由此完成对一些未 知谱线的指认。在二维谱中由于一些相关峰的强度较 弱,在实验中常常未被检测到,另外在图谱中还常常会 出现假峰,这些在二维谱的解析中应特别注意。 423 1.确定样品的纯度。 2.确定分子式。确定分子式的方法有: (1) 质谱法测定未知物的分子量,结合元素分析结果 可以计算出化合物的分子式。 (2) 根据高分辨质谱给出的分子离子的精确质量数, 查Beynon表或Lederberg表计算得出,也可根据低 分辨质谱中的分子离子峰和M+1,M+2同位素峰的 相对丰度比,查Beynon表来推算分子式。 综合解析谱图的一般程序

3.计算化合物的不饱和度。计算不饱和度对到 3)由核磁共损CNR桌带去偶誉的修数和峰的强度估 算碳原子数,结合分子重,判断分子对称性。由偏共 断化合物类型很有必要。如不饱和度在1一3之 振去得请减DEPT请得到与各碳相违的氢原子最,由 间,分子中可能含有C-C、C=0或环,如不饱和 度≥4,分子中可能有苯环. 物分于式 4.结构单元的确定! 5。可能结构式的推导。 含有N、S、X《肉)等) 子是否含氧,可从红外光谱含氧团(OH、C-0 C.0)的吸收峰判条。 说明结论都很可能是误的, 小 结 Comparing UV,IR,MS and NMR 1 Cost:NMR>MS>IR>UV ·MR信号组的位置告诉我们分子中每一种等价质子的 2.Ease of Usage:UV>IR>NMR>MS 化学环, 强。峰面积积分比时应于分子中名修价质子的表 3.Scope and Information Provided NMR>MS>IR>UV 。裂分:裂分的峰最与相邻的另一组等价质子的最目。 4.Theoretical backero ·根擦偶合常数可判别哪调组等价质子为相邻关系。 UV-IR ·不-氢交换:判别活淀氢的常用方法, Sensitivity MS>UV>IR >H-NMR C-NMR 色含图三 品近血面向宿接 8 3D Struct Evaluate Structure By Theory
5 424 (3) 由核磁共振13C-NMR宽带去偶谱的峰数和峰的强度估 算碳原子数,结合分子量,判断分子对称性。由偏共 振去偶谱或DEPT谱得到与各碳相连的氢原子数,由 1H-NMR的积分曲线高度比也可认别各基团含氢数目 比,确定化合物分子式。 可通过元素定性分析确定分子中是否含有杂原子,如 含有N、S、X(卤素)等元素,还需测定其含量。分 子是否含氧,可从红外光谱含氧基团(OH、C=O、 C-O等)的吸收峰判断。 425 3.计算化合物的不饱和度。计算不饱和度对判 断化合物类型很有必要。如不饱和度在1~3之 间,分子中可能含有C=C、C=O或环,如不饱和 度≥4,分子中可能有苯环。 4.结构单元的确定。 5.可能结构式的推导。 6.化合物的确定。 7. 验证:推出的结构和任何一个数据有矛盾 说明结论都很可能是错误的。 426 小 结 u 化学位移:NMR 信号组的数目代表了分子中的等价质 子的数目。 u NMR 信号组的位置告诉我们分子中每一种等价质子的 化学环境。 u 峰强度:峰面积积分比对应于分子中各等价质子的数目 比。 u 裂分:裂分的峰数与相邻的另一组等价质子的数目。 u 根据偶合常数可判别哪两组等价质子为相邻关系。 u 氘-氢交换:判别活泼氢的常用方法。 427 1. Cost: NMR > MS > IR > UV 2. Ease of Usage: UV > IR > NMR > MS 3. Scope and Information Provided: NMR > MS > IR > UV 4. Theoretical background: NMR >> MS > UV = IR 5. Sensitivity MS > UV > IR > 1H-NMR > 13C-NMR Comparing UV, IR, MS and NMR 428 1 2 3 (Atom connect) 4 Assemble Substru. 5 Cluster Substru 6 Total Strut 7 Re-Eval Structs 8 3D Struct. 9 Evaluate Structure By Theory 429
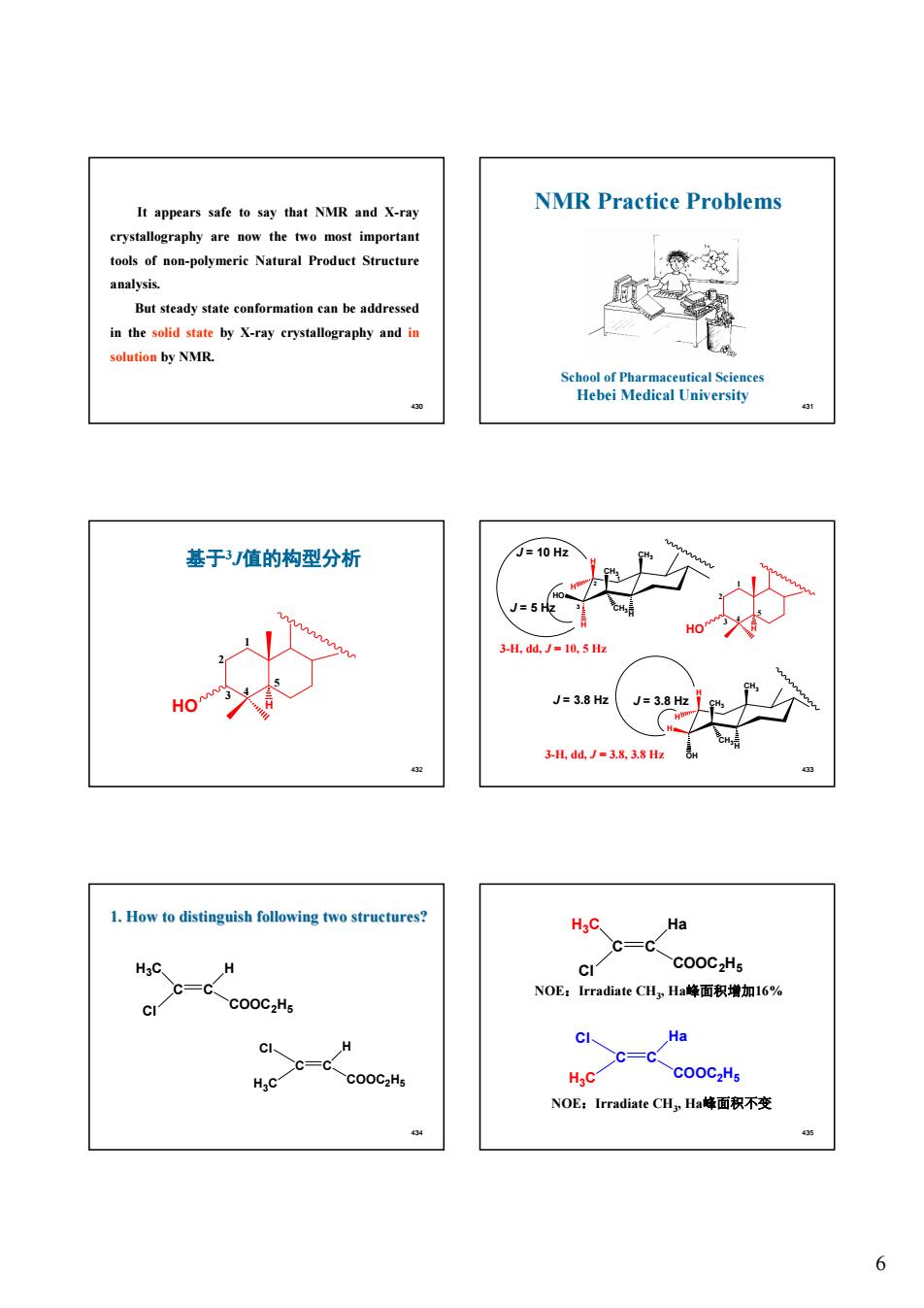
It appears safe to say that NMR and X-ray NMR Practice Problems meric Natural Product Stru But steady state conformation can be addressed in the solid state by X-ray crystallography and in solution by NMR. 基于3J值的构型分析 3-H,dd.J-10.5Hz J=3.8 分 1.How to distinguish following two structures? Ha c-c CI COOC2Hs COOCzHs NOE:Irradiate CH,Ha峰面积增加16% CC=C c-c Ha Hac NOE:Irradiate CH Ha峰面积不变 6
6 430 It appears safe to say that NMR and X-ray crystallography are now the two most important tools of non-polymeric Natural Product Structure analysis. But steady state conformation can be addressed in the solid state by X-ray crystallography and in solution by NMR. 431 NMR Practice Problems School of Pharmaceutical Sciences Hebei Medical University 432 HO H 1 2 3 4 5 基于3J值的构型分析 433 CH3 H H H CH3 H HO CH3 CH3 H OH H CH3 H H CH3 1 2 3 J = 10 Hz J = 5 Hz J = 3.8 Hz J = 3.8 Hz HO H 3-H, dd, J = 10, 5 Hz 3-H, dd, J = 3.8, 3.8 Hz 1 2 3 4 5 434 C C H COOC2H5 H3C Cl C C H H COOC2H5 3C Cl 1. How to distinguish following two structures? 435 C C Ha COOC2H5 H3C Cl NOE:Irradiate CH3, Ha峰面积增加16% C C Ha H COOC2H5 3C Cl NOE:Irradiate CH3, Ha峰面积不变
sing a Structure by NOE 女女 平 安 3.C,H,O,的HNMR谱如下,推导其结构 H 0 4.4dd1 )116630 L IH 4.C,H,O的HNMR谱如下,推导其结构 H.dd 28叫 3H.s 4
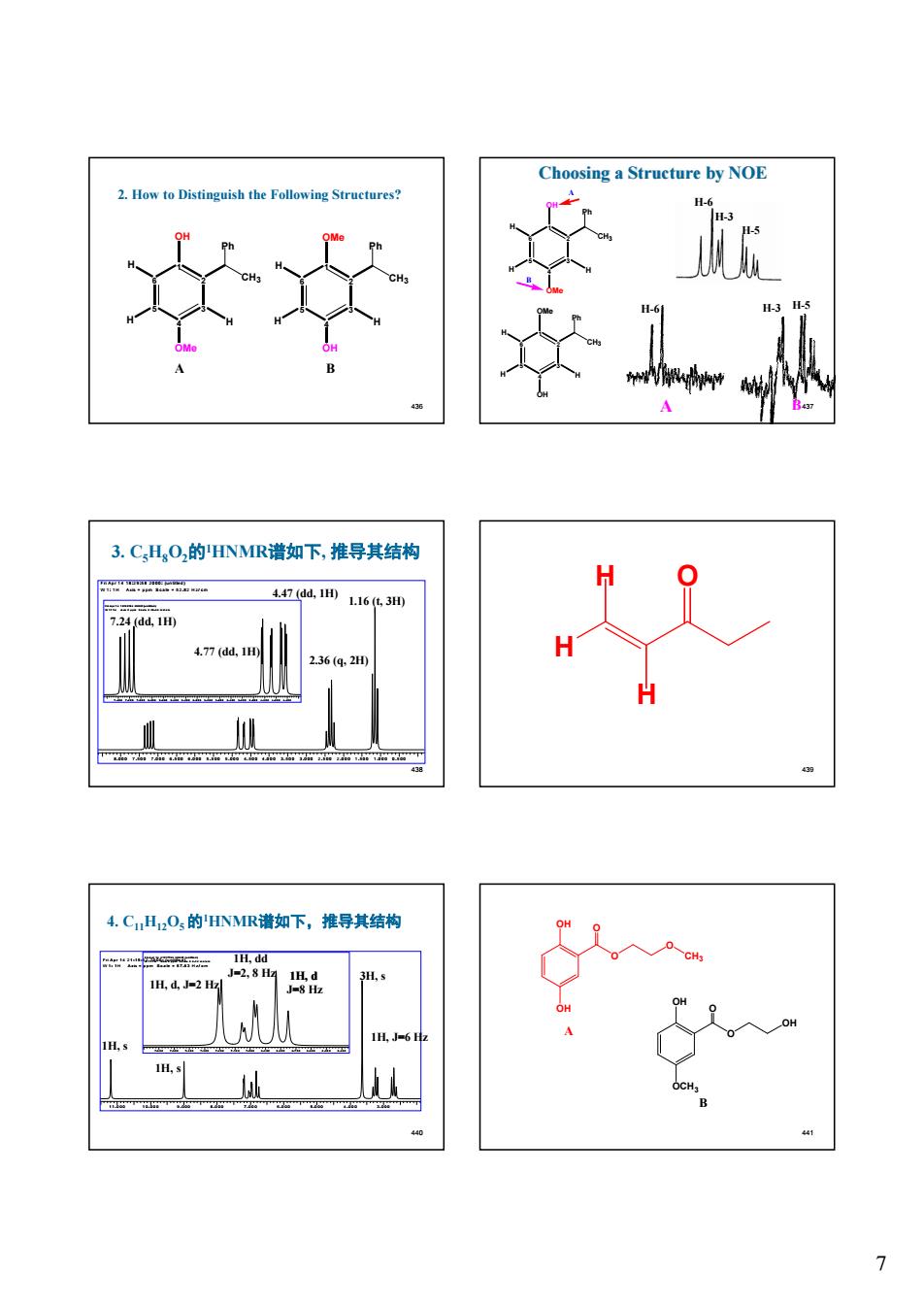
7 436 6 5 4 3 2 1 Ph CH3 OMe OH H H H A 6 5 4 3 2 1 Ph CH3 OH OMe H H H B 2. How to Distinguish the Following Structures? 437 Choosing a Structure by NOE H-6 H-3 H-5 H H-5 H-6 -3 A B 6 5 4 3 2 1 Ph CH3 OH OMe H H H 6 5 4 3 2 1 Ph CH3 OMe OH H H H A B 438 3. C5H8O2的1HNMR谱如下, 推导其结构 Fri A p r 1 4 1 8 :2 9 :5 8 2 0 0 0 : (u n title d ) W 1 : 1 H A xis = p p m S c a le = 5 2 .8 2 H z/ c m 8 .0 0 0 7 .5 0 0 7 .0 0 0 6 .5 0 0 6 .0 0 0 5 .5 0 0 5 .0 0 0 4 .5 0 0 4 .0 0 0 3 .5 0 0 3 .0 0 0 2 .5 0 0 2 .0 0 0 1 .5 0 0 1 .0 0 0 0 .5 0 0 Fri A p r 1 4 1 8 :3 2 :0 4 2 0 0 0 : (u n title d ) W 1 : 1 H A xis = p p m Sc a le = 2 0 .9 9 H z/ c m 7 .4 0 0 7 .2 0 0 7 .0 0 0 6 .8 0 0 6 .6 0 0 6 .4 0 0 6 .2 0 0 6 .0 0 0 5 .8 0 0 5 .6 0 0 5 .4 0 0 5 .2 0 0 5 .0 0 0 4 .8 0 0 4 .6 0 0 4 .4 0 0 7.24 (dd, 1H) 4.47 (dd, 1H) 1.16 (t, 3H) 2.36 (q, 2H) 4.77 (dd, 1H) 439 H H O H 440 4. C11H12O5 的1HNMR谱如下,推导其结构 Fri A p r 1 4 2 1 :1 5 :1 2 2 0 0 0 : (u n title d ) W 1 : 1 H A xis = p p m S c a le = 5 7 .8 3 H z/ c m 1 1 .0 0 0 1 0 .0 0 0 9 .0 0 0 8 .0 0 0 7 .0 0 0 6 .0 0 0 5 .0 0 0 4 .0 0 0 3 .0 0 0 Fri A p r 1 4 2 1 : 1 7 :3 9 2 0 0 0 : (u n t itle d ) W 1 : 1 H A xis = p p m S c a le = 8 .1 2 H z/ c m 7 .6 0 0 7 .5 0 0 7 . 4 0 0 7 .3 0 0 7 .2 0 0 7 . 1 0 0 7 .0 0 0 6 .9 0 0 6 .8 0 0 6 .7 0 0 6 .6 0 0 6 .5 0 0 6 .4 0 0 1H, d, J=2 Hz J=2, 8 Hz J=8 Hz 1H, J=6 Hz 1H, d 3H, s 1H, dd 1H, s 1H, s 441 OH OH O O O CH3 OH OCH3 O O OH A B

5.How to distinguish A and B 6.How to distinguish A and B Choosing a structure by HMBC 7.How to distinguish A and B 森有 流 Most Protons Were Omited for the Clearity Choosing a skeleton by HMBC 从某一豆科植物中分离出一化合物,分子式为 Cw,0,m192.04.根据下列信息 准断其可能的 结构 H-NMR H 滋如 IH
8 442 OGlc OCH3 A O OCH3 OGlc B O 1 2 3 4 5 6 2 5 6 5. How to distinguish A and B 443 OAc OAc O H HO AcO O CH3 O O OAc OAc O H HO AcO O H3C O O A B 10 9 10 9 6. How to distinguish A and B 444 Choosing a structure by HMBC Most Protons Were Omited for the Clearity OAc OAc O H HO AcO O CH3 O O OAc OAc O H HO AcO O H3C O O A B 10 9 10 9 445 OAc OAc O H HO AcO O H3C O O OAc OAc O H AcO O H3C O O HO A B 1 11 14 1 11 14 17 17 7. How to distinguish A and B 446 Choosing a skeleton by HMBC H C OAc OAc O H HO AcO O H3C O O OAc OAc O H AcO O H3C O O HO A B 1 11 14 1 11 14 17 17 447 从某一豆科植物中分离出一化合物, 分子式为 C10H8O4 m/z: 192.04. 根据下列信息, 推断其可能的 结构。 J=10 Hz J=10 Hz 3H 1H 1H 1H 1H 7 6 5 4 3 1H-NMR

H-H COSY 13C-NMR C1oHsO 448 49 HMOC ‖ 450 8 9
9 448 160 140 110 100 60 13C-NMR C10H8O4 449 1H-1H COSY 450 HMQC 8 6 4