
用曲 Deep-Kcr:accurate detection of lysine crotonylation sites using deep learning method /966 可▣ Center for Information Biology,UESTC,China,Chengdu 司第 Hao Lin INDING 2020-09- Group
Center for Information Biology, UESTC, China, Chengdu Hao Lin 2020-09- Deep-Kcr: accurate detection of lysine crotonylation sites using deep learning method
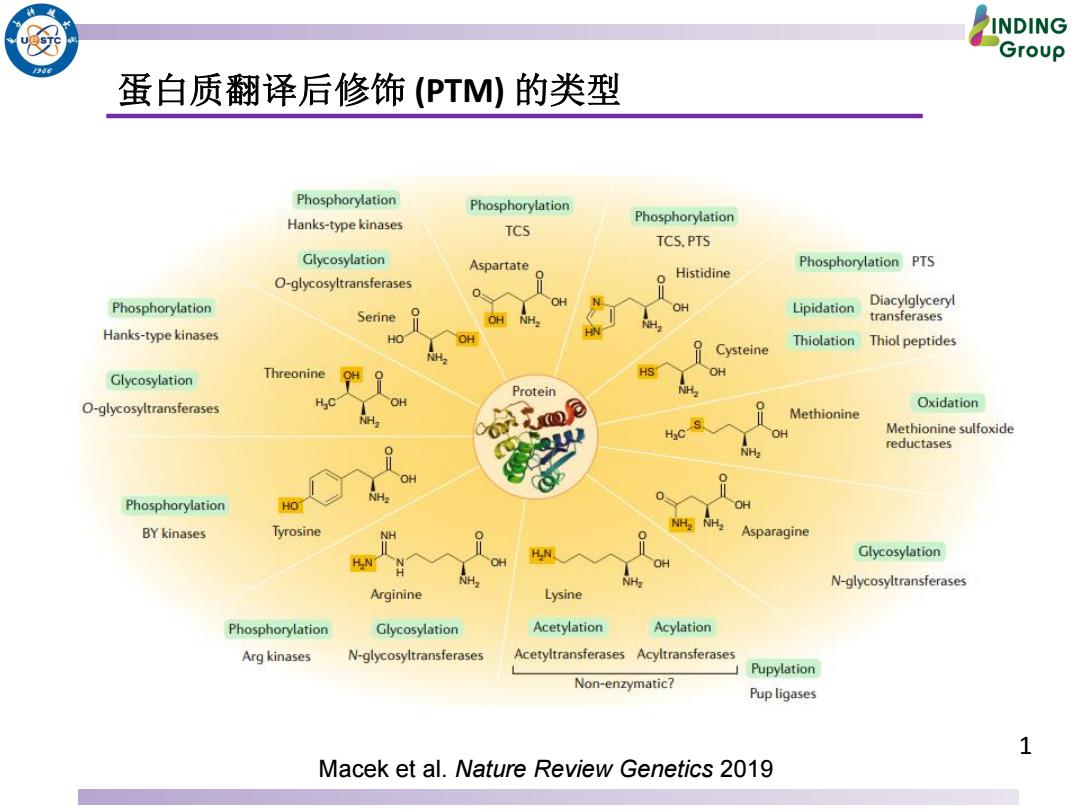
INDING Group 956 蛋白质翻译后修饰(PTM)的类型 Phosphorylation Phosphorylation Hanks-type kinases Phosphorylation TCS TCS.PTS Glycosylation Aspartate Phosphorylation PTS Histidine O-glycosyltransferases Phosphorylation OH Serine Lipidation Diacylglyceryl OH transferases Hanks-type kinases HO Cysteine Thiolation Thiol peptides HS OH Glycosylation Threonine Protein O-glycosyltransferases Oxidation Methionine OH Methionine sulfoxide reductases NH. Phosphorylation NH NH BY kinases Tyrosine Asparagine N Glycosylation N-glycosyltransferases Arginine Lysine Phosphorylation Glycosylation Acetylation Acylation Arg kinases N-glycosyltransferases Acetyltransferases Acyltransferases Pupylation Non-enzymatic? Pup ligases 1 Macek et al.Nature Review Genetics 2019
1 蛋白质翻译后修饰 (PTM) 的类型 Macek et al. Nature Review Genetics 2019

INDING Group 9s6 组蛋白巴豆酰化(Kcr)的发生过程 DNA转录为mRNA并出核 mRNA d Ribosomal 核糖体将mRNA翻译成蛋白质 subunits Amino acids Peptide chain lysine RNA 蛋白质穿过内质网、高尔基体 包装 Crotonyl lysine ribosome 进入囊泡 巴豆酰基 Kcr的翻译后修饰 囊泡中,蛋白质被共价修饰 2
2 组蛋白巴豆酰化(Kcr)的发生过程 Kcr的翻译后修饰 DNA转录为mRNA并出核 核糖体将mRNA翻译成蛋白质 蛋白质穿过内质网、高尔基体 进入囊泡 包装 巴豆酰基 囊泡中,蛋白质被共价修饰

24N9 Group Kcr功能:调控精子发生的成熟过程 Cell Resource Identification of 67 Histone Marks and Histone Lysine Crotonylation as a New Type of Histone Modification 中国科学院生物 图 Minjia Tan.Hao Luo.Sangkyu Lee Fulai Jin,Jeor and YingingYen hu Joanina WocYang Ye. Nish Ben May Department of Canoor Research,The University of Chicago,Chicago,IL 80637,USA udwig institute for Cancer Research and Department af Celluar and Molecular Medicine,Universty of Caitomia San Diego School of 4 ledcine,9500 Giman Drt,La Jola.CAg20G3.5A INSERM.UB23:Universite Joseph Fourier-Grenoble 1:Institut Albert Bonniot.Facut de Mddecine,Domaine de la Merci. 38706 La Tronche Cedex.France Shanghai Institute of Materia Medica.Chinese Academy of Sciences,555 Zu Chong Zhi Road Shanghai 201203,P.R.China Department ol Chemical and Systems Biology.Starford University School of Medicine,Starlord.CA 94305.USA Dr.Yingming Zhao 0010.1018c201108.008 M MIV VII VII X细 Hyper-crotonylation wave in elongating spermatids 9-11 3 图巴豆酰化修饰促进精子发育成熟
3 Kcr功能:调控精子发生的成熟过程 Dr. Yingming Zhao 图 巴豆酰化修饰促进精子发育成熟

INDING Group Kcr调控压力胁迫下的抑郁症反应 ARTICLE IN PRESS Archival Report Chromodomain Y-like Protein-Mediated Histone Crotonylation Regulates Stress-Induced Depressive Behaviors Yongqing Liu,Minghua Li,Minghua Fan,Yan Song,Huajing Yu,Xiaojie Zhi,Kuo Xiao, Shirong Lai,Jingliang Zhang,Xueqin Jin,Yongfeng Shang,Jing Liang,and Zhuo Huang Dr.Jing Liang Control or Resilient CDYL EZH2 SStOISY PL dendritic spines CDYL Susceptible EZH2 CDYL CDYL Dendritic spines loss H3K27me3 PanKcr 4 图转录抑制因子CDYL和巴豆酰化修饰降低转录抑制神经肽VGF的表达从而促进抑郁症
4 Kcr调控压力胁迫下的抑郁症反应 图 转录抑制因子CDYL和巴豆酰化修饰降低转录抑制神经肽VGF的表达从而促进抑郁症 Dr. Jing Liang
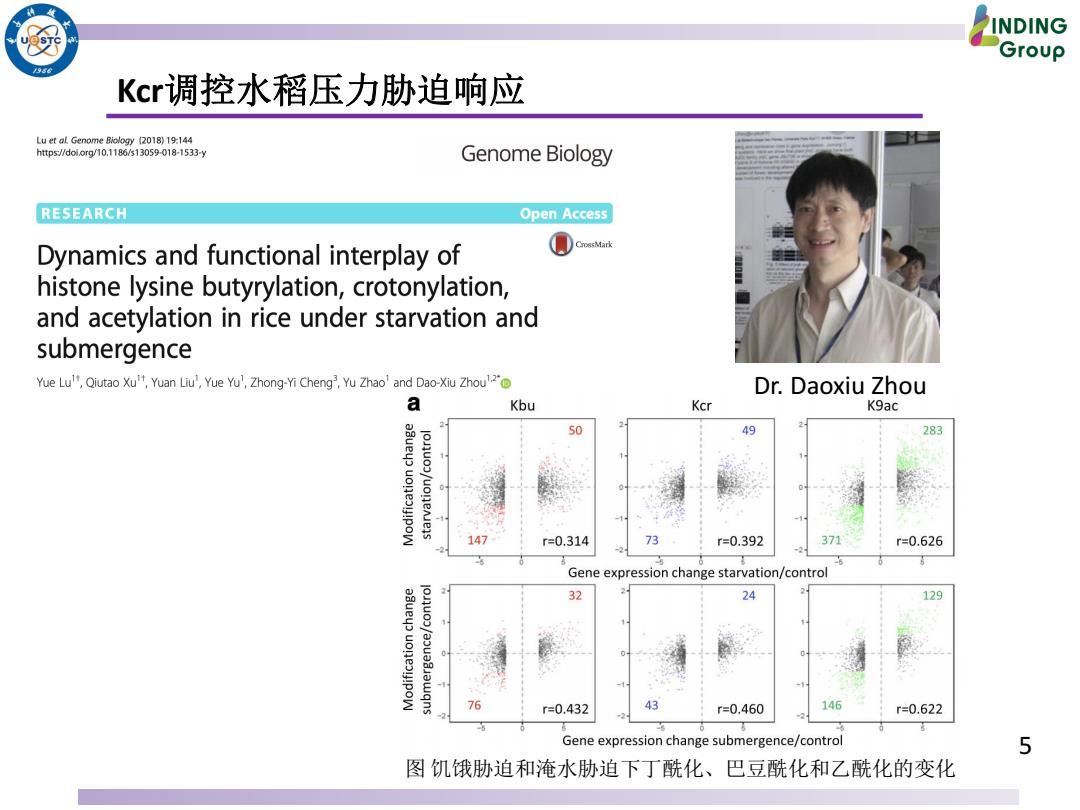
INDING Group Kcr调控水稻压力胁迫响应 Lu et aL Genome Biology (2018)19:144 http5/doi.org/10,1186/513059-018-1533-y Genome Biology RESEARCH Open Access Dynamics and functional interplay of histone lysine butyrylation,crotonylation, and acetylation in rice under starvation and submergence Yue Lut,Qiutao Xu't,Yuan Liu',Yue Yu',Zhong-Yi Cheng3,Yu Zhao'and Dao-Xiu Zhou' Dr.Daoxiu Zhou a Kbu Kcr K9ac 50 49 283 147 r=0.314 73 r=0.392 371 r=0.626 Gene expression change starvation/control 32 24 129 76 r=0.432 43 r=0.460 146 r=0.622 Gene expression change submergence/control 5 图饥饿胁迫和淹水胁迫下丁酰化、巴豆酰化和乙酰化的变化
5 Kcr调控水稻压力胁迫响应 图 饥饿胁迫和淹水胁迫下丁酰化、巴豆酰化和乙酰化的变化 Dr. Daoxiu Zhou
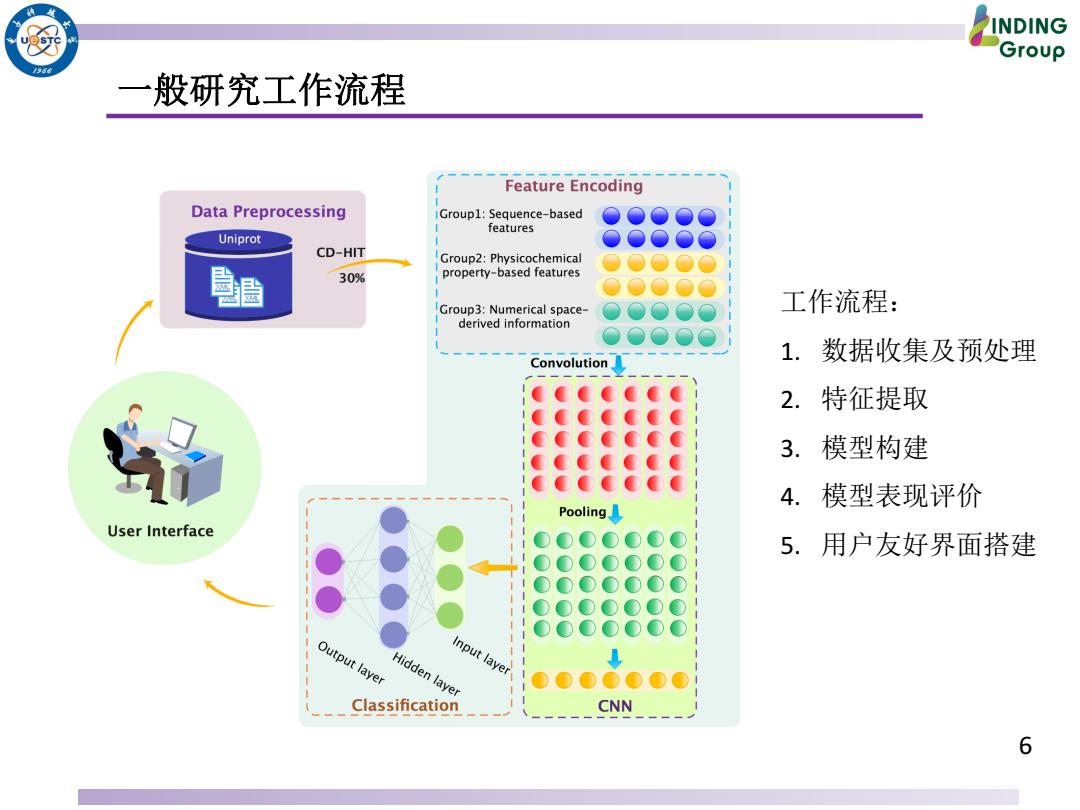
INDING Group 一般研究工作流程 Feature Encoding Data Preprocessing Groupl:Sequence-based features Uniprot CD-HIT Group2:Physicochemical 30% property-based features Group3:Numerical space- 工作流程: derived information 1.数据收集及预处理 Convolution 2.特征提取 3.模型构建 4.模型表现评价 Pooling User Interface 5.用户友好界面搭建 Output layer Hidden layer Input layer Classification CNN 6
6 一般研究工作流程 工作流程: 1. 数据收集及预处理 2. 特征提取 3. 模型构建 4. 模型表现评价 5. 用户友好界面搭建
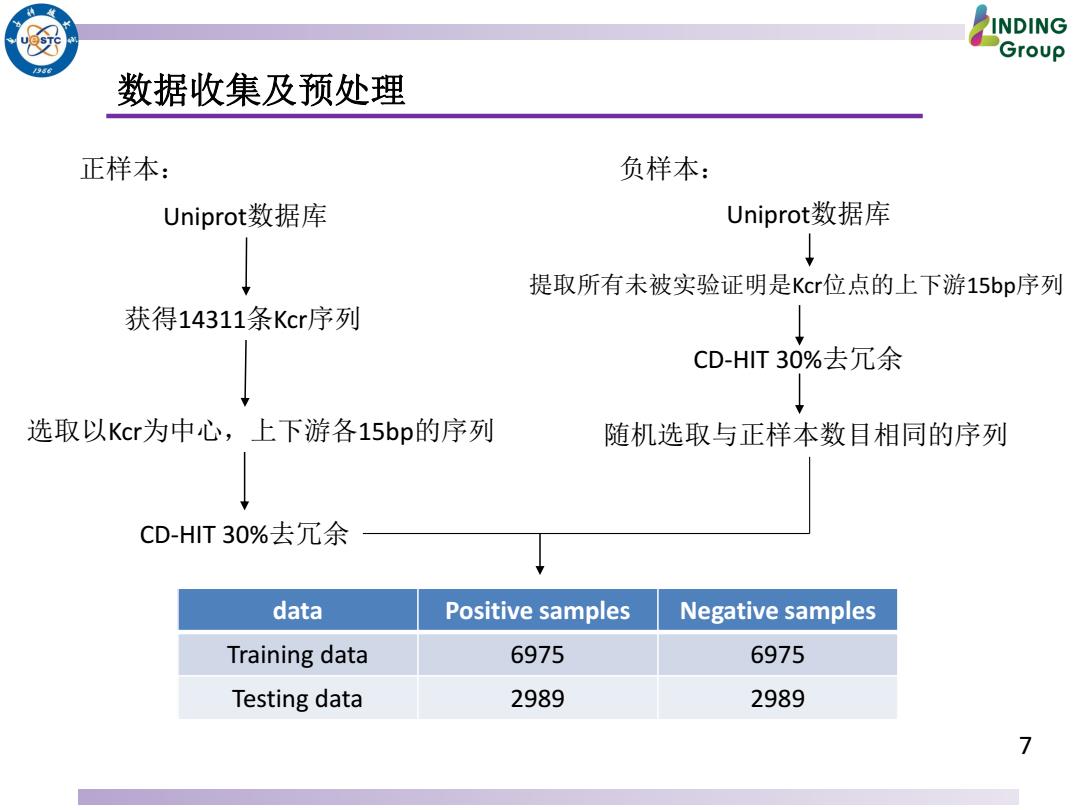
INDING Group 数据收集及预处理 正样本: 负样本: Uniprot数据库 Uniprot数据库 提取所有未被实验证明是Kcr位点的上下游15bp序列 获得14311条Kcr序列 CD-HIT30%去冗余 选取以Kcr为中心,上下游各15bp的序列 随机选取与正样本数目相同的序列 CD-HIT30%去冗余 data Positive samples Negative samples Training data 6975 6975 Testing data 2989 2989 7
7 数据收集及预处理 Uniprot数据库 获得14311条Kcr序列 选取以Kcr为中心,上下游各15bp的序列 CD-HIT 30%去冗余 data Positive samples Negative samples Training data 6975 6975 Testing data 2989 2989 正样本: 负样本: Uniprot数据库 提取所有未被实验证明是Kcr位点的上下游15bp序列 CD-HIT 30%去冗余 随机选取与正样本数目相同的序列

ov 96 特征提取 》基于序列信息的特征 》基于物化性质的特征 》基于数值空间信息的特征 8
8 特征提取 基于序列信息的特征 基于物化性质的特征 基于数值空间信息的特征

INDING Group 基于序列信息的特征 1.K-space氨基酸对组分(CKSAAP) CKSAAP反映了氨基酸对之间的短程相互作用信息。假设K=0,那么有400(20*20) 个间隔为0的残基对(即AA,AC,AD,,YY).可以用下式计算特征向量: NAA NAC NAP NYY NTotal'NTotal'NTotal 'NTotal/400 其中,NTotau是总的残基组分的长度(例如,如果长度为L的蛋白质片段为31且 k=0、1、2、3、4和5,则NTotal=L-k-1,分别对应为为30、29、28、27、 26和25)。NAA、W4c、NAD、Nyy分别代表氨基酸对的频率。在本研究中设k= 0、1、2、3、4和5,因此基于CKSAAP的特征向量的总维数为400×6=2400。 9
9 基于序列信息的特征 1. K-space氨基酸对组分 (CKSAAP) CKSAAP反映了氨基酸对之间的短程相互作用信息。假设K = 0, 那么有400 (20*20) 个间隔为0的残基对 (即AA, AC, AD, …, YY). 可以用下式计算特征向量: 𝑁𝐴𝐴 𝑁𝑇𝑜𝑡𝑎𝑙 , 𝑁𝐴𝐶 𝑁𝑇𝑜𝑡𝑎𝑙 , 𝑁𝐴𝐷 𝑁𝑇𝑜𝑡𝑎𝑙 , ⋯ , 𝑁𝑌𝑌 𝑁𝑇𝑜𝑡𝑎𝑙 400 其中, 𝑁𝑇𝑜𝑡𝑎𝑙是总的残基组分的长度(例如,如果长度为 𝐿 的蛋白质片段为31且 k = 0、1、2、3、4和5,则𝑁𝑇𝑜𝑡𝑎𝑙 = 𝐿 − 𝑘 − 1,分别对应为为30、29、28、27、 26和25)。 𝑁𝐴𝐴 、𝑁𝐴𝐶 、𝑁𝐴𝐷 、𝑁𝑌𝑌分别代表氨基酸对的频率。 在本研究中设𝑘 = 0、1、2、3、4和5,因此基于CKSAAP的特征向量的总维数为400×6 = 2400