
第六章生物信息学和进化的研究方法 2007 W.H.Freeman and Company GLSDGEWQLVLNVWGKVEADIPGHGQEVLIRLFKGHPETLEKFDKFKHLKSEDEMKASEDLKKHGATVLTALGGIL- GLSDGEWOLVLNVWGKVEADIPGHGOEVLIRLFKGHPETLEKFDKFKHLKSEDEMKASEDLKKHGATVLTALGGIL- KKKGHHEAEIKPLAQSHATKHKIPVKYLEFISECIIQVLHSKHPGDFGADAQGAMNKALELFRKDMASNYKELGFQG KKKGHHEAEIKPLAQSHATKHKIPVKYLEFISECIIQVLQSKHPGDFGADAQGAMNKALELFRKDMASNYKELGFQG Chapter 6 Opener part 2 Biochemistry.Sixth Edition 2007 W.H.Freeman and Company 蛋白质序列清楚地反映出生物的进化关系。Jane Goodall和一只黑猩猩互动提示人类与黑猩 猩亲缘关系近。血红蛋白的氨基酸序列也证明人类与黑猩猩的亲缘关系。在血红蛋白153 个氨基酸残基中,人血红蛋白(红色)与黑猩猩血红蛋白(蓝色)只有一个氨基酸残基不同。 [左边,Kennan Ward/Corbis.] 如同人类家族的成员一样,分子家族的成员有一些共同的特征。比较蛋白质的三维结构 (即与蛋白功能关系最密切的参数),我们很容易鉴定蛋白家族成员。我们在蛋白质折叠章 节介绍过的牛核酸酶,其三维结构与人核酸酶的三维结构颇为类似(图6.1)。虽然这种类似 性在意料之中(因为这两个蛋白质的功能是一样的),但是有些情况下这种比较却获得了我们 意料之外的结果。例如血管生成素(angiogenin)是能够刺激血管生成的蛋白质。但是血管生 成素的三维结构与核酸酶三维结构非常相似,说明血管生成素和核酸酶属于同一蛋白家族 (图62)。因此它们肯定来自同一祖先分子
第六章 生物信息学和进化的研究方法 蛋白质序列清楚地反映出生物的进化关系。Jane Goodall 和一只黑猩猩互动提示人类与黑猩 猩亲缘关系近。血红蛋白的氨基酸序列也证明人类与黑猩猩的亲缘关系。在血红蛋白 153 个氨基酸残基中,人血红蛋白(红色)与黑猩猩血红蛋白(蓝色)只有一个氨基酸残基不同。 [左边, Kennan Ward/Corbis.] 如同人类家族的成员一样,分子家族的成员有一些共同的特征。比较蛋白质的三维结构 (即与蛋白功能关系最密切的参数),我们很容易鉴定蛋白家族成员。我们在蛋白质折叠章 节介绍过的牛核酸酶,其三维结构与人核酸酶的三维结构颇为类似(图 6.1)。虽然这种类似 性在意料之中(因为这两个蛋白质的功能是一样的),但是有些情况下这种比较却获得了我们 意料之外的结果。例如血管生成素(angiogenin)是能够刺激血管生成的蛋白质。但是血管生 成素的三维结构与核酸酶三维结构非常相似,说明血管生成素和核酸酶属于同一蛋白家族 (图 6.2)。因此它们肯定来自同一祖先分子

Bovine ribonuclease Human ribonuclease adoen 图6.1牛和人核酸酶结构。功能类似性的蛋白质常常具有结构类似性。Drawn from8 RAT.pdb. and 2RNF.pdb] Angiogenin 的n 图62血管生成素的结构。该蛋白质能促进血管生长,具有与核酸酶非常相似的三维结构。 遗憾的是,知道三维结构的蛋白质数量有限。我们知道很多蛋白质的编码基因序列或氨 基酸序列(得盏于DNA克隆和DNA序列测定)。在蛋白质氨基酸序列水平就能观测到蛋白 质的进化关系。例如牛核糖核酸酶和血管生成素的氨基酸序列有35%的氨基酸残基是一致 的。那么这种相似性水平是否足以保证蛋白质的进化关系?如果不够,需要多高的相似性才 能说明蛋白质有进化关系?本章介绍氨基酸序列比较的方法和推测蛋白质进化关联的方法。 序列比较已经成为现代生物化学研究的最有力工具之一。最新鉴定的新序列与数据库比 较,确定与该序列相关的分子。利用这些信息,能够研究具有这种新近测序分子功能和机制。 如果三维结构也清楚,我们能比较三维结构、证实序列比较的结论,还能揭示那些序列比较 没有发现的成员。 考察现有蛋白序列的脚印,生化学家成了分子考古学家,能了解分子进化史所发生的事 件。序列比较能够确定进化途径、估计特定进化事件发生的时间。利用这些信息能够构建特 定蛋白质或核酸从古生菌和细菌到真核生物(包括人类)的进化树。也可以用实验方法研究 分子进化。有些化石的DNA分子也可以进行PCR扩增和测序,直接观察古代的分子序列。 此外科研人员也可以进行一些核酸复制实验观察实验室的分子进化。这些研究的结果能揭示 更多的分子进化机制
图 6.1 牛和人核酸酶结构。功能类似性的蛋白质常常具有结构类似性。[Drawn from 8RAT.pdb. and 2RNF.pdb] 图 6.2 血管生成素的结构。该蛋白质能促进血管生长,具有与核酸酶非常相似的三维结构。 遗憾的是,知道三维结构的蛋白质数量有限。我们知道很多蛋白质的编码基因序列或氨 基酸序列(得益于 DNA 克隆和 DNA 序列测定)。在蛋白质氨基酸序列水平就能观测到蛋白 质的进化关系。例如牛核糖核酸酶和血管生成素的氨基酸序列有 35%的氨基酸残基是一致 的。那么这种相似性水平是否足以保证蛋白质的进化关系?如果不够,需要多高的相似性才 能说明蛋白质有进化关系?本章介绍氨基酸序列比较的方法和推测蛋白质进化关联的方法。 序列比较已经成为现代生物化学研究的最有力工具之一。最新鉴定的新序列与数据库比 较,确定与该序列相关的分子。利用这些信息,能够研究具有这种新近测序分子功能和机制。 如果三维结构也清楚,我们能比较三维结构、证实序列比较的结论,还能揭示那些序列比较 没有发现的成员。 考察现有蛋白序列的脚印,生化学家成了分子考古学家,能了解分子进化史所发生的事 件。序列比较能够确定进化途径、估计特定进化事件发生的时间。利用这些信息能够构建特 定蛋白质或核酸从古生菌和细菌到真核生物(包括人类)的进化树。也可以用实验方法研究 分子进化。有些化石的 DNA 分子也可以进行 PCR 扩增和测序,直接观察古代的分子序列。 此外科研人员也可以进行一些核酸复制实验观察实验室的分子进化。这些研究的结果能揭示 更多的分子进化机制

6.1同源物来自同一祖先 生物化学进化的研究主要是确定蛋白质、其它生物分子、以及生物化学途径如何随着时 间发生的变化。来自同一祖先的两个物体就是同源关系。同源(homologus)分子或同源物 (homologs)可以分为两类(图6.3)。侧向同源物(paralogs)指同种生物的同源物。直向同源 物(orthologs)指不同物种间具有非常相似甚至一致功能的同源物。了解分子间的同源性能够 揭示分子的进化历史和它们的生物功能。如果新近测序的蛋白质与功能已知蛋白具有同源 性,那么这个新近测序的蛋白质也可能具有这样的生物功能。 如何确定人类两种蛋白质具有侧向同源?如何确定人类蛋白和酵母蛋白具有直向同 源?我们在62节要介绍这方面的内容。核酸序列或氨基酸序列之间有显著的类似性常常表 明这些物质之间有同源性。当然,三维结构比较更能揭示生物分子的同源性。 Bovine ribonudlease (digestive enzyme) Orthologs HUMAN BEING Paralogs Human ribonuclease Angiogenin (digestive enzyme) (stimulates blood-vessel growth) 图63两类同源蛋白。不同生物体内执行类似甚至相同功能蛋白质属于垂直同源物 (ortholog)。而同一物种内执行不同功能的相似蛋白叫侧向同源蛋白(paralog)。 6.2序列比对的统计分析检测同源性(omol0gy) 两个分子之间有显著的序列类似性提示这两个分子可能有共同的进化祖先,因此有同样 的三维结构、生物功能和作用机制。虽然核酸序列和蛋白质序列都可用来进行序列比对,但 是蛋白质氨基酸序列比对更好。最显著的原因是构建蛋白质的单体有20种,而构建核酸的 单体只有4种。 为了解释蛋白质比较的方法,我们先考察球蛋白。肌红蛋白是肌肉组织的氧结合蛋白, 而血红蛋白是血液的载氧蛋白(第7章)。这两种蛋白质都有血红素辅基(一种有铁的有机 分子,能结合氧)。人血红蛋白有4个多肽链,每个多肽链结合一个血红素辅基。这四个多 肽链分别是两个完全一样的-链和两个完全一致的B-链。此处我们仅考察-链。研究人血 红蛋白-链的氨基酸序列与人肌红蛋白氨基酸序列之间的类似性(图6.4)。为了确定这两 个序列之间的类似性,建立了序列比对方法
6.1 同源物来自同一祖先 生物化学进化的研究主要是确定蛋白质、其它生物分子、以及生物化学途径如何随着时 间发生的变化。来自同一祖先的两个物体就是同源关系。同源(homologus)分子或同源物 (homologs)可以分为两类(图 6.3)。侧向同源物(paralogs)指同种生物的同源物。直向同源 物(orthologs)指不同物种间具有非常相似甚至一致功能的同源物。了解分子间的同源性能够 揭示分子的进化历史和它们的生物功能。如果新近测序的蛋白质与功能已知蛋白具有同源 性,那么这个新近测序的蛋白质也可能具有这样的生物功能。 如何确定人类两种蛋白质具有侧向同源?如何确定人类蛋白和酵母蛋白具有直向同 源?我们在 6.2 节要介绍这方面的内容。核酸序列或氨基酸序列之间有显著的类似性常常表 明这些物质之间有同源性。当然,三维结构比较更能揭示生物分子的同源性。 图 6.3 两类同源蛋白。不同生物体内执行类似甚至相同功能蛋白质属于垂直同源物 (ortholog)。而同一物种内执行不同功能的相似蛋白叫侧向同源蛋白(paralog)。 6.2 序列比对的统计分析检测同源性(homology) 两个分子之间有显著的序列类似性提示这两个分子可能有共同的进化祖先,因此有同样 的三维结构、生物功能和作用机制。虽然核酸序列和蛋白质序列都可用来进行序列比对,但 是蛋白质氨基酸序列比对更好。最显著的原因是构建蛋白质的单体有 20 种,而构建核酸的 单体只有 4 种。 为了解释蛋白质比较的方法,我们先考察球蛋白。肌红蛋白是肌肉组织的氧结合蛋白, 而血红蛋白是血液的载氧蛋白(第 7 章)。这两种蛋白质都有血红素辅基(一种有铁的有机 分子,能结合氧)。人血红蛋白有 4 个多肽链,每个多肽链结合一个血红素辅基。这四个多 肽链分别是两个完全一样的链和两个完全一致的链。此处我们仅考察链。研究人血 红蛋白链的氨基酸序列与人肌红蛋白氨基酸序列之间的类似性(图 6.4)。为了确定这两 个序列之间的类似性,建立了序列比对方法
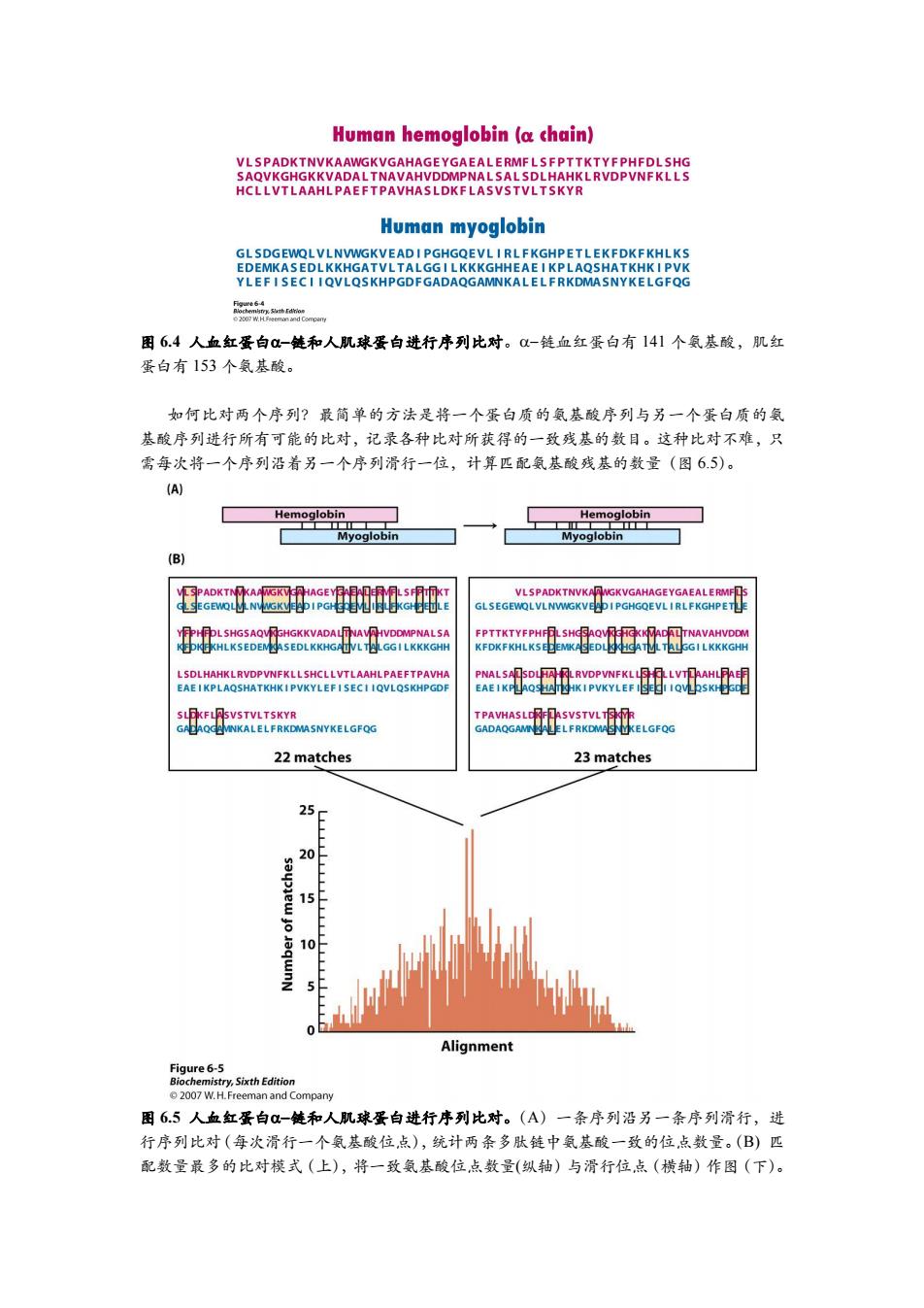
Human hemoglobin (a chain) VLSPADKTNVKAAWGKVGAHAGEYGAEALERMFLSFPTTKTYFPHFDLSHG SAQVKGHGKKVADALTNAVAHVDDMPNALSALSDLHAHKLRVDPVNFKLLS HCLLVTLAAHLPAEFTPAVHASLDKFLASVSTVLTSKYR Human myoglobin GLSDGEWQLVLNVWGKVEADI PGHGQEVL IRLFKGHPETLEKFDKFKHLKS EDEMKASEDLKKHGATVLTALGGI LKKKGHHEAE I KPLAQSHATKHKI PVK YLEFISECI IQVLQSKHPGDFGADAQGAMNKALELFRKDMASNYKELGFQG Figure 困6.4人血红蛋白-链和人肌球蛋白进行序列比对。0-链血红蛋白有141个氨基酸,肌红 蛋白有153个氨基酸。 如何比对两个序列?最简单的方法是将一个蛋白质的氨基酸序列与另一个蛋白质的氨 基酸序列进行所有可能的比对,记录各种比对所获得的一致残基的数目。这种比对不难,只 需每次将一个序列沿着另一个序列滑行一位,计算匹配氨基酸残基的数量(图6.5)。 (A) Hemoglobin Hemoglobin T Myoglobin Myoglobin (B) GLSEGEWQLVLNWGKVEADI PGHGQEVL I RLFKGHPETL YFPHFDLSHGSAQVGHGKKVADALTNAVAHVDDMPNALSA FPTTKTYFPHFDLSHGSAOVGHGKKMADALTNAVAHVDDM LSDLHAHKLRVDPVNFKLLSHCLLVTLAAHLPAEFTPAVHA EAEIKPLAQSHATKHKIPVKYLEFISECI IQVLQSKHPGDF SLDKFL可SVSTVLTSKYR T PAVHASLDRFLASVSTVLTSKYR 22 matches 23 matches 15 10 Alignment Figure 6-5 Biochemistry,Sixth Edition 2007 W.H.Freeman and Company 图6.5人血红蛋白α-链和人肌球蛋白进行序列比对。(A)一条序列沿另一条序列滑行,进 行序列比对(每次滑行一个氨基酸位点),统计两条多肽链中氨基酸一致的位点数量。(B)匹 配数量最多的比对模式(上),将一致氨基酸位点数量(纵轴)与滑行位点(横轴)作图(下)
图 6.4 人血红蛋白链和人肌球蛋白进行序列比对。链血红蛋白有 141 个氨基酸,肌红 蛋白有 153 个氨基酸。 如何比对两个序列?最简单的方法是将一个蛋白质的氨基酸序列与另一个蛋白质的氨 基酸序列进行所有可能的比对,记录各种比对所获得的一致残基的数目。这种比对不难,只 需每次将一个序列沿着另一个序列滑行一位,计算匹配氨基酸残基的数量(图 6.5)。 图 6.5 人血红蛋白链和人肌球蛋白进行序列比对。(A)一条序列沿另一条序列滑行,进 行序列比对(每次滑行一个氨基酸位点),统计两条多肽链中氨基酸一致的位点数量。(B) 匹 配数量最多的比对模式(上),将一致氨基酸位点数量(纵轴)与滑行位点(横轴)作图(下)

-链和肌红蛋白最佳比对只有23个位点是一致的,分散在多肽链中央区域。但是,相 近的比对有22个一致位点,而且这些位,点集中于蛋白质的N-端。如果在比对过程中引入缺 口,就能将上述两种比对的大多数一致位点都囊括进来(图66)。在比对过程中需要添加这 种缺口以补偿基因进化过程中出现的插入或缺失。 Hemoglobin VLSPADK TNVKAAWGKVGAHAGE YGAEALERMFL S FPTTKTYFP HF- Gap Myoglobin L SHGSAQVKGHGKKVADALTNAVAHVDDMPNA L SALSDLHAHKL RVDPVNKK L EMKASEDLKKHGATVLTALGGI LKKKGHHEAE I KPLAQSHATKHK I PVKYLE F HOLLVTLAAHDPADEARRASLDKFLASVSTVLTSK GADAQGAMN LFRKDMA$NU冰ELGFQG 2007 W.H.Freeman and Company 图6.6添加缺口的比对。人血红蛋白0-链和肌红蛋白在加入缺口后的序列比对。 但是用缺口的办法增加了序列比对的复杂性,因为序列比对时所选择的缺口大小有人为 因素。现在已建立了计算机自动进行序列比对时添加缺口的方案。这种方法采用打分系统评 估各种比对方素,利用缺口惩罚来排除序列比对时出现的不合理缺口。例如一致位点加10 分,一个缺口(无论缺口大小)扣25分。图6.6的比对方案得355分(38个一致位点和一个 缺口,38x10-1x25=355)。这样,这两个蛋白质之间氨基酸一致性就达到25.9%(两 个蛋白质平均长度是147氨基酸残基,有38个一致位,点)。下面的问题是,这样的一致性是 否属于明显类似? 用重排的方法估计比对的统计学意义 图65展示的序列类似性似乎是显著的,但是不能排除偶然出现了这些一致性的可能。 如何评估这些偶然性?采用的策略是将某一蛋白质的氨基酸序列进行随机重排(shuffling), 然后将重排所得蛋白序列与目标序列比对(图67)。重复这一过程,构建出各种重排蛋白的 得分。 采用这种策略,肌红蛋白和血红蛋白-链之间权威比对就凸现出来(图68)。权咸比对 的数值远高于随机重排蛋白的平均值。随机重排10次,才出现一次这种比对值。因此我 们可以轻松地认为这两个序列确实是类似的。最简单的解释是这两种蛋白序列来自同一祖 先,属于同源物。 THISISTHEAUTHENTICSEQUENCE Shuffling SNUCSNSEATEEITUHEQIHHTTCEI m 图6.7重排产生的一个新序列
链和肌红蛋白最佳比对只有 23 个位点是一致的,分散在多肽链中央区域。但是,相 近的比对有 22 个一致位点,而且这些位点集中于蛋白质的 N-端。如果在比对过程中引入缺 口,就能将上述两种比对的大多数一致位点都囊括进来(图 6.6)。在比对过程中需要添加这 种缺口以补偿基因进化过程中出现的插入或缺失。 图 6.6 添加缺口的比对。人血红蛋白链和肌红蛋白在加入缺口后的序列比对。 但是用缺口的办法增加了序列比对的复杂性,因为序列比对时所选择的缺口大小有人为 因素。现在已建立了计算机自动进行序列比对时添加缺口的方案。这种方法采用打分系统评 估各种比对方案,利用缺口惩罚来排除序列比对时出现的不合理缺口。例如一致位点加 10 分,一个缺口(无论缺口大小)扣 25 分。图 6.6 的比对方案得 355 分(38 个一致位点和一个 缺口, 38 x 10 - 1 x 25 = 355)。这样,这两个蛋白质之间氨基酸一致性就达到 25.9% (两 个蛋白质平均长度是 147 氨基酸残基,有 38 个一致位点)。下面的问题是,这样的一致性是 否属于明显类似? 用重排的方法估计比对的统计学意义 图 6.5 展示的序列类似性似乎是显著的,但是不能排除偶然出现了这些一致性的可能。 如何评估这些偶然性?采用的策略是将某一蛋白质的氨基酸序列进行随机重排(shuffling), 然后将重排所得蛋白序列与目标序列比对(图 6.7)。重复这一过程,构建出各种重排蛋白的 得分。 采用这种策略,肌红蛋白和血红蛋白链之间权威比对就凸现出来(图 6.8)。权威比对 的数值远高于随机重排蛋白的平均值。随机重排 10 20次,才出现一次这种比对值。因此我 们可以轻松地认为这两个序列确实是类似的。最简单的解释是这两种蛋白序列来自同一祖 先,属于同源物。 图 6.7 重排产生的一个新序列
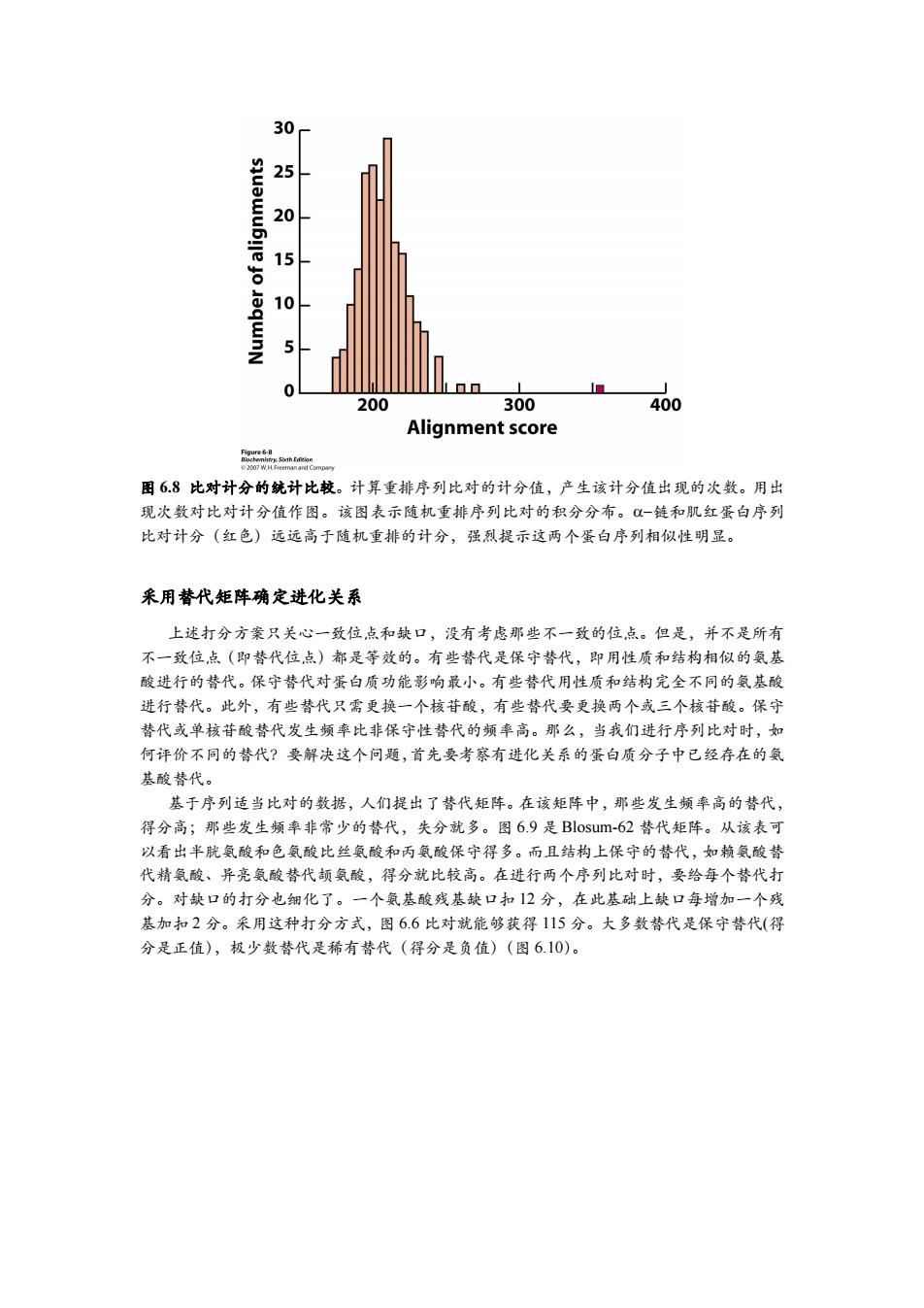
2520 15 10 L▣口 200 300 400 Alignment score 图68比对计分的统计比较。计算重排序列比对的计分值,产生该计分值出现的次数。用出 现次数对比对计分值作图。该图表示随机重排序列比对的积分分布。一链和肌红蛋白序列 比对计分(红色)远远高于随机重排的计分,强烈提示这两个蛋白序列相似性明显。 采用替代矩阵确定进化关系 上述打分方案只关心一致位点和缺口,没有考虑那些不一致的位点。但是,并不是所有 不一致位点(即替代位点)都是等效的。有些替代是保守替代,即用性质和结构相似的氨基 酸进行的替代。保守替代对蛋白质功能影响最小。有些替代用性质和结构完全不同的氨基酸 进行替代。此外,有些替代只需更换一个核苷酸,有些替代要更换两个或三个核苷酸。保守 替代或单核苷酸替代发生频率比非保守性替代的频率高。那么,当我们进行序列比对时,如 何评价不同的替代?要解决这个问题,首先要考察有进化关系的蛋白质分子中已经存在的氨 基酸替代。 基于序列适当比对的数据,人们提出了替代矩阵。在该矩阵中,那些发生频率高的替代, 得分高;那些发生频率非常少的替代,失分就多。图69是Blosum-62替代矩阵。从该表可 以看出半胱氨酸和色氨酸比丝氨酸和丙氨酸保守得多。而且结构上保守的替代,如赖氨酸替 代精氨酸、异亮氨酸替代颉氨酸,得分就比较高。在进行两个序列比对时,要给每个替代打 分。对缺口的打分也细化了。一个氨基酸残基缺口扣12分,在此基础上缺口每增加一个残 基加扣2分。采用这种打分方式,图6.6比对就能够获得115分。大多数替代是保守替代(得 分是正值),极少数替代是稀有替代(得分是负值)(图6.10)
图 6.8 比对计分的统计比较。计算重排序列比对的计分值,产生该计分值出现的次数。用出 现次数对比对计分值作图。该图表示随机重排序列比对的积分分布。链和肌红蛋白序列 比对计分(红色)远远高于随机重排的计分,强烈提示这两个蛋白序列相似性明显。 采用替代矩阵确定进化关系 上述打分方案只关心一致位点和缺口,没有考虑那些不一致的位点。但是,并不是所有 不一致位点(即替代位点)都是等效的。有些替代是保守替代,即用性质和结构相似的氨基 酸进行的替代。保守替代对蛋白质功能影响最小。有些替代用性质和结构完全不同的氨基酸 进行替代。此外,有些替代只需更换一个核苷酸,有些替代要更换两个或三个核苷酸。保守 替代或单核苷酸替代发生频率比非保守性替代的频率高。那么,当我们进行序列比对时,如 何评价不同的替代?要解决这个问题,首先要考察有进化关系的蛋白质分子中已经存在的氨 基酸替代。 基于序列适当比对的数据,人们提出了替代矩阵。在该矩阵中,那些发生频率高的替代, 得分高;那些发生频率非常少的替代,失分就多。图 6.9 是 Blosum-62 替代矩阵。从该表可 以看出半胱氨酸和色氨酸比丝氨酸和丙氨酸保守得多。而且结构上保守的替代,如赖氨酸替 代精氨酸、异亮氨酸替代颉氨酸,得分就比较高。在进行两个序列比对时,要给每个替代打 分。对缺口的打分也细化了。一个氨基酸残基缺口扣 12 分,在此基础上缺口每增加一个残 基加扣 2 分。采用这种打分方式,图 6.6 比对就能够获得 115 分。大多数替代是保守替代(得 分是正值),极少数替代是稀有替代(得分是负值)(图 6.10)

Score 15 圆 © 风 回 可 国 四 国 ⊙ 圆四回 a 圆 国 回 @ ▣四 ⊙ -¥V 1- 发 3 M1- ¥ -HW- EN -D -0 -5- LM- M 0 HR ER -NS 器 -NAV TGV -SA DH N5- RG TAM- R EKO TA c痛 留 ST AV LV- -1 AY ( -2一 P DA GPM 路 -PY DRG HRN PLY -DKT 贤 ACY 警 -3 6M -TGP PMY- -¥ 部 KQ 4 CIW 新 W 野 CMW 供 肤 響 GP ERN CP -5 CH 思 W 860 6 DHG DG -6 Figure6-9 Biochemistry,Sixth Edition 2007 W.H.Freeman and Company 图6.9Bl0sum-62替代矩阵。考察同源蛋白各种替代发生的机会,制定该打分方案。氨基酸 归为四类:带电氨基酸(红色);极性氨基酸(绿色);大的非极性氨基酸(蓝色);其它氨 基酸(黑色)。只需要改变一个核苷酸的替代用阴影标出。当你寻找一种替代应该给出的打 分时,你在顶部寻找原来的氨基酸(在每列的顶部),然后向下寻找替代氨基酸,从表格左 边查找相应的得分
图 6.9 Blosum-62 替代矩阵。考察同源蛋白各种替代发生的机会,制定该打分方案。氨基酸 归为四类:带电氨基酸(红色);极性氨基酸(绿色);大的非极性氨基酸(蓝色);其它氨 基酸(黑色)。只需要改变一个核苷酸的替代用阴影标出。当你寻找一种替代应该给出的打 分时,你在顶部寻找原来的氨基酸(在每列的顶部),然后向下寻找替代氨基酸,从表格左 边查找相应的得分
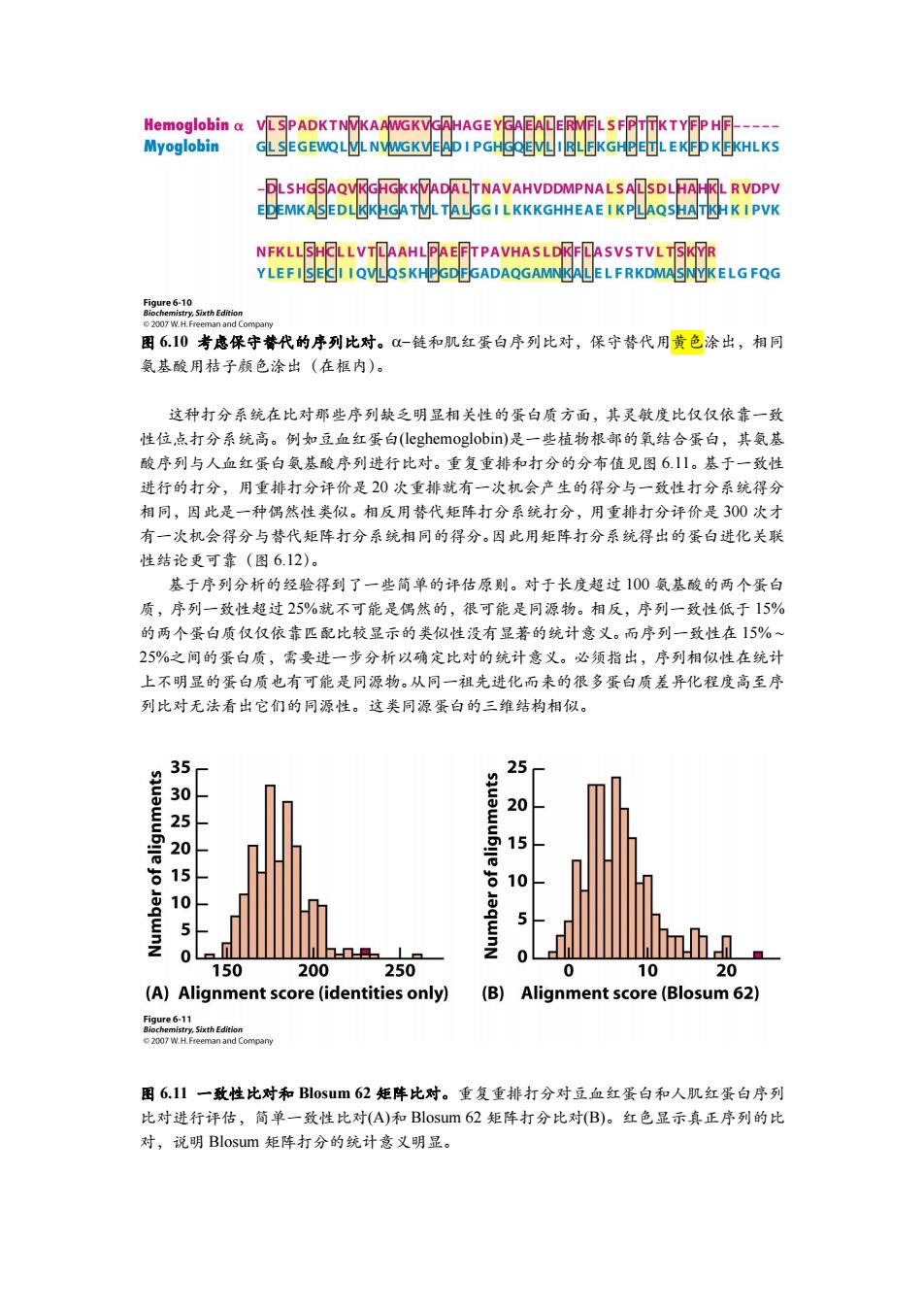
Hemoglobin VLSPADKTNVKAAWGKVGAHAGE YGAEALERMFL S FPTTKTYFP HF----- Myoglobin GLSEGEWQLVLNVGKVEAD I PGHGQEVLIRLFKGHPETLEKFD KFKHLKS DLSHGSAQVKGHGKKVADALTNAVAHVDDMPNAL SALSDLHAHKL RVDPV EDEMKASEDLKKHGATVLTALGGI LKKKGHHEAE I KPLAQSHATKHK I PVK NFKLLSHOLLVTLAAHLPAEFTPAVHASLDKFLASVSTVLTSKYR YLEFISECI I QVLQSKHPGDFGADAQGAMNKALEL FRKDMASNYKELG FQG 2007 W.H.Freeman and Company 图610考虑保守替代的序列比对。0-链和肌红蛋白序列比对,保守替代用黄色涂出,相同 氨基酸用桔子颜色涂出(在框内)。 这种打分系统在比对那些序列缺乏明显相关性的蛋白质方面,其灵敏度比仅仅依靠一致 性位,点打分系统高。例如豆血红蛋白(leghemoglobin)是一些植物根部的氧结合蛋白,其氨基 酸序列与人血红蛋白氨基酸序列进行比对。重复重排和打分的分布值见图6.11。基于一致性 进行的打分,用重排打分评价是20次重排就有一次机会产生的得分与一致性打分系统得分 相同,因此是一种偶然性类似。相反用替代矩阵打分系统打分,用重排打分评价是300次才 有一次机会得分与替代矩阵打分系统相同的得分。因此用矩阵打分系统得出的蛋白进化关联 性结论更可靠(图6.12)。 基于序列分析的经验得到了一些简单的评估原则。对于长度超过100氨基酸的两个蛋白 质,序列一致性超过25%就不可能是偶然的,很可能是同源物。相反,序列一致性低于15% 的两个蛋白质仅仅依靠匹配比较显示的类似性没有显著的统计意义。而序列一致性在15%~ 25%之间的蛋白质,需要进一步分析以确定比对的统计意义。必须指出,序列相似性在统计 上不明显的蛋白质也有可能是同源物。从同一祖先进化而来的很多蛋白质差异化程度高至序 列比对无法看出它们的同源性。这类同源蛋白的三维结构相似。 3 25 20 25 15 5 10 aqwnN 5 5 0 150 200 250 O 10 20 (A)Alignment score(identities only) (B) Alignment score(Blosum 62) Figure 6-11 Biochemistry,Sixth Edition 2007 W.H.Freeman and Company 图6.1山1一致性比对和Bl0sum62矩阵比对。重复重排打分对豆血红蛋白和人肌红蛋白序列 比对进行评估,简单一致性比对(A)和Blosum62矩阵打分比对(B)。红色显示真正序列的比 对,说明Blosum矩阵打分的统计意义明显
图 6.10 考虑保守替代的序列比对。链和肌红蛋白序列比对,保守替代用黄色涂出,相同 氨基酸用桔子颜色涂出(在框内)。 这种打分系统在比对那些序列缺乏明显相关性的蛋白质方面,其灵敏度比仅仅依靠一致 性位点打分系统高。例如豆血红蛋白(leghemoglobin)是一些植物根部的氧结合蛋白,其氨基 酸序列与人血红蛋白氨基酸序列进行比对。重复重排和打分的分布值见图 6.11。基于一致性 进行的打分,用重排打分评价是 20 次重排就有一次机会产生的得分与一致性打分系统得分 相同,因此是一种偶然性类似。相反用替代矩阵打分系统打分,用重排打分评价是 300 次才 有一次机会得分与替代矩阵打分系统相同的得分。因此用矩阵打分系统得出的蛋白进化关联 性结论更可靠(图 6.12)。 基于序列分析的经验得到了一些简单的评估原则。对于长度超过 100 氨基酸的两个蛋白 质,序列一致性超过 25%就不可能是偶然的,很可能是同源物。相反,序列一致性低于 15% 的两个蛋白质仅仅依靠匹配比较显示的类似性没有显著的统计意义。而序列一致性在 15% ~ 25%之间的蛋白质,需要进一步分析以确定比对的统计意义。必须指出,序列相似性在统计 上不明显的蛋白质也有可能是同源物。从同一祖先进化而来的很多蛋白质差异化程度高至序 列比对无法看出它们的同源性。这类同源蛋白的三维结构相似。 图 6.11 一致性比对和 Blosum 62 矩阵比对。重复重排打分对豆血红蛋白和人肌红蛋白序列 比对进行评估,简单一致性比对(A)和 Blosum 62 矩阵打分比对(B)。红色显示真正序列的比 对,说明 Blosum 矩阵打分的统计意义明显

Myoglobin GDs目GEWOLVLNY网GKVEADI PGHGQEVLIRLFKGHPETLEKFDKFKHLK邓EDpM Leghemoglobin GALTESQAALVKS SWWWFNANI PKHTHRF FILVLE I APAAK---DLESFLKSTSEV KASE-DLKKHGATVLTALGGI---LKKKGH--HEAE IKPLAQSHATKHKI PVKYLE PONNP ELQAHAGKVFKLVYEAA I QLEVTGVVVTDATLKNLGSVHVSKG-VADAHFP FI SECTIQVLOS KHPGDFGADAQGAMNKALELFRKDMASNYK-ELGFOG VVKEAILKT I KEV----VGAKWSEELNSAWT I ATDE LAIVIKKEMDDAA pigure 6-12 图6.12人肌红蛋白和豆血红蛋白进行的序列比对(采用B10sum62替代矩阵打分系统)。保 守替代用黄色涂出,相同氨基酸加框,用桔子颜色涂出。 搜寻数据库、确定同源序列 确定一个蛋白序列后,首先要做的事情就是将这个蛋白质的氨基酸序列与所有已经鉴定 的蛋白序列比较,从而了解这种蛋白质的进化、结构与功能。就是采用前面介绍的比对方案 将这个蛋白质的氨基酸序列与序列已知蛋白数据库的所有成员进行比对。 最常用的同源序列搜寻可以在国立生物技术信息中心网站(ww.ncbi.nih.gov)完成。所用 的方法是BLAST搜寻(基本局部比对搜寻工具,basic local alignment search tool))。氨基酸 序列可以打印或粘贴到网站上,进行搜寻。最常用的是进行所有已知序列非冗余数据库搜寻。 截至2004年,该数据库有300多万序列。BLAST搜寻产生一个序列比对表,每栏数据后标 出了偶然发生这种相似性的几率(图6.13)。 1995年,研究人员报道了第一例自由生活生物(Hamophilus fluenza,.嗜血流感细菌)的 全基因组序列。将这个基因组序列推测出来的编码蛋白序列进行BLAST搜寻,1743种蛋白 质中有1007种蛋白质(58%)与早先鉴定的其它生物的功能已知蛋白密切相关。还有347种蛋 白与数据库内其它生物的"假定蛋白"相关。其余389种序列与数据库的任何序列没有关联 性。因此纯粹进行序列比对就能够确定该生物半数以上蛋白质的功能
图 6.12 人肌红蛋白和豆血红蛋白进行的序列比对(采用 Blosum 62 替代矩阵打分系统)。保 守替代用黄色涂出,相同氨基酸加框,用桔子颜色涂出。 搜寻数据库、确定同源序列 确定一个蛋白序列后,首先要做的事情就是将这个蛋白质的氨基酸序列与所有已经鉴定 的蛋白序列比较,从而了解这种蛋白质的进化、结构与功能。就是采用前面介绍的比对方案 将这个蛋白质的氨基酸序列与序列已知蛋白数据库的所有成员进行比对。 最常用的同源序列搜寻可以在国立生物技术信息中心网站(www.ncbi.nih.gov)完成。所用 的方法是 BLAST 搜寻(基本局部比对搜寻工具,basic local alignment search tool)。氨基酸 序列可以打印或粘贴到网站上,进行搜寻。最常用的是进行所有已知序列非冗余数据库搜寻。 截至 2004 年,该数据库有 300 多万序列。BLAST 搜寻产生一个序列比对表,每栏数据后标 出了偶然发生这种相似性的几率(图 6.13)。 1995 年,研究人员报道了第一例自由生活生物(Hamophilus fluenza, 嗜血流感细菌)的 全基因组序列。将这个基因组序列推测出来的编码蛋白序列进行 BLAST 搜寻,1743 种蛋白 质中有 1007 种蛋白质(58%)与早先鉴定的其它生物的功能已知蛋白密切相关。还有 347 种蛋 白与数据库内其它生物的"假定蛋白"相关。其余 389 种序列与数据库的任何序列没有关联 性。因此纯粹进行序列比对就能够确定该生物半数以上蛋白质的功能

BLA8TP2.2,10【0ct-19-2004] Identifier of query Query=gi12517444gbAAG58041.1AE005521 9 ribosephosphate sequence isomerase, constitutive [Escherichia coli 0157:H7] (219 letters) Database:All non-redundant GenBank CDS translations+PDB+SwissProt+PIR+PRF excluding environmental samples 2,205,431 sequences;747,548,157 total letters Distribution of 268 Blast Hits on the Query Sequence Score E ldentifier of Sequences producing significant alignments: (bits)value homologous sequence gi42943 emb|CAA47309.1 unnamed protein product [E.coli] 427 e-118 bond in search gi12517444gbAAG58041.1 ribosephosphate isomerase,const... 927 e-118 Name [species] of homologous gi33126317gbAAK95569.1 ribose 5-phosphate isomerase [H.sapiens]117 2e-25 protein gi29897136gbAAP10413.1 Phosphoglycerate mutase [B.cereus] 370.35 gi|33126317|gb|AAK95569.1| ribose 5-phosphate isomerase [Homo sapiens] Length 237 Amino acid Score 117 bits (294),Expect 2e-25 Sequence 1dent1t1es=82/224(36告),Positives=118/224(52告),Gaps=15/224(6者) being queried Query:4 DELKKAVGWAALQ-YVQPGTIVGVGTGSTAAHPIDALGTMKGQIE---GAVSSSDASTEK 59 Sequence of +E KK G AA+++V+ ++G+G+GST H++ Q ++S++ homologous Sbict:5 EEAKKLAGRAAVENHVRNNOVLGIGSGSTIVHAVORIAERVKOENLNLVCIPTSFQAROL 64 protein from Homo sapiens Query:60 LKSLGIHVFDLNEVDSLGIYVDGADEINGHMQMIKGGGAALTREKIIASVAEKFICIADA 119 G++DL+ ++DGADE++ +IKGGG LT+EKI+AA +FI IAD Plus sign Sbict:65 ILQYGLTLSDLDRHPEIDLAIDGADEVDADLNLIKGGGGCLTQEKIVAGYASRFIVIADF 124 "positive,"a frequent Query:120 SKOVDILG---KFPLPVEVIPMARSAVARQL-VKLGGRPEYROG------VVTDNGNVIL 169 substitution IG +P+EVIPMA V+R+K GG E R VVTDNGN IL Sbjct:125 RKDSKNLGDQWHKGIPIEVIPMAYVPVSRAVSQKFGGVVELRMAVNKAGPVVTDNGNFIL 184 Letter= Query:170 DVHGMEILDPIAMENAINAIPGVVTVGLFANRGADVALIGTPDG 213 identity +AI IPGVV GLE N A+ G DG i.e the two Sbjct:185 DWKFDRVHKWSEVNTAIKMIPGVVDTGLFINM-AERVYFGMODG 227 sequences Figure 6-13 Biochemistry,Sixth Edition 2007 W.H.Freeman and Company 图6.13 BLAST搜寻的结果。用Ecoli核糖5-磷酸异构酶(也称为磷酸戊糖异构酶)的序列 BLAST搜寻非冗余序列(n)数据库的部分结果。其中有268个序列是人类同源蛋白的垂直同 源物。这些序列的比对用黄色标出。E数据(用红色标出)显示偶然出现同一水平类似性的 几率是2x1025。由于这个数据远远低于1,因此这种序列比对具有显著的统计意义。 6.3三维结构比较有助于进化关系研究 蛋白质序列比较是了解蛋白质功能和亲缘关系的有力工具。但是,生物分子的功能更取 决于它的三维结构。只有功能才能反映序列变异的效果,而功能是由分子的空间结构决定的。 因此,为了深入了解蛋白质的进化关系,我们必须考虑蛋白质的三维结构,尤其是与三维结 构相关的氨基酸序列。第3章我们介绍了测定蛋白质结构的方法
图 6.13 BLAST 搜寻的结果。用 Ecoli 核糖 5-磷酸异构酶(也称为磷酸戊糖异构酶)的序列 BLAST 搜寻非冗余序列(nr)数据库的部分结果。其中有 268 个序列是人类同源蛋白的垂直同 源物。这些序列的比对用黄色标出。E 数据(用红色标出)显示偶然出现同一水平类似性的 几率是 2 x 10 -25。由于这个数据远远低于 1,因此这种序列比对具有显著的统计意义。 6.3 三维结构比较有助于进化关系研究 蛋白质序列比较是了解蛋白质功能和亲缘关系的有力工具。但是,生物分子的功能更取 决于它的三维结构。只有功能才能反映序列变异的效果,而功能是由分子的空间结构决定的。 因此,为了深入了解蛋白质的进化关系,我们必须考虑蛋白质的三维结构,尤其是与三维结 构相关的氨基酸序列。第 3 章我们介绍了测定蛋白质结构的方法