
第2章延伸阅读 延伸阅读2-1基因概念的发展 基因(遗传因子)是产生一条多肽链或功能RNA所需的全部核苷酸序列。1909年丹麦遗传学家约翰逊(W, Johansen1859~1927)在《精密遗传学原理》一书中正式提出基因°概念,用以替代孟德尔(G.J.Mendel)早年所提出 的遗传因子(genetic factor)一词。经过l00多年的发展,基因的概念也在不断更新,经历了经典基因概念到现代新的 基因概念的形成过程。早期的基因概念简单,就是能够编码蛋白质的DNA片段,现在己很难用几句话把基因概念表 述清楚。下面是目前常见的几类基因及其概念。 跳跃基因(leap gene),是指能够改变自身位置的一段DNA序列,也叫“转座元”,可在染色体内或不同染色体 间移动,其本身可编码转座酶(transposase),也含有非蛋白编码序列。1956年,麦克林托克(B.McClintock)在研 究玉米籽粒的色素斑点时,首先提出了一个可在染色体上移动的“控制因子”,一个控制因子整合到一个基因位点上, 可产生一种新的突变型。这种可从染色体的一个位置转移到另一个位置,或从一条染色体转移到另一条染色体的 DNA片段叫做跳跃基因。跳跃基因的发现使人们进一步认识到基因不是稳定、静止不动的实体,它在结构上有明确 的界限,在功能上是一个单独的遗传单位,它可以通过自身的运动调节基因的活性。 重复基因(repeat gene),指在真核基因组中具有一份以上拷贝的基因,这些拷贝或在一条染色体上串联排列, 或分散到多条染色体上。I968年,布里顿(R.J.Britten)和科恩(S.S.Kohne)通过DNA分子复性动力学研究发现, 在真核细胞DNA中,存在着高度重复序列(重复频率大于105)、中度重复序列(重复频率在102~10之间)和非重 复序列(单一序列)。重复基因的发现,使人们从“量”上对基因的概念又有了新的认识。 TTAGCCGTAACTCCAATTCCGTCATAATGCCTCTATTGGC 3 图S2-1重复基因 重叠基因(overlapping gene),指公用一段DNA序列的两个或两个以上结构基因的互称,这与“互不沾染、单个 分离”的传统基因概念相悖。该基因1977年由桑格(F.Sanger)在研究DX174噬菌体DNA的核苷酸序列时发现的,它 的同一部分DNA序列能够编码两种不同的蛋白质,即不同基因的核苷酸序列有时是可以共同的,它们的核苷酸序列 彼此重叠,这样的基因称为重叠基因。这在低等生物尤其在病毒中重叠基因是普遍存在的。因为病毒基因组都很小, 为了利用有限的DNA序列编码更多的蛋白质,不得不使几个基因的编码序列重叠在一起,这是病毒等增加自身基因 遗传信息量的一种进化策略
1 第2章 延伸阅读 延伸阅读 2-1 基因概念的发展 基因(遗传因子)是产生一条多肽链或功能RNA所需的全部核苷酸序列。1909年丹麦遗传学家约翰逊(W. Johansen 1859~1927)在《精密遗传学原理》一书中正式提出“基因”概念,用以替代孟德尔(G.J. Mendel)早年所提出 的遗传因子(genetic factor)一词。经过100多年的发展,基因的概念也在不断更新,经历了经典基因概念到现代新的 基因概念的形成过程。早期的基因概念简单,就是能够编码蛋白质的DNA片段,现在已很难用几句话把基因概念表 述清楚。下面是目前常见的几类基因及其概念。 跳跃基因(leap gene),是指能够改变自身位置的一段DNA序列,也叫“转座元”,可在染色体内或不同染色体 间移动,其本身可编码转座酶(transposase),也含有非蛋白编码序列。1956年,麦克林托克(B. McClintock)在研 究玉米籽粒的色素斑点时,首先提出了一个可在染色体上移动的“控制因子”,一个控制因子整合到一个基因位点上, 可产生一种新的突变型。这种可从染色体的一个位置转移到另一个位置,或从一条染色体转移到另一条染色体的 DNA片段叫做跳跃基因。跳跃基因的发现使人们进一步认识到基因不是稳定、静止不动的实体,它在结构上有明确 的界限,在功能上是一个单独的遗传单位,它可以通过自身的运动调节基因的活性。 重复基因(repeat gene),指在真核基因组中具有一份以上拷贝的基因,这些拷贝或在一条染色体上串联排列, 或分散到多条染色体上。1968年,布里顿(R.J. Britten)和科恩(S.S. Kohne)通过DNA分子复性动力学研究发现, 在真核细胞DNA中,存在着高度重复序列(重复频率大于10 5)、中度重复序列(重复频率在10 2~10 5之间)和非重 复序列(单一序列)。重复基因的发现,使人们从“量”上对基因的概念又有了新的认识。 图S2-1 重复基因 重叠基因(overlapping gene),指公用一段DNA序列的两个或两个以上结构基因的互称,这与“互不沾染、单个 分离”的传统基因概念相悖。该基因1977年由桑格(F. Sanger)在研究ΦX174噬菌体DNA的核苷酸序列时发现的,它 的同一部分DNA序列能够编码两种不同的蛋白质,即不同基因的核苷酸序列有时是可以共同的,它们的核苷酸序列 彼此重叠,这样的基因称为重叠基因。这在低等生物尤其在病毒中重叠基因是普遍存在的。因为病毒基因组都很小, 为了利用有限的DNA序列编码更多的蛋白质,不得不使几个基因的编码序列重叠在一起,这是病毒等增加自身基因 遗传信息量的一种进化策略
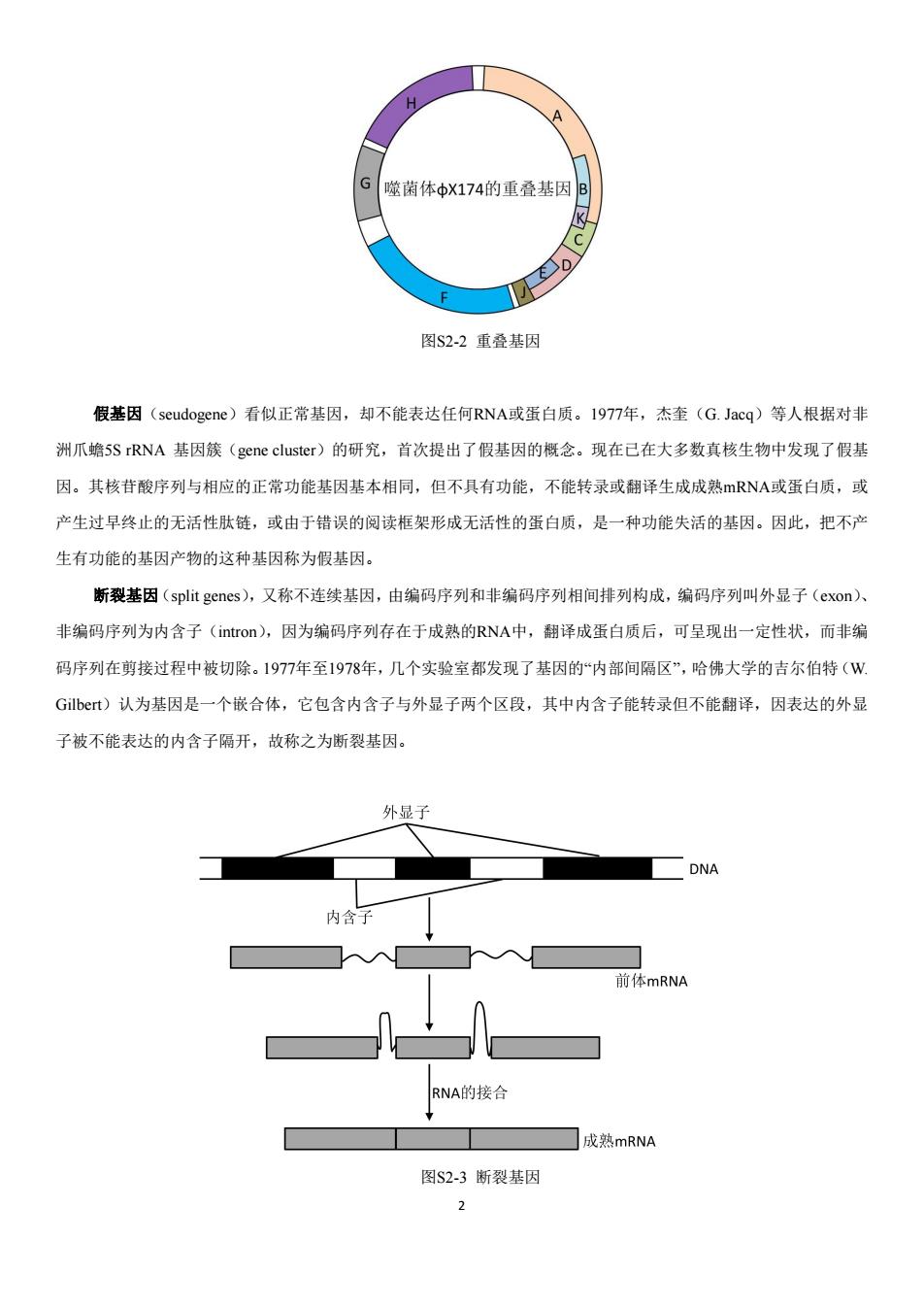
G 噬菌体中X174的重叠基因B 图S2-2重叠基因 假基因(seudogene)看似正常基因,却不能表达任何RNA或蛋白质。1977年,杰奎(G.Jacq)等人根据对非 洲爪蟾5 S rRNA基因簇(gene cluster)的研究,首次提出了假基因的概念。现在已在大多数真核生物中发现了假基 因。其核苷酸序列与相应的正常功能基因基本相同,但不具有功能,不能转录或翻译生成成熟mRNA或蛋白质,或 产生过早终止的无活性肽链,或由于错误的阅读框架形成无活性的蛋白质,是一种功能失活的基因。因此,把不产 生有功能的基因产物的这种基因称为假基因。 断裂基因(split genes),又称不连续基因,由编码序列和非编码序列相间排列构成,编码序列叫外显子(cxon)、 非编码序列为内含子(intron),因为编码序列存在于成熟的RNA中,翻译成蛋白质后,可呈现出一定性状,而非编 码序列在剪接过程中被切除。1977年至1978年,几个实验室都发现了基因的“内部间隔区”,哈佛大学的吉尔伯特(W Gilbert)认为基因是一个嵌合体,它包含内含子与外显子两个区段,其中内含子能转录但不能翻译,因表达的外显 子被不能表达的内含子隔开,故称之为断裂基因。 外显子 DNA 内含子 前体mRNA RNA的接合 成熟mRNA 图S2-3断裂基因
2 图S2-2 重叠基因 假基因(seudogene)看似正常基因,却不能表达任何RNA或蛋白质。1977年,杰奎(G. Jacq)等人根据对非 洲爪蟾5S rRNA 基因簇(gene cluster)的研究,首次提出了假基因的概念。现在已在大多数真核生物中发现了假基 因。其核苷酸序列与相应的正常功能基因基本相同,但不具有功能,不能转录或翻译生成成熟mRNA或蛋白质,或 产生过早终止的无活性肽链,或由于错误的阅读框架形成无活性的蛋白质,是一种功能失活的基因。因此,把不产 生有功能的基因产物的这种基因称为假基因。 断裂基因(split genes),又称不连续基因,由编码序列和非编码序列相间排列构成,编码序列叫外显子(exon)、 非编码序列为内含子(intron),因为编码序列存在于成熟的RNA中,翻译成蛋白质后,可呈现出一定性状,而非编 码序列在剪接过程中被切除。1977年至1978年,几个实验室都发现了基因的“内部间隔区”,哈佛大学的吉尔伯特(W. Gilbert)认为基因是一个嵌合体,它包含内含子与外显子两个区段,其中内含子能转录但不能翻译,因表达的外显 子被不能表达的内含子隔开,故称之为断裂基因。 图S2-3 断裂基因

隐蔽基因(cryptogene)的编码序列与mRNA的相应序列有差异,转录产物上需插入、删除或取代一些核苷酸 才能生成有翻译功能的mRNA分子。一般来说,用做翻译模板的mRNA分子应该与其编码基因有对应关系,也就是 说它的核苷酸碱基与基因的核苷酸碱基互补,而且数量相等,对真核基因来说应与外显子序列相对应。但是自1985 年以来,在某些病毒、植物和动物中发现mRNA前体在成熟过程中发生了碱基的增加、缺失或替换,mRNA与基因 之间失去了一一对应关系。这一现象首先在原生动物中发现,并称之为NA编辑。这种需要编辑才能正常表达的基 因称为隐蔽基因。 反转录基因(retron)是一群古老的以反转录作用合成RNA/mDNA复合msDNA分子的遗传单元。retron是1987 年由病毒学家塔戴尔(A.Dhundale)等在一种古老的细菌Myxococcus xanthus中通过一种叫msDNA(multicopy single-stranded DNA)的新型卫星DNA而发现的。以后的研究证明msDNA广泛存在于细菌类如Myxobacter的许 多属和种中,它由细菌基因组上一段叫retron的DNA序列经转录和反转录产生,称之为反转录基因
3 隐蔽基因(cryptogene)的编码序列与mRNA的相应序列有差异,转录产物上需插入、删除或取代一些核苷酸 才能生成有翻译功能的mRNA分子。一般来说,用做翻译模板的mRNA分子应该与其编码基因有对应关系,也就是 说它的核苷酸碱基与基因的核苷酸碱基互补,而且数量相等,对真核基因来说应与外显子序列相对应。但是自1985 年以来,在某些病毒、植物和动物中发现mRNA前体在成熟过程中发生了碱基的增加、缺失或替换,mRNA与基因 之间失去了一一对应关系。这一现象首先在原生动物中发现,并称之为RNA编辑。这种需要编辑才能正常表达的基 因称为隐蔽基因。 反转录基因(retron)是一群古老的以反转录作用合成 RNA/mDNA 复合 msDNA 分子的遗传单元。retron 是 1987 年由病毒学家塔戴尔(A. Dhundale)等在一种古老的细菌 Myxococcus xanthus 中通过一种叫 msDNA(multicopy single-stranded DNA)的新型卫星 DNA 而发现的。以后的研究证明 msDNA 广泛存在于细菌类如 Myxobacter 的许 多属和种中,它由细菌基因组上一段叫 retron 的 DNA 序列经转录和反转录产生,称之为反转录基因

延伸阅读2-2DNA体外重组技术的建立 DNA体外重组技术是指在体外将两个或多个来源相同或不相同的DNA片段连接成新的重组DNA分子,再转 到特定宿主细胞中进行自主复制并表达。这项技术大约建立于20世纪70年代初期。 l972年,美国斯坦福大学的伯格(P.Brg)等人将一种猿猴病毒的DNA与噬菌体DNA用同一种限制性内切 酶切割后,再用DNA连接酶把这两种DNA分子连接起来,构建了世界上第一个重组DNA分子,并因此获得了1980 年度诺贝尔化学奖。 1973年,美国斯坦福大学的科恩(S.Cohen)等人首次通过将大肠杆菌的抗四环素(Tetr)质粒PSCI01和抗新 霉素(N)及抗磺胺(S)的质粒R6-3,在体外用限制性内切酶EcoR I切割,用连接酶连接成新的重组质粒,然 后转化到大肠杆菌中,在含四环素和新霉素的平板上选出了抗四环素和抗新霉素的重组菌落,完成了体外DNA重 组和扩增的全过程。该实验的成功是基因工程发展史上的一个里程碑,它不仅说明质粒分子可以作为基因克隆的载 体,将外源DNA导入寄主细胞,还说明真核生物的基因可以转移到原核细胞中,并实现其功能表达,同时还建立 了质粒一大肠杆菌这样一个基因克隆模式,使人们对基因工程前景充满了希望。因此,1973年定为基因工程元年
4 延伸阅读 2-2 DNA 体外重组技术的建立 DNA 体外重组技术是指在体外将两个或多个来源相同或不相同的 DNA 片段连接成新的重组 DNA 分子,再转 到特定宿主细胞中进行自主复制并表达。这项技术大约建立于 20 世纪 70 年代初期。 1972 年,美国斯坦福大学的伯格(P. Berg)等人将一种猿猴病毒的 DNA 与噬菌体 DNA 用同一种限制性内切 酶切割后,再用 DNA 连接酶把这两种 DNA 分子连接起来,构建了世界上第一个重组 DNA 分子,并因此获得了 1980 年度诺贝尔化学奖。 1973 年,美国斯坦福大学的科恩(S. Cohen)等人首次通过将大肠杆菌的抗四环素(Tet r)质粒 PSCI01 和抗新 霉素(Ne r)及抗磺胺(S r)的质粒 R6-3,在体外用限制性内切酶 EcoR I 切割,用连接酶连接成新的重组质粒,然 后转化到大肠杆菌中,在含四环素和新霉素的平板上选出了抗四环素和抗新霉素的重组菌落,完成了体外 DNA 重 组和扩增的全过程。该实验的成功是基因工程发展史上的一个里程碑,它不仅说明质粒分子可以作为基因克隆的载 体,将外源 DNA 导入寄主细胞,还说明真核生物的基因可以转移到原核细胞中,并实现其功能表达,同时还建立 了质粒—大肠杆菌这样一个基因克隆模式,使人们对基因工程前景充满了希望。因此,1973 年定为基因工程元年

延伸阅读2-3人类基因组计划 人类基因组计划(human genome project,HGP)是由美国科学家于1985年率先提出,于1990年正式启动,由 美国、英国、法国、德国、日本和我国科学家共同参与测定组成人类染色体(指单倍体中所包含的30亿个碱基对 组成的核苷酸序列,绘制人类基因组图谱的一项科学探索工程。其目的是希望能辨识人类染色体上载有的基因及其 序列,并破译其中的遗传信息。这是人类为了探索自身的奥秘所迈出的重要一步,是人类科学史上的一个伟大工程。 ①主要内容。人类基因组计划的研究内容主要表现在四张图上:遗传图、物理图、序列图和转录图,主要是绘 制人类基因组序列框架图。I993年马里兰州Hunt,Valley会议上,经美国国立人类基因组研究中心(National Human Genome Research Institute,.NHGRI)修订后的HGP内容包括:人类基因组作图及序列分析:基因的鉴定:基因组研 究技术的建立、创新与改进:模式生物(主要包括大肠杆菌、酵母、果蝇、线虫、小鼠、水稻、拟南芥等)基因组 的作图及测序:信息系统的建立,信息的储存、处理及相应的软件开发:与人类基因组相关的伦理学、法学和社会 影响与结果的研究:研究人员的培训:技术转让及产业开发:研究计划的外延等几方面,这些内容构成了20世纪 到21世纪最大的系统工程。 ②基本策略。测定整个基因组的经典方法是先作图再测序。研究的基本策略包括1)cDNA策略:人类基因组 中发生转录表达的序列(即基因)仅占总序列的约5%,对这一部分序列进行测定将直接导致基因的发现。其方法 是从cDNA文库中选取基因并克隆,进行序列测定,然后将结果通过计算机与己知的数据库进行比较,最终在染色 体上进行定位。2)基因鉴定的策略:通常采用定位克隆或候选定位克隆的策略,即利用与疾病连锁的标记在基因 组文库中找到表达序列,再利用这一表达序列去识别全部cDNA,找到该基因。 ③科学意义。HGP研究已经最终破译了人类DNA分子的全部核苷酸序列,建立了人类遗传物质的一整套信息 数据库;可以在DNA全序列阐明的基础上建立染色体的三维结构,进一步来解释扩增、插入、缺失、易位、重组 等过程的分子机制:可以使医学上的5千多种遗传疾病以及恶性肿瘤、心血管疾病等由此得到预测、预防、早期诊 断和治疗:HGP作为整个生命科学发展的“突破口”,可以带动生命科学其他领域及应用生物技术的发展,并对所涉 及的伦理、法律等社会科学领域也将产生巨大的影响。因此,HGP的研究不仅具有深刻的科学意义,也具有深远的 社会意义。HG对生命科学研究与生物产业发展的导向性意义,可以用规模化、序列化、信息化、产业化、医学化、 人文化来归纳
5 延伸阅读 2-3 人类基因组计划 人类基因组计划(human genome project, HGP)是由美国科学家于 1985 年率先提出,于 1990 年正式启动,由 美国、英国、法国、德国、日本和我国科学家共同参与测定组成人类染色体(指单倍体中所包含的 30 亿个碱基对 组成的核苷酸序列,绘制人类基因组图谱的一项科学探索工程。其目的是希望能辨识人类染色体上载有的基因及其 序列,并破译其中的遗传信息。这是人类为了探索自身的奥秘所迈出的重要一步,是人类科学史上的一个伟大工程。 ①主要内容。人类基因组计划的研究内容主要表现在四张图上:遗传图、物理图、序列图和转录图,主要是绘 制人类基因组序列框架图。1993 年马里兰州 Hunt, Valley 会议上,经美国国立人类基因组研究中心(National Human Genome Research Institute, NHGRI)修订后的 HGP 内容包括:人类基因组作图及序列分析;基因的鉴定;基因组研 究技术的建立、创新与改进;模式生物(主要包括大肠杆菌、酵母、果蝇、线虫、小鼠、水稻、拟南芥等)基因组 的作图及测序;信息系统的建立,信息的储存、处理及相应的软件开发;与人类基因组相关的伦理学、法学和社会 影响与结果的研究;研究人员的培训;技术转让及产业开发;研究计划的外延等几方面,这些内容构成了 20 世纪 到 21 世纪最大的系统工程。 ②基本策略。测定整个基因组的经典方法是先作图再测序。研究的基本策略包括 1)cDNA 策略:人类基因组 中发生转录表达的序列(即基因)仅占总序列的约 5%,对这一部分序列进行测定将直接导致基因的发现。其方法 是从 cDNA 文库中选取基因并克隆,进行序列测定,然后将结果通过计算机与已知的数据库进行比较,最终在染色 体上进行定位。2)基因鉴定的策略:通常采用定位克隆或候选定位克隆的策略,即利用与疾病连锁的标记在基因 组文库中找到表达序列,再利用这一表达序列去识别全部 cDNA,找到该基因。 ③科学意义。HGP 研究已经最终破译了人类 DNA 分子的全部核苷酸序列,建立了人类遗传物质的一整套信息 数据库;可以在 DNA 全序列阐明的基础上建立染色体的三维结构,进一步来解释扩增、插入、缺失、易位、重组 等过程的分子机制;可以使医学上的 5 千多种遗传疾病以及恶性肿瘤、心血管疾病等由此得到预测、预防、早期诊 断和治疗;HGP 作为整个生命科学发展的“突破口”,可以带动生命科学其他领域及应用生物技术的发展,并对所涉 及的伦理、法律等社会科学领域也将产生巨大的影响。因此,HGP 的研究不仅具有深刻的科学意义,也具有深远的 社会意义。HGP 对生命科学研究与生物产业发展的导向性意义,可以用规模化、序列化、信息化、产业化、医学化、 人文化来归纳

延伸阅读2-4柯斯质粒载体的构建 柯斯质粒是一类由人工构建的含有-DNA的cos序列和质粒复制子的特殊类型的载体。是1978年由collins和 hohn发明构建的一种新型大肠杆菌克隆载体,用正常的质粒与噬菌体的cos位点构成。 柯斯质粒载体pHC79,是由1.8kb的-DNA片段和pBR322质粒DNA片段联合组成。插入柯斯质粒载体的外 源DNA片段的长度范围为31-45kb(图S2-4)。 BamH I Pst l Sal I Tc Ap pHC79 (6400bp) 入-DNA片段 ori cos 图S2-4柯斯质粒载体pHC79 6
6 延伸阅读 2-4 柯斯质粒载体的构建 柯斯质粒是一类由人工构建的含有λ-DNA 的 cos 序列和质粒复制子的特殊类型的载体。是 1978 年由 collins 和 hohn 发明构建的一种新型大肠杆菌克隆载体,用正常的质粒与噬菌体λ的 cos 位点构成。 柯斯质粒载体 pHC79,是由 1.8 kb 的λ-DNA 片段和 pBR322 质粒 DNA 片段联合组成。插入柯斯质粒载体的外 源 DNA 片段的长度范围为 31-45 kb(图 S2-4)。 图 S2-4 柯斯质粒载体 pHC79

延伸阅读2-5PCR反应合成目的DNA示例 PCR扩增技术也称为聚合酶链式反应(polymerase chain reaction),是以DNA变性、复制的某些特性为原理设 计的,可从痕量的DNA样品中特异快速扩增某一区域的DNA片段。通过PCR技术获取所需要的特异DNA片段 在实际应用中用得非常多,但是前提条件是必须对目的基因有一定的了解,通过目的基因序列设计相应的引物序列。 在微量离心管中,加入待扩增的DNA片段两端互补的两个引物、适量的缓冲液、微量的DNA模板、4种dNTP为 底物,耐热Tag DNA聚合酶等,在高温下使DNA双链解旋变性,然后降低温度,以单链DNA为模板,引物与模 板结合,形成部分双链:再升温至合适温度,以dNTP为原料,在Tag DNA聚合酶的催化下,引物沿5'→3方向延 伸,形成新的DNA片段,该片段又可作为下一轮反应的模板,如此循环往复,使目的DNA得以迅速扩增。例如: 【实验目的】体外获得人α-乳白蛋白质基因片段 【实验原理】通过PCR方法从人类基因组DNA中扩增人a-乳白蛋白质基因。 【实验试剂与器材】①器材:PCR反应管,微量移液器,PCR仪等:②人基因组DNA,PCR反应buffer,dNTP (四种脱氧核糖核酸混合液),Taq聚合酶,上游引物:5'-ATGCTTCCATTTCAGGTTCT-3':下游引物: 5'-CAGTGTCCACCTACTCATCG-3',双蒸水. 【实验方法与步骤】①根据GeneBank上人a-乳白蛋白质DNA序列(GeneBank Acession No.AC000055),利用 引物设计软件设计一对特异性引物,预期扩增片段长度为2446bp。②在PCR反应管中依次加入4l5×Primer反应 buffer,1.6 ul dNTP Mixture(2.5mmol/L),1l(约100ng)人基因组DNA,上下游引物各0.6l,0.2l的Prime HS Taq, 加入双蒸水至20l。反应程序:98℃变性10s,56℃退火10s,72℃延伸2min30s,进行35个循环。③由此扩 增获得的DNA片段即为人a-乳白蛋白质基因。 利用PCR技术可以大量扩增包括目的基因在内的DNA特定靶序列,但在某些情况下,PCR扩增产物仍需要克 隆在受体细胞中,如目的基因的高效表达以及永久保存等。当目的基因或目的基因组过长,用Taq DNA聚合酶难 以一次扩增时,通常采用分段扩增的方法,以I~2kb为一个扩增单位,然后将多个扩增DNA片段拼接成全长基 因。PCR技术不仅能扩增两段已知序列之间的DNA区域,而且还可克隆一段已知序列两侧的DNA片段,所用方 法称为染色体步移(genome walking)和LA PCR(linker-adaptor PCR)
7 延伸阅读 2-5 PCR 反应合成目的 DNA 示例 PCR 扩增技术也称为聚合酶链式反应(polymerase chain reaction),是以 DNA 变性、复制的某些特性为原理设 计的,可从痕量的 DNA 样品中特异快速扩增某一区域的 DNA 片段。通过 PCR 技术获取所需要的特异 DNA 片段 在实际应用中用得非常多,但是前提条件是必须对目的基因有一定的了解,通过目的基因序列设计相应的引物序列。 在微量离心管中,加入待扩增的 DNA 片段两端互补的两个引物、适量的缓冲液、微量的 DNA 模板、4 种 dNTP 为 底物,耐热 Taq DNA 聚合酶等,在高温下使 DNA 双链解旋变性,然后降低温度,以单链 DNA 为模板,引物与模 板结合,形成部分双链;再升温至合适温度,以 dNTP 为原料,在 Taq DNA 聚合酶的催化下,引物沿 5’→3’方向延 伸,形成新的 DNA 片段,该片段又可作为下一轮反应的模板,如此循环往复,使目的 DNA 得以迅速扩增。 例如: 【实验目的】体外获得人ɑ-乳白蛋白质基因片段 【实验原理】通过 PCR 方法从人类基因组 DNA 中扩增人ɑ-乳白蛋白质基因。 【实验试剂与器材】① 器材:PCR 反应管,微量移液器,PCR 仪等;② 人基因组 DNA,PCR 反应 buffer,dNTP ( 四 种 脱 氧 核 糖 核 酸 混 合 液 ), Taq 聚 合 酶 , 上 游 引 物 : 5’-ATGCTTCCATTTCAGGTTCT-3’ ; 下 游 引 物 : 5’-CAGTGTCCACCTACTCATCG-3’,双蒸水。 【实验方法与步骤】① 根据 GeneBank 上人ɑ-乳白蛋白质 DNA 序列(GeneBank Acession No. AC000055),利用 引物设计软件设计一对特异性引物,预期扩增片段长度为 2 446 bp。② 在 PCR 反应管中依次加入 4 μl 5×Primer 反应 buffer,1.6 μl dNTP Mixture (2.5 mmol/L),1 μl(约 100 ng)人基因组 DNA,上下游引物各 0.6 μl,0.2 μl 的 Prime HS Taq, 加入双蒸水至 20 μl。反应程序:98 OC 变性 10 s,56 OC 退火 10 s,72 OC 延伸 2 min 30 s,进行 35 个循环。③ 由此扩 增获得的 DNA 片段即为人ɑ-乳白蛋白质基因。 利用 PCR 技术可以大量扩增包括目的基因在内的 DNA 特定靶序列,但在某些情况下,PCR 扩增产物仍需要克 隆在受体细胞中,如目的基因的高效表达以及永久保存等。当目的基因或目的基因组过长,用 Taq DNA 聚合酶难 以一次扩增时,通常采用分段扩增的方法,以 1~2 kb 为一个扩增单位,然后将多个扩增 DNA 片段拼接成全长基 因。PCR 技术不仅能扩增两段已知序列之间的 DNA 区域,而且还可克隆一段已知序列两侧的 DNA 片段,所用方 法称为染色体步移(genome walking)和 LA PCR(linker-adaptor PCR)
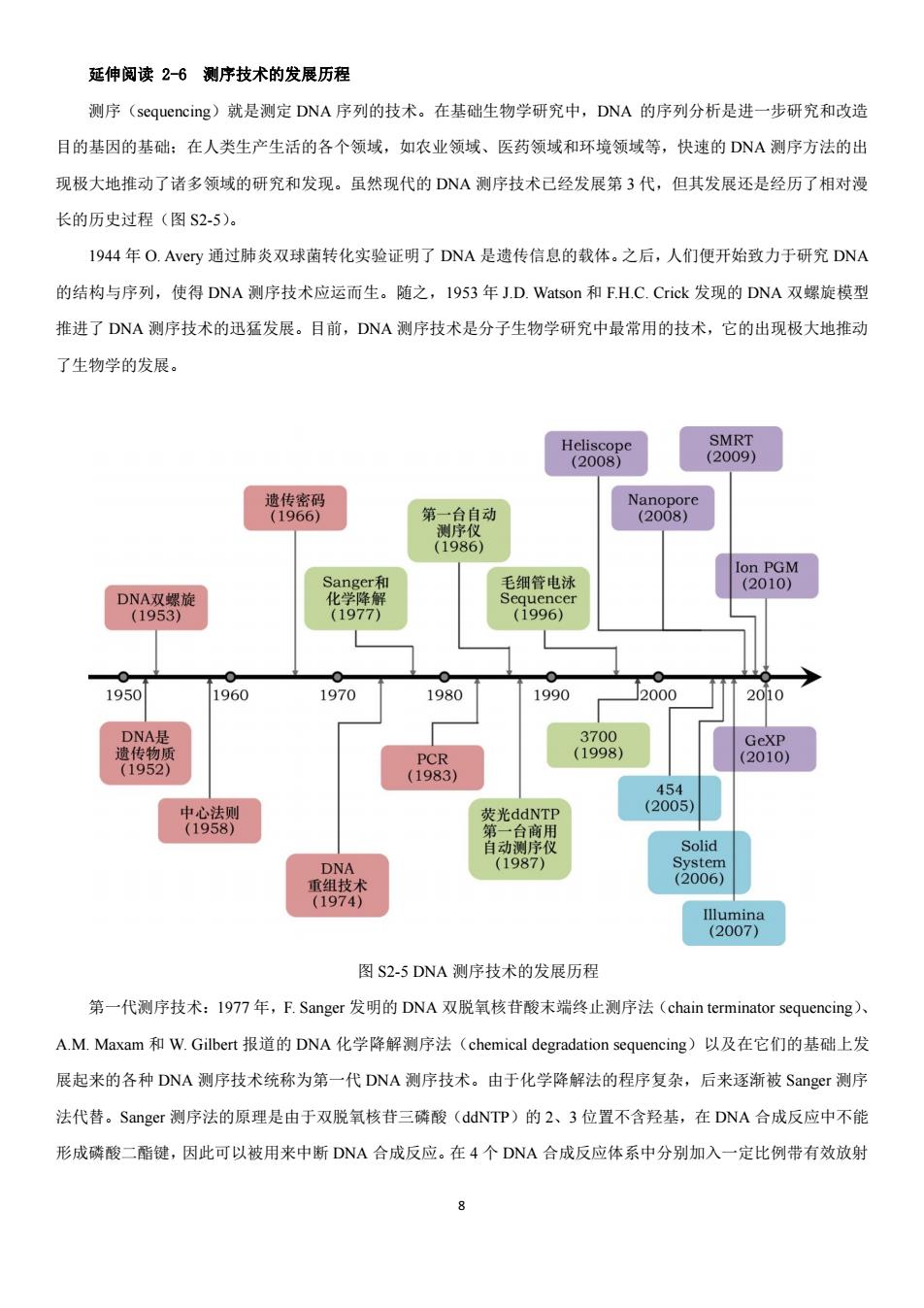
延伸阅读26测序技术的发展历程 测序(sequencing)就是测定DNA序列的技术。在基础生物学研究中,DNA的序列分析是进一步研究和改造 目的基因的基础:在人类生产生活的各个领域,如农业领域、医药领域和环境领域等,快速的DNA测序方法的出 现极大地推动了诸多领域的研究和发现。虽然现代的DNA测序技术已经发展第3代,但其发展还是经历了相对漫 长的历史过程(图S2-5)。 1944年O.Avery通过肺炎双球菌转化实验证明了DNA是遗传信息的载体。之后,人们便开始致力于研究DNA 的结构与序列,使得DNA测序技术应运而生。随之,1953年J.D.Watson和FH.C.Crick发现的DNA双螺旋模型 推进了DNA测序技术的迅猛发展。目前,DNA测序技术是分子生物学研究中最常用的技术,它的出现极大地推动 了生物学的发展。 Heliscope SMRT (2008) (2009) 遗传密码 Nanopore (1966) 第一台自动 (2008) 测序仪 (1986) Ion PGM Sanger和 毛细管电泳 (2010) DNA双螺旋 化学降解 Sequencer (1953) (1977) (1996) 0 0 ● 1950 1960 1970 1980 1990 2000 2010 DNA是 3700 GeXP 遗传物质 PCR (1998) (2010) (1952) (1983) 454 中心法则 荧光ddNTP (2005) (1958) 第一台商用 自动测序仪 Solid DNA (1987) System 重组技术 (2006) (1974) Illumina (2007) 图S2-5DNA测序技术的发展历程 第一代测序技术:1977年,F.Sanger发明的DNA双脱氧核苷酸末端终止测序法(chain terminator sequencing)、 A.M.Maxam和W.Gilbert报道的DNA化学降解测序法(chemical degradation sequencing)以及在它们的基础上发 展起来的各种DNA测序技术统称为第一代DNA测序技术。由于化学降解法的程序复杂,后来逐渐被Sanger测序 法代替。Sanger测序法的原理是由于双脱氧核苷三磷酸(ddNTP)的2、3位置不含羟基,在DNA合成反应中不能 形成磷酸二酯键,因此可以被用来中断DNA合成反应。在4个DNA合成反应体系中分别加入一定比例带有效放射
8 延伸阅读 2-6 测序技术的发展历程 测序(sequencing)就是测定 DNA 序列的技术。在基础生物学研究中,DNA 的序列分析是进一步研究和改造 目的基因的基础;在人类生产生活的各个领域,如农业领域、医药领域和环境领域等,快速的 DNA 测序方法的出 现极大地推动了诸多领域的研究和发现。虽然现代的 DNA 测序技术已经发展第 3 代,但其发展还是经历了相对漫 长的历史过程(图 S2-5)。 1944 年 O. Avery 通过肺炎双球菌转化实验证明了 DNA 是遗传信息的载体。之后,人们便开始致力于研究 DNA 的结构与序列,使得 DNA 测序技术应运而生。随之,1953 年 J.D. Watson 和 F.H.C. Crick 发现的 DNA 双螺旋模型 推进了 DNA 测序技术的迅猛发展。目前,DNA 测序技术是分子生物学研究中最常用的技术,它的出现极大地推动 了生物学的发展。 图 S2-5 DNA 测序技术的发展历程 第一代测序技术:1977 年,F. Sanger 发明的 DNA 双脱氧核苷酸末端终止测序法(chain terminator sequencing)、 A.M. Maxam 和 W. Gilbert 报道的 DNA 化学降解测序法(chemical degradation sequencing)以及在它们的基础上发 展起来的各种 DNA 测序技术统称为第一代 DNA 测序技术。由于化学降解法的程序复杂,后来逐渐被 Sanger 测序 法代替。Sanger 测序法的原理是由于双脱氧核苷三磷酸(ddNTP)的 2、3 位置不含羟基,在 DNA 合成反应中不能 形成磷酸二酯键,因此可以被用来中断 DNA 合成反应。在 4 个 DNA 合成反应体系中分别加入一定比例带有效放射

性同位素标记的某种ddNTP,通过凝胶电泳和放射自显影后,可以根据电泳带的位置确定待测分子的DNA序列。 Sanger测序技术由于操作快速、简单,准确率高和有较长读长的优势而被广泛应用,人类基因组测序正是基于该技 术而完成的。但这一技术容易受到成本低、速度慢和通量低等因素的限制,无法满足大型基因组组装方面的需要。 3'-.CAGTGAATTCGAGCTCGC.-5DNA模板 5'-..GTCAC 引物 ...GTCACT* ..GTCACTT* .GTCACTTA* ...GTCACTTAA* ...GTCACTTAAG* ...GTCACTTAAGC ...GTCACTTAAGCT* ...GTCACTTAAGCTC* 凝胶电泳和放射自显影 图S2-6 Sanger测序法原理 第二代测序技术:随着人类基因组计划的完成,人们进入了后基因组时代,即功能基因组时代,传统的测序方 法已经不能满足深度测序和重复测序等大规模基因组测序的需求,这促使了新一代DNA测序技术的诞生。新一代 测序技术也称为第二代测序技术,主要包括罗氏454公司的GS FLX测序平台、Illumina公司的Solexa Genome Analyzer测序平台和ABI公司的SOLiD测序平台。454生命科学公司测序技术采用的是焦磷酸测序原理,主要步骤 包括:①文库准备,将基因组DNA打碎成300~800bp长的片段,在单链DNA的3'-端和5'-端分别连上不同的接 头。②连接,带有接头的单链DNA被固定在DNA捕获磁珠上,每一个磁珠携带一个单链DNA片段,扩增试剂将 磁珠乳化,形成油包水的混合物,这样就形成了许多个包含一个磁珠和一个独特片段的微反应器。③扩增,每个独 特的片段在自己的微反应器里进行独立的扩增(乳液PCR,emulsion PCR),从而排除了其它序列的竞争。整个DNA 片段文库的扩增平行进行。对于每一个片段而言,扩增产生几百万个相同拷贝。乳液PC终止后,扩增的片段仍 然结合在磁珠上。④测序,携带DNA的捕获磁珠被放入PTP板中进行测序。PTP孔的直径(29)只能容纳一 个磁珠(20um)。放置在4个单独的试剂瓶里的4种碱基,依照T、A、C、G的顺序依次循环进入PTP板,每次 只进入一个碱基。如果发生碱基配对,就会释放一个焦磷酸。这个焦磷酸在ATP硫酸化酶和荧光素酶的作用下,释 放出光信号,并实时地被仪器配置的高灵敏度CCD捕获到。有一个碱基和测序模板进行配对,就会捕获到一分子 的光信号:由此一一对应,就可以准确、快速地确定待测模板的碱基序列。第二代测序技术最显著的特征是高通量, 一次能对几十万到几百万条DNA分子进行序列测序,但是其较短读长不利于生物信息学分析,而且,扩增PCR前 9
9 性同位素标记的某种 ddNTP,通过凝胶电泳和放射自显影后,可以根据电泳带的位置确定待测分子的 DNA 序列。 Sanger 测序技术由于操作快速、简单,准确率高和有较长读长的优势而被广泛应用,人类基因组测序正是基于该技 术而完成的。但这一技术容易受到成本低、速度慢和通量低等因素的限制,无法满足大型基因组组装方面的需要。 图 S2-6 Sanger 测序法原理 第二代测序技术:随着人类基因组计划的完成,人们进入了后基因组时代,即功能基因组时代,传统的测序方 法已经不能满足深度测序和重复测序等大规模基因组测序的需求,这促使了新一代 DNA 测序技术的诞生。新一代 测序技术也称为第二代测序技术,主要包括罗氏 454 公司的 GS FLX 测序平台、Illumina 公司的 Solexa Genome Analyzer 测序平台和 ABI 公司的 SOLiD 测序平台。454 生命科学公司测序技术采用的是焦磷酸测序原理,主要步骤 包括:①文库准备,将基因组 DNA 打碎成 300~800 bp 长的片段,在单链 DNA 的 3’-端和 5’-端分别连上不同的接 头。②连接,带有接头的单链 DNA 被固定在 DNA 捕获磁珠上,每一个磁珠携带一个单链 DNA 片段,扩增试剂将 磁珠乳化,形成油包水的混合物,这样就形成了许多个包含一个磁珠和一个独特片段的微反应器。③扩增,每个独 特的片段在自己的微反应器里进行独立的扩增(乳液 PCR,emulsion PCR),从而排除了其它序列的竞争。整个 DNA 片段文库的扩增平行进行。对于每一个片段而言,扩增产生几百万个相同拷贝。乳液 PCR 终止后,扩增的片段仍 然结合在磁珠上。④测序,携带 DNA 的捕获磁珠被放入 PTP 板中进行测序。PTP 孔的直径(29 μm)只能容纳一 个磁珠(20 μm)。放置在 4 个单独的试剂瓶里的 4 种碱基,依照 T、A、C、G 的顺序依次循环进入 PTP 板,每次 只进入一个碱基。如果发生碱基配对,就会释放一个焦磷酸。这个焦磷酸在 ATP 硫酸化酶和荧光素酶的作用下,释 放出光信号,并实时地被仪器配置的高灵敏度 CCD 捕获到。有一个碱基和测序模板进行配对,就会捕获到一分子 的光信号;由此一一对应,就可以准确、快速地确定待测模板的碱基序列。第二代测序技术最显著的特征是高通量, 一次能对几十万到几百万条 DNA 分子进行序列测序,但是其较短读长不利于生物信息学分析,而且,扩增 PCR 前

后的DNA分子片段数目比例有偏差,这对基因表达,尤其是对大量表达的基因影响会更大。 第三代测序技术:为了补充和进一步完善测序技术,第三代测序技术出现,即单分子测序技术,主要包括Helicos 公司的单分子测序技术(True single-.molecule sequencing,tSMSTM)、Oxford Nanopore公司的单分子纳米孔测序技 术(The single--molecule nanopore DNA sequencing)、Pacific Biosciences(PacBio)公司的单分子实时测序技术 (Single-molecule real--time,SMRT)等。PacBio RS测序技术是第三代测序中应用比较广泛且己商业化的核心技术。 该测序是基于荧光标记的边合成边测序技术,其原理是:DNA模板被聚合酶捕获后,4种不同荧光标记的脱氧核糖 核苷三磷酸(dNTP)通过布朗运动随机进入检测区域与聚合酶结合,与DNA模板匹配的碱基生成化学键的时间比 其他碱基停留的时间长很多,这有利于荧光标记的dNTP被激发而检测到相应的荧光信号,根据发射的不同波长的 荧光基团,可以识别延伸的碱基种类,之后经过信息处理可测定DNA模板序列。SMRT测序具有超长读长、测序 时间短、无需模板扩增和直接检测表观修饰位点等特点,但也存在测序原始数据准确率低,有效反应孔不足,通量 比二代测序低等缺点。 10
10 后的 DNA 分子片段数目比例有偏差,这对基因表达,尤其是对大量表达的基因影响会更大。 第三代测序技术:为了补充和进一步完善测序技术,第三代测序技术出现,即单分子测序技术,主要包括 Helicos 公司的单分子测序技术(True single-molecule sequencing,tSMSTM)、Oxford Nanopore 公司的单分子纳米孔测序技 术(The single-molecule nanopore DNA sequencing)、Pacific Biosciences(PacBio)公司的单分子实时测序技术 (Single-molecule real-time,SMRT)等。PacBio RS 测序技术是第三代测序中应用比较广泛且已商业化的核心技术。 该测序是基于荧光标记的边合成边测序技术,其原理是:DNA 模板被聚合酶捕获后,4 种不同荧光标记的脱氧核糖 核苷三磷酸(dNTP)通过布朗运动随机进入检测区域与聚合酶结合,与 DNA 模板匹配的碱基生成化学键的时间比 其他碱基停留的时间长很多,这有利于荧光标记的 dNTP 被激发而检测到相应的荧光信号,根据发射的不同波长的 荧光基团,可以识别延伸的碱基种类,之后经过信息处理可测定 DNA 模板序列。SMRT 测序具有超长读长、测序 时间短、无需模板扩增和直接检测表观修饰位点等特点,但也存在测序原始数据准确率低,有效反应孔不足,通量 比二代测序低等缺点